Геномные браузеры
В самом начале бросилось в глаза выделение 3 организмов - человека, мыши и зебрафиши. Так же сбоку интересная штучка - "Знаете ли вы"))
В качестве объекта для работы был выбран самый длинный кодирующий участок 1339504..1340259 моего гена FB19 (из первого занятия блока). Его последовательность была помещена в специальное окошко интерфеса
BLAST/BLAT, достаточно удобного и похожего на интерфейс BLAST'а на NCBI. Был осуществлен поиск по кДНК в человеческом геноме.
На странице сервиса BLAST/BLAT помимо различных способов задания искомых последовательностей можно выбрать
вид, по геному которого будет осуществляться поиск, причем их далеко не очень много (около 60ти), а также программы поиска последовательностей (BLASTN, TBLASTX и пр.) и базу данных: нуклеотидную (кДНК, РНК и пр.) или белковую.
Кроме того, можно настроить чувствительность поиска в разделе Search sensitivity.
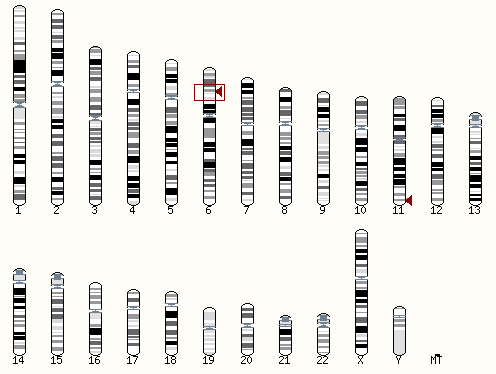
В выдаче наглядно продемонстрирована локализация данного фрагмента в кариотипе(см. ниже).
Помимо этого, так же, как и в BLAST'е можно регулировать количество выданных последовательностей (по умолчанию 100).
Также в выдаче имеется наглядное представление качества находок (линиями разного цвета). Внизу страницы результата представлен
список найденных гомологов, рядом с каждым АС имеется ссылка на выравнивание (см. ниже),а также - более итересно - ссылка
на карту расположения различных генов, их экзонов и интронов в области расположения найденного фрагмента - точнее сказать, расположение контигов (Contig view).
В Contig view наглядно представлено расположение заданного фрагмента в системе контигов, т.е. с какими генами он соседствует, это экзон или интрон.
Можно, кликнув на участок схемы, увеличить её масштаб и тем самым с большей подробностью рассмотреть структуру генома.
Под заголовком Region in detail можно увидеть более подробную структуру хромосомы (а именно, какие
контиги известны), сама хромосома обозначена лентой с перемеживающимися синими и голубыми участками - разными контигами.
Под этой лентой-хромосомой размещены ID генов из Ensembl/Havana в точности там, где и лежат сами гены.
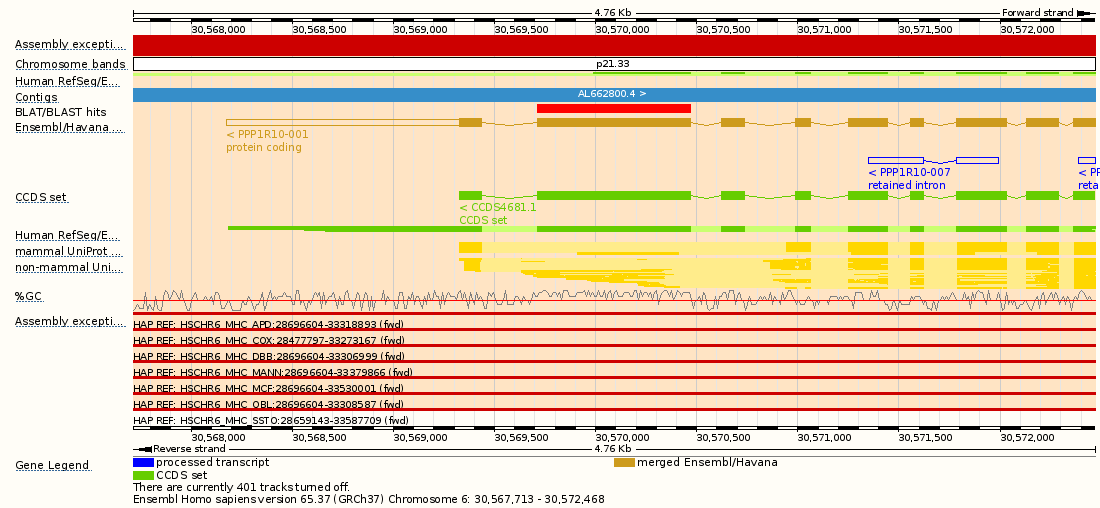
Еще ниже на следующей схеме зеленой лентой обозначена кодирующая ДНК из этого участка из EMBL/NCBI, выровненная с геномом.
А красной линий под ней - тот участок, который кодирует белок.
Все гены оснащены ссылками на различные базы данных, в частности, кодирующие белок - на UniProt (необходимо навести курсор для появления всплывающего окна).
Помимо поиска гомологичного участка посредством BLAST/BLAT можно попробовать ввести названия гена - в моем случае FB19 - в строку быстрого поиска по какому-то из геномов - я выбрала человека.
Выдача будет организована в том же формате, что и была выше, т.е. так же наклядно представлено расположение гена в геноме и системе контигов.
В разделе Genetic Variation можно посмотреть, как могут варьировать определенные нуклеотиды среди индивидуумов вида. Например, фрагмент моего объкта - человеческого гена FB19.
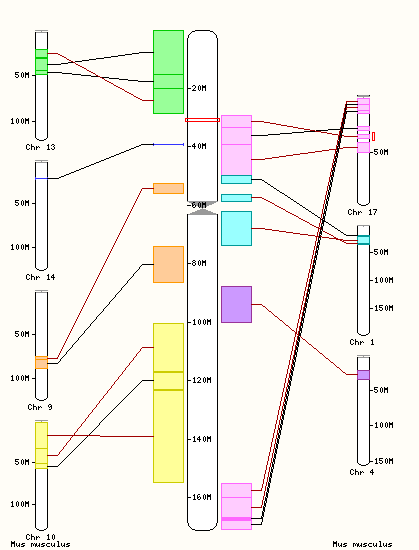
Так же очень интересно, если кликнуть на сслыку sinteny слева, то можно посмотреть сходство групп сцепления у разных организмов.
Весьма полезным мне представляется раздел Help & Documentation , где можно найти подробную и понятную информацию
относительно того, как работать с теми или иными сервисами . Что меня приятно удивило, так это ролики из YouTube в подразделе Tutorials,
где ясно объясняется, как работать с сервисом и как анализировать выдачу, что показалось мне очень привлекательным особенно для написания своей курсовой.
2. Другие геномные браузеры
1.UCSC - по сравнению с "Ансамблем" визуализация результатов не такая легкая на восприятие, к тому же нет "Help".
Но сразу представлены данные сравнительной геномики. Так, для меня показалось интересным, что моего гена нет у слона, ноесть у мыши например.
2.NCBI - достаточно удобный интерфейс, практически такой же, как и в "Ансамблем", но нет, к сожалению, таких же понятных Tutorials.
3.Vega - очень похожа на "Ансамбль", но меньше инструментов для работы.
© Garanina Irina