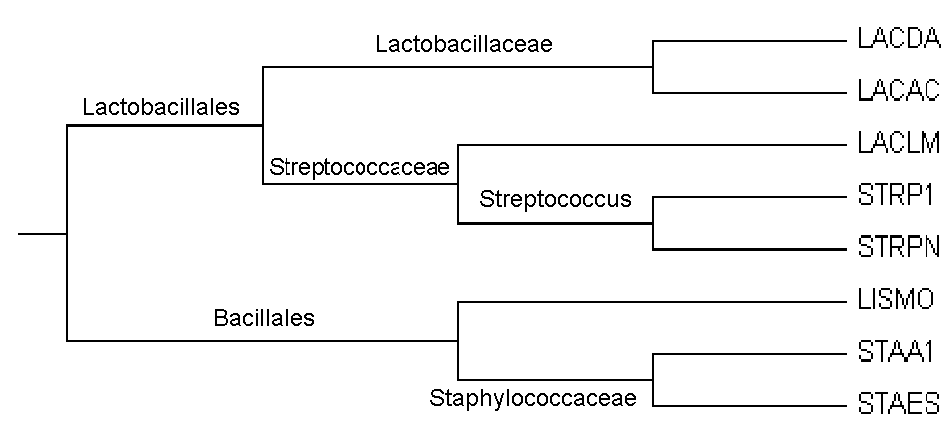
1) {LACDA,LACAC}{LACLM,STRP1,STRPN,LISMO,STAA1,STAES}
2) {LACLM,STRP1,STRPN}{LISMO,STAA1,STAES,LACDA,LACAC}
3) {LACDA,LACAC,LACLM,STRP1,STRPN}{LISMO,STAA1,STAES}
4) {STRP1,STRPN}{LACDA,LACAC,LACLM,LISMO,STAA1,STAES}
5) {LACLM,STRP1,STRPN,LISMO}{STAA1,STAES,LACDA,LACAC}
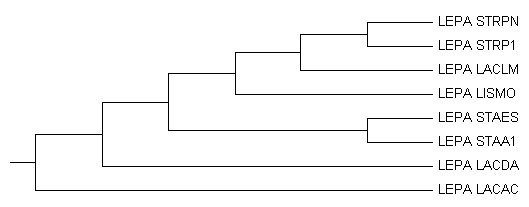
1) {LACDA,LACAC}{LACLM,STRP1,STRPN,LISMO,STAA1,STAES}
2) {LACDA,LACAC,LACLM,STRP1,STRPN,LISMO}{STAA1,STAES}
3) {LACDA,LACAC,LACLM,STRP1,STRPN}{LISMO,STAA1,STAES}
4) {STRP1,STRPN}{LACDA,LACAC,LACLM,LISMO,STAA1,STAES}
5) {LACDA,LACAC,LACLM}{STRP1,STRPN,LISMO,STAA1,STAES}