Гомологичное моделирование комплекса белка с лигандом.
1.
Мне был дан белок LYS_CRAGI, гомологичный белку форели 1lmp с известной структурой комплекса с лигандом.
Задача по выравниванию и
изветной структуре с помощью программы Modeler смоделировать комплекс белка LYS_CRAGI с лигандом.
Для этого сначала построим выранивание в JalView
алгоритмом Clustal и сохраним с некоторыми модификациями в формате pir
Выравнивание
2.
Модифицируем координаты PDB
3.
Создание управляющего скрипта
1)С помощью Pymol определим атомы, образующие воородные связи с лигандом.
1. ('OD2:101:A', 'O6:130:A')
2. ('ND2:103:A', 'O7:130:A')
3. ('OH:62:A', 'O6:130:B')
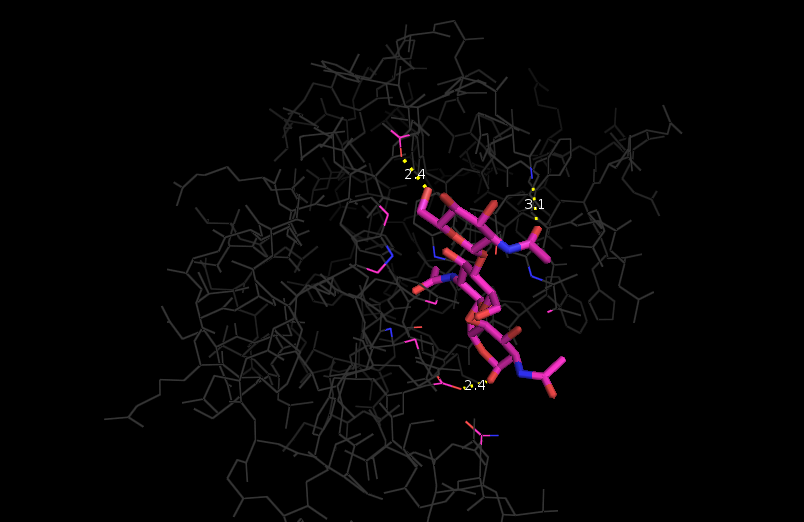
по выравниванию нашла те же атомы в моем белке
2)Сам скрипт
4.
Результаты
Получили 5 моделей
модель1
модель2
модель3
модель4
модель5
Чтобы сравнить их визуально, выравняла их в PyMol
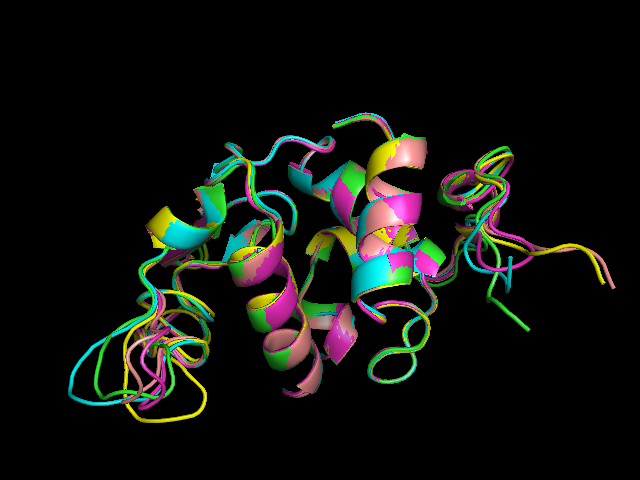
Видно, что се спирали хорошо накладываются друг на друга и лиганд везде расположен там, где надо, поэтому сложно оценить какая модель лучше, для этого используем специальные критерии.
Для анализа полученных структур я выбрала RMS Z-score for bond lengths, RMS Z-score for bond angles
1. 1.039 1.462
2. 1.067 1.500
3. 1.044 1.459
4. 1.036 1.405
5. 1.044 1.431
1lmp 0.839 1.752
Лучшаую модель выбрать сложно, так как по длине связи лучшая будет 4, но она же и худшая по углу, поэтому я выбрала что то среднее
Получилась лучшая модель 1
© Garanina Irina

