1. Подготовка файлов.
В банке pdb находим SMILES нотацию для NAG: nag.smi
C помощью obgen строим 3D структуру этого сахара в pdb формате:
obgen nag.smi > nag.mol
babel -imol nag.mol -opdb nag.pdb
nag.mol
nag.pdb
Создаем pdbqt файл лиганда:
python /home/preps/golovin/progs/bin/prepare_ligand4.py -l nag.pdb
nag.pdbqt
Создаем pdbqt файл белка:
python /home/preps/golovin/progs/bin/prepare_receptor4.py -r seq.B99990001.pdb
seq.B99990001.pdbqt
Файл с параметрами докинга:
vina.cfg
2. Докинг.
vina --config vina.cfg --receptor seq.B9990001.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
babel -ipdbqt nag_prot.pdbqt -opdb nag_prot.pdb
Энергии 3ёх лучших расположений и геометрическая разница между ними
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.9 0.000 0.000
2 -6.9 2.202 5.067
3 -6.7 2.200 2.889
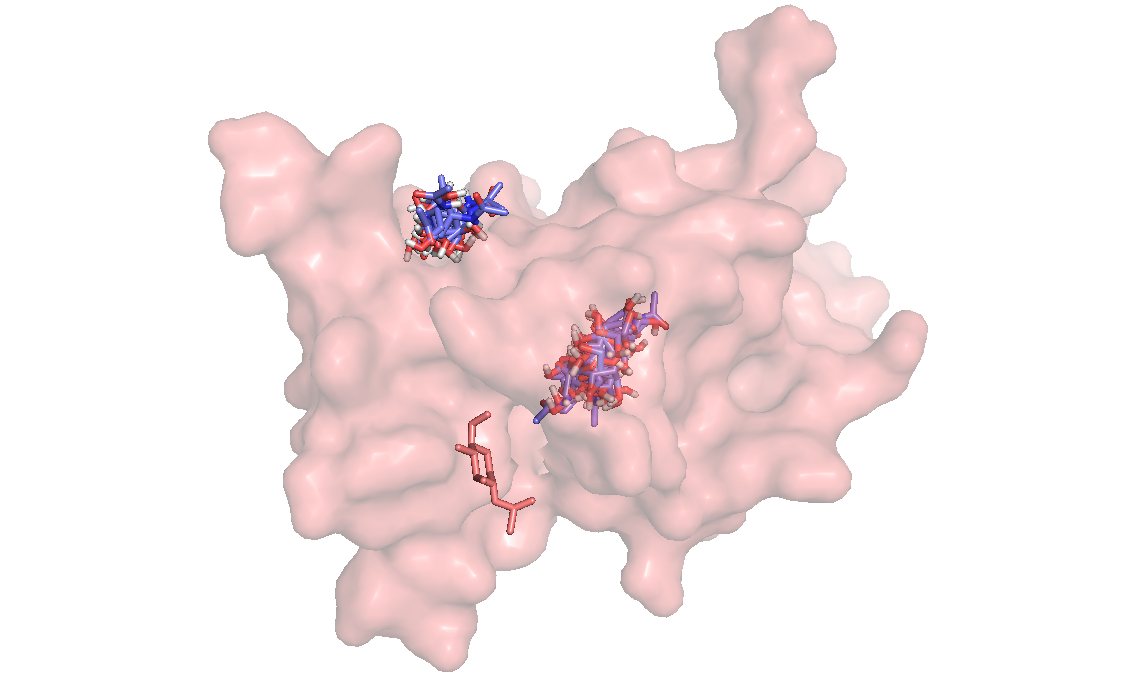
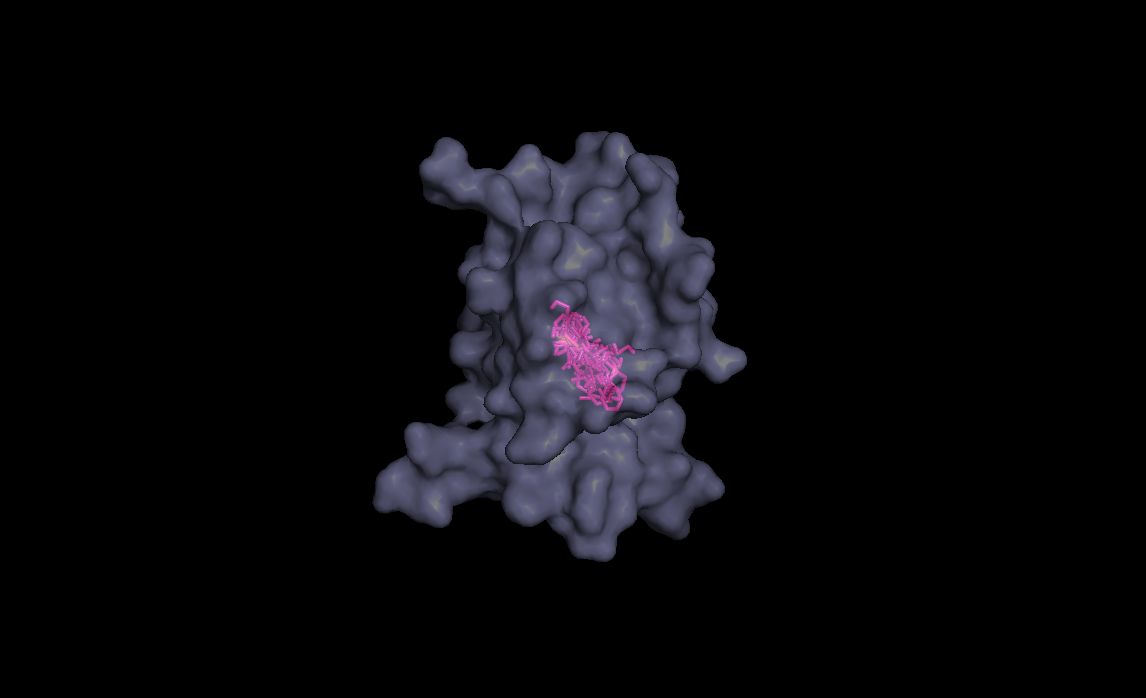
здесь видно, что не один из лигандов, которые мы получили в результате докинга не попал в нужное место, так как там находится сам лиганд
Что бы сам лиганд не мешал, я удалила его и в новом докинге лиганд попал туда куда надо
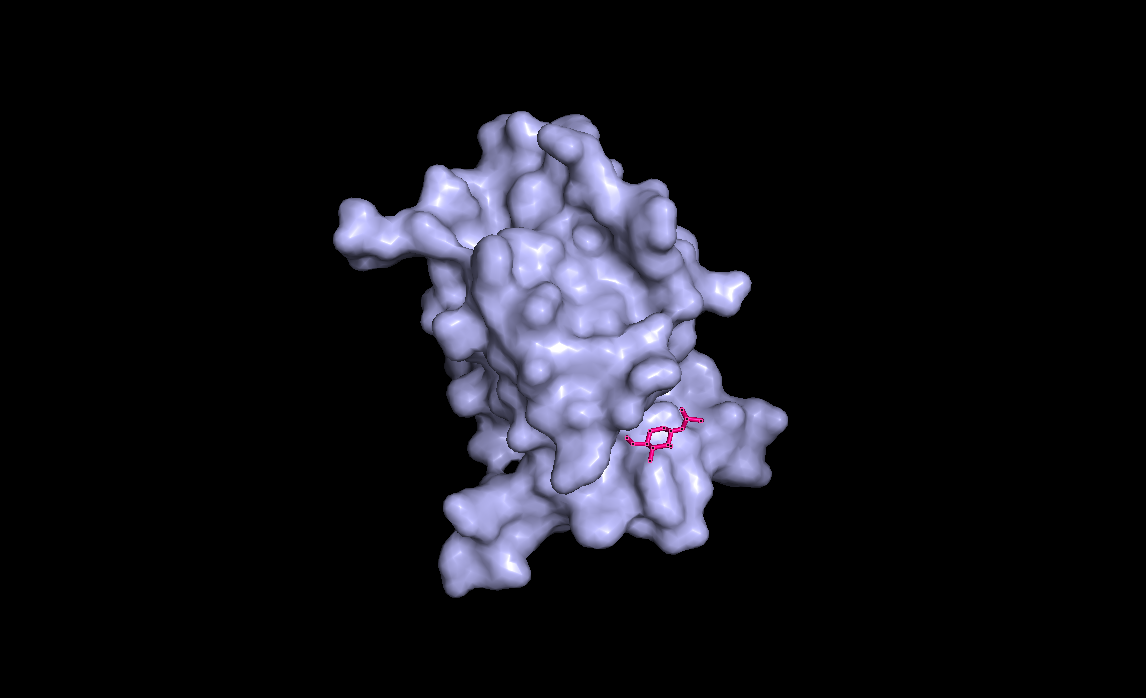
Это положение лиганда, расчитанное Modeller, возьмем его за образец, так как в реальном белке лиганд в 3 раза больше
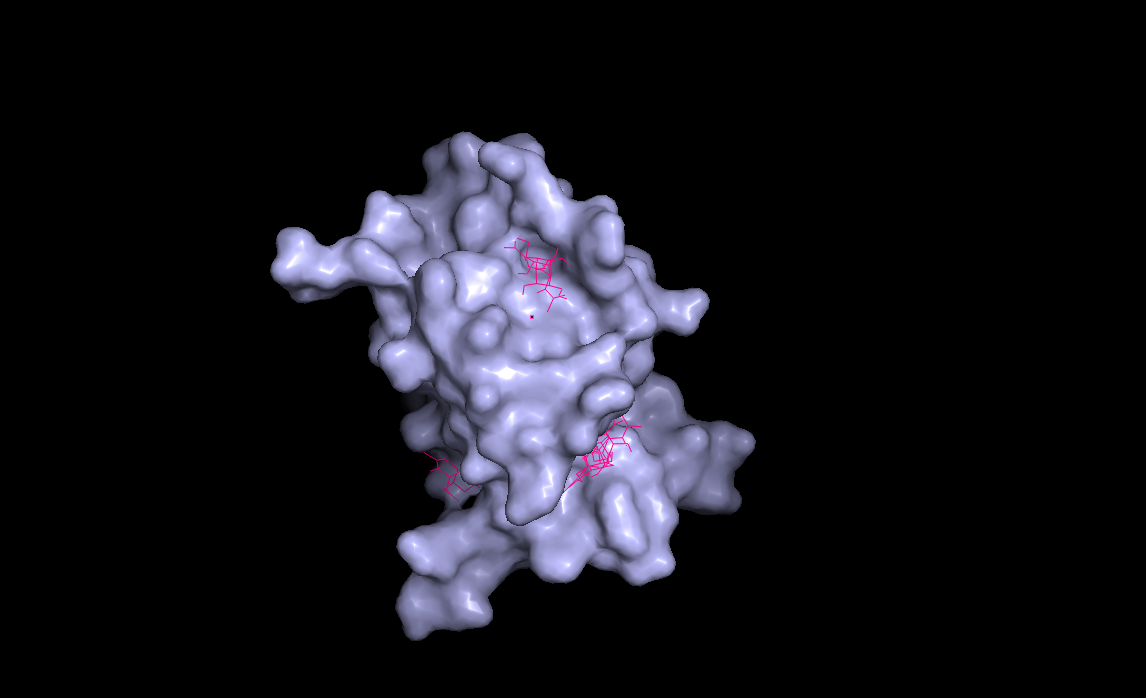
Мы видим, что в части состояний лиганд таки попал в нужное место
3. Докинг с учетом подвижности боковых радикалов белка
Сначала разобьем белок на две части, подвижную и неподвижную. Для подвижной
части выберем 2 аминокислоты, которые использовали в прошлом задании для позиционирования лиганда: TYR107_ARG105
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r seq.B999990001.pdbqt -s TYR107_ARG105
Докинг
vina --config vina.cfg --receptor seq.B99990001_rigid.pdbqt --flex seq.B99990001_flex.pdbqt --ligand nag.pdbqt --log nag_prot_flex.log --out nag_prot_flex.pdbqt
Энергии 3ёх лучших расположений и геометрическая разница между ними
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.8 0.000 0.000
2 -6.8 1.602 3.670
3 -6.4 1.679 3.869
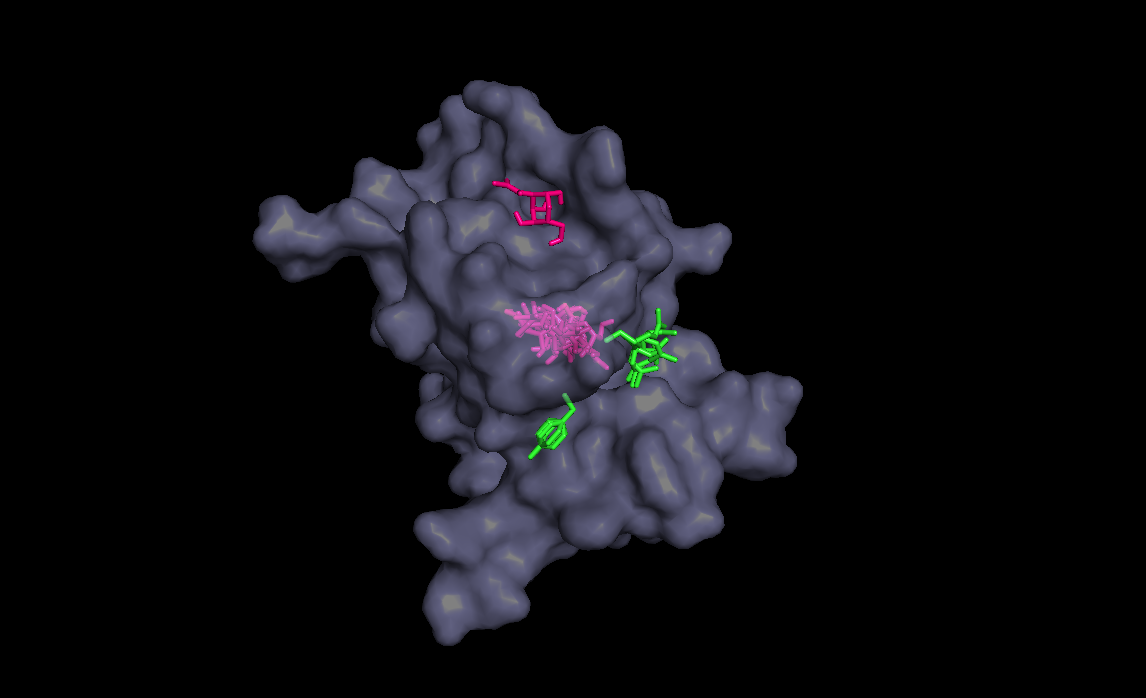
Мы видим, что ни один лиганд не попал туда куда надо, возможно, как раз таки излишняя подвижность радикалов ему и помешала, или стоило сделать подвижными не только эти радикалы но и еще некоторые вокруг предполагаемого места связывания
По времени такой докинг немного дольше обычного
4. Докинг с измененнным радикалом
Для следующего задания использовался небольшой скриптик
import os
a=['oh', 'h', 'ph', 'n']
os.system('export PATH=${PATH}:/home/preps/golovin/progs/bin')
for i in a:
os.system('obgen '+i+'.smi > '+i+'.mol')
os.system('babel -imol '+i+'.mol -opdb '+i+'.pdb')
os.system('python /home/preps/golovin/progs/bin/prepare_ligand4.py -l '+i+'.pdb -o '+i+'.pdbqt')
os.system('vina --config vina.cfg --receptor seq.B99990001.pdbqt --ligand '+i+'.pdbqt --out '+i+'_prot.pdbqt --log '+i+'_prot.log')
os.system('vina --config vina.cfg --receptor seq.B99990001_rigid.pdbqt --flex seq.B99990001_flex.pdbqt --ligand '+i+'.pdbqt --out '+i+'_prot_flex.pdbqt --log '+i+'_prot_flex.log')
Метильный радикал в этой группе заменили на H3C(=O)NH :
-H h.smi
-OH oh.smi
-NH2 n.smi
-фенил ph.smi
Обычный докинг
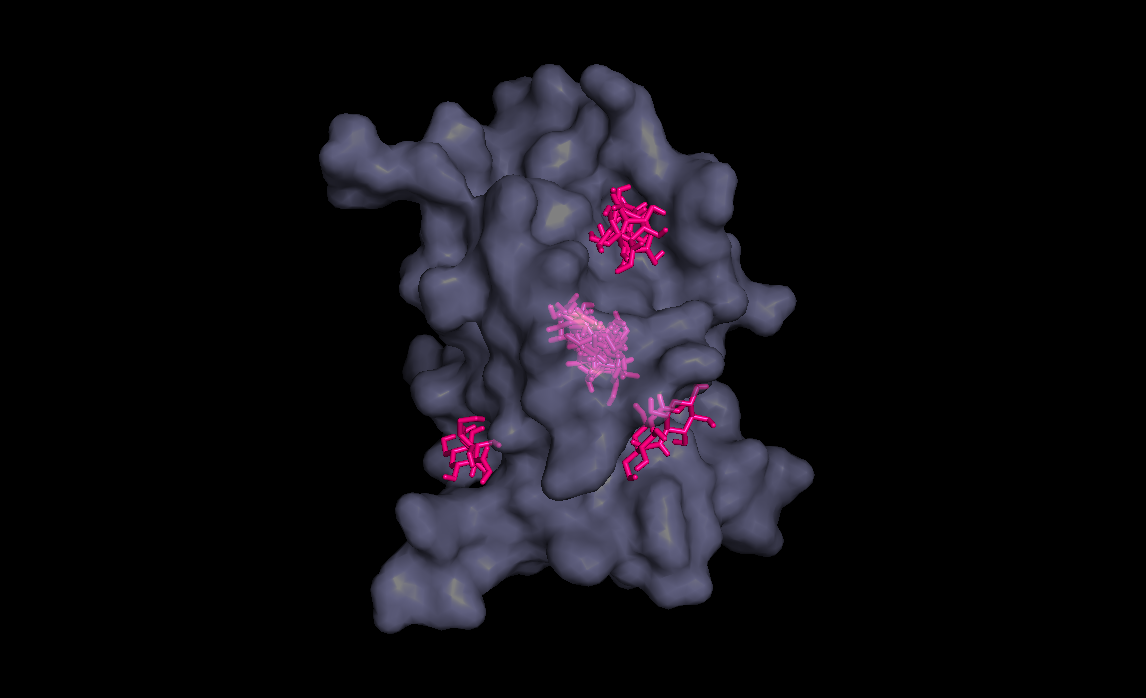
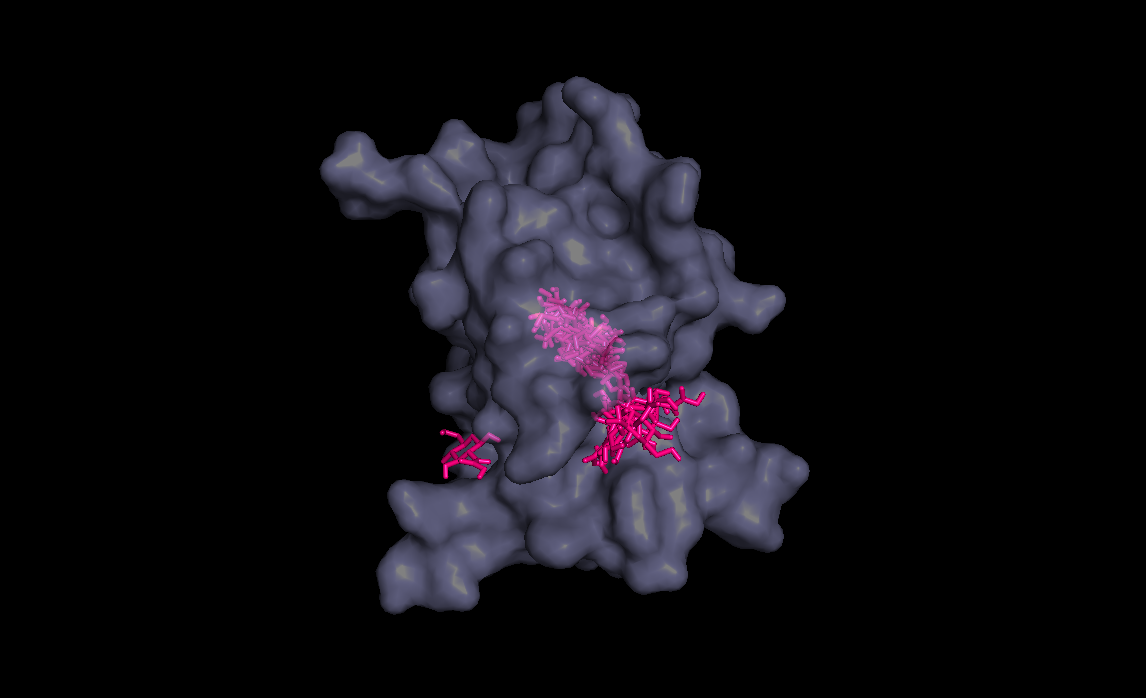
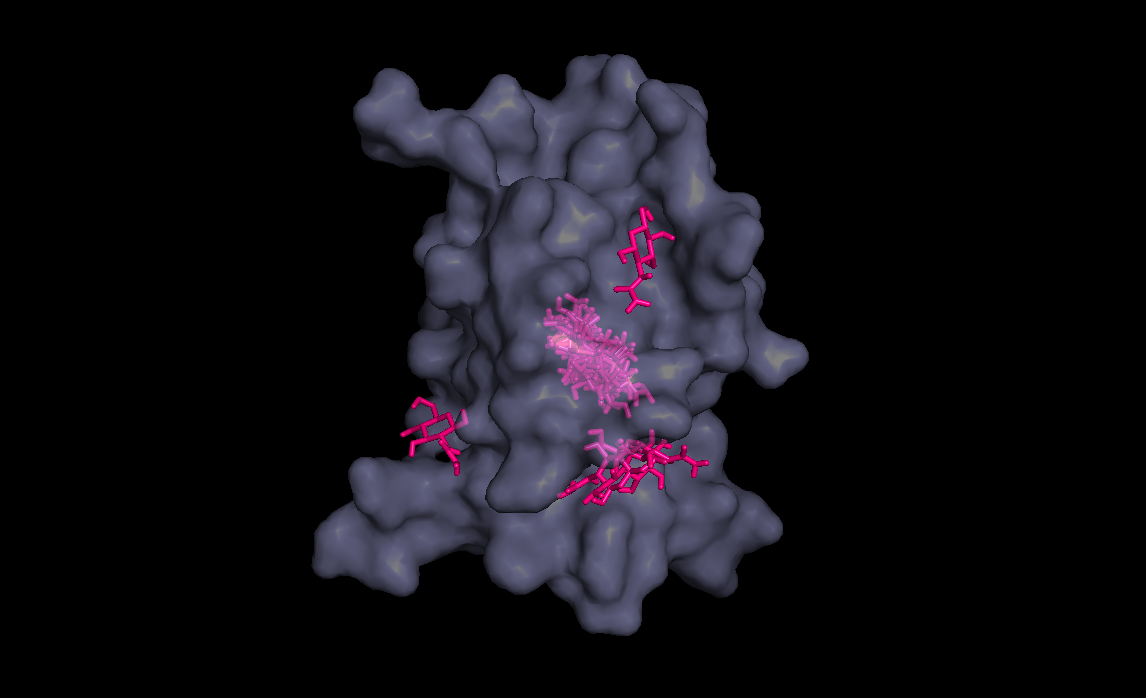
Три лучшие конформации для кадого лиганда :
| конформация |
Радикал |
|||
| H |
OH |
Nh2 |
Ph |
|
|
|
|
|
|
| 1 |
-6.1 |
-6.8 |
-6.6 |
-8.1 |
| 2 |
-6.0 |
-6.6 |
-6.6 |
-7.9 |
| 3 |
-5.9 |
-6.6 |
-6.6 |
-7.7 |
В таблице мы видим, что положение разных модифицированных лигандов отличаются. Лучше всего докинг прошел с радикалом ОН,
так как в большинстве состояний лиганд попал туда куда надо и энергия у состояний низкая по сравнению с другими радикалами
С радикалом Ph лиганд вообще ни в одном состоянии не попал в нужную ямку
С учетом подвижностей
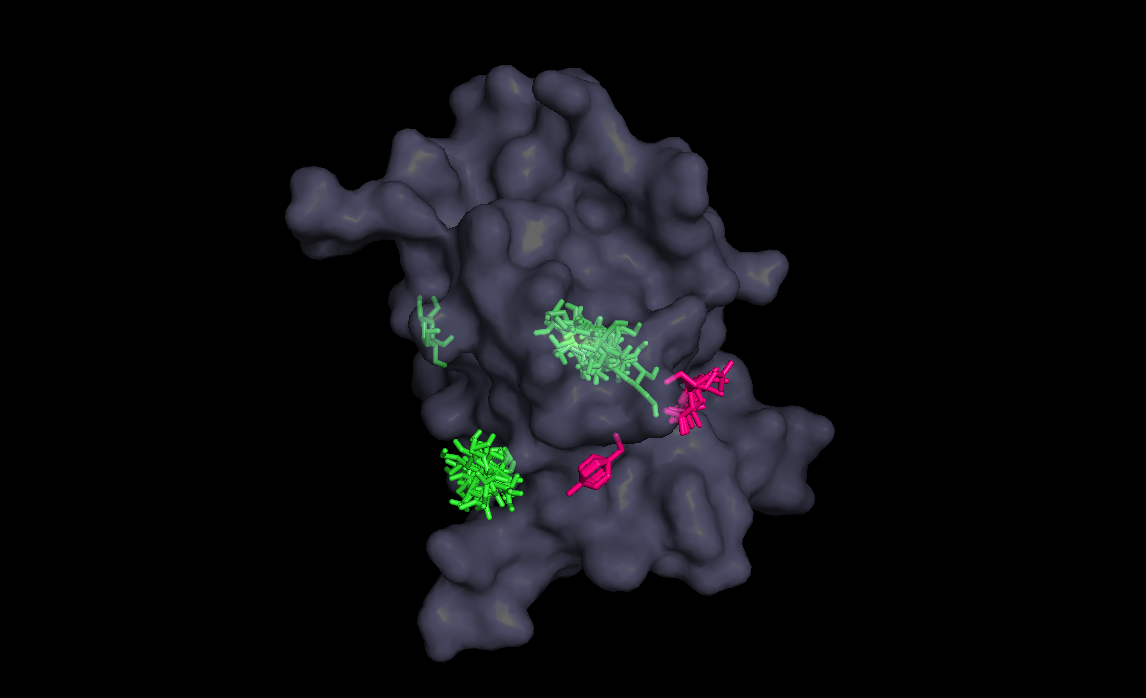
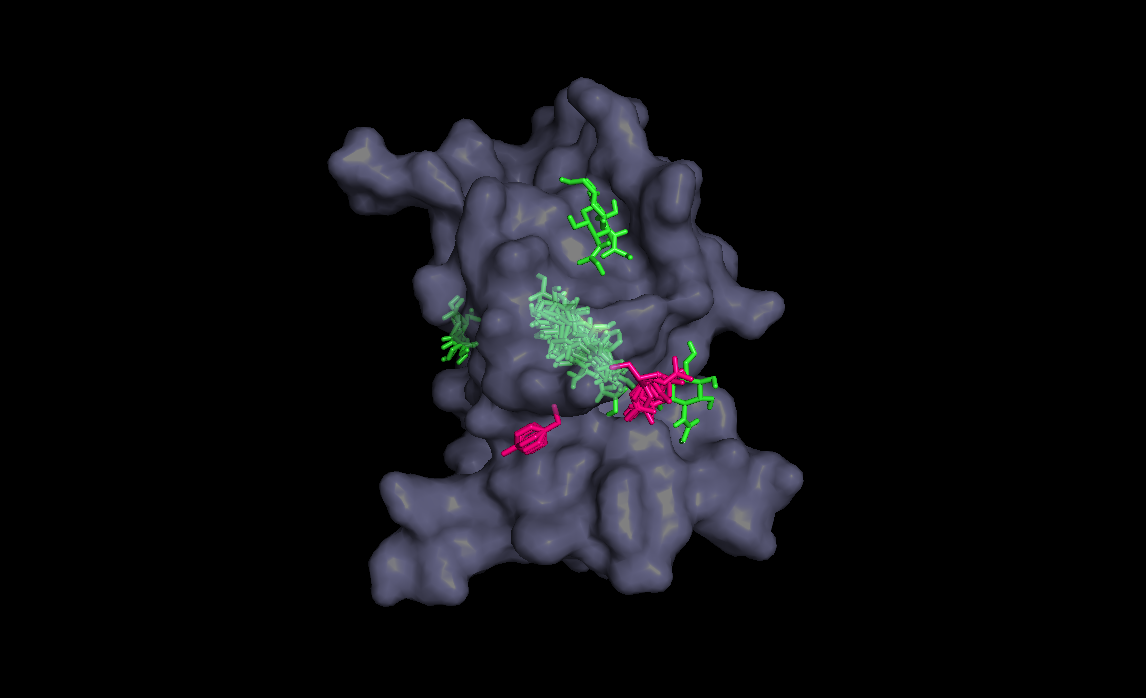
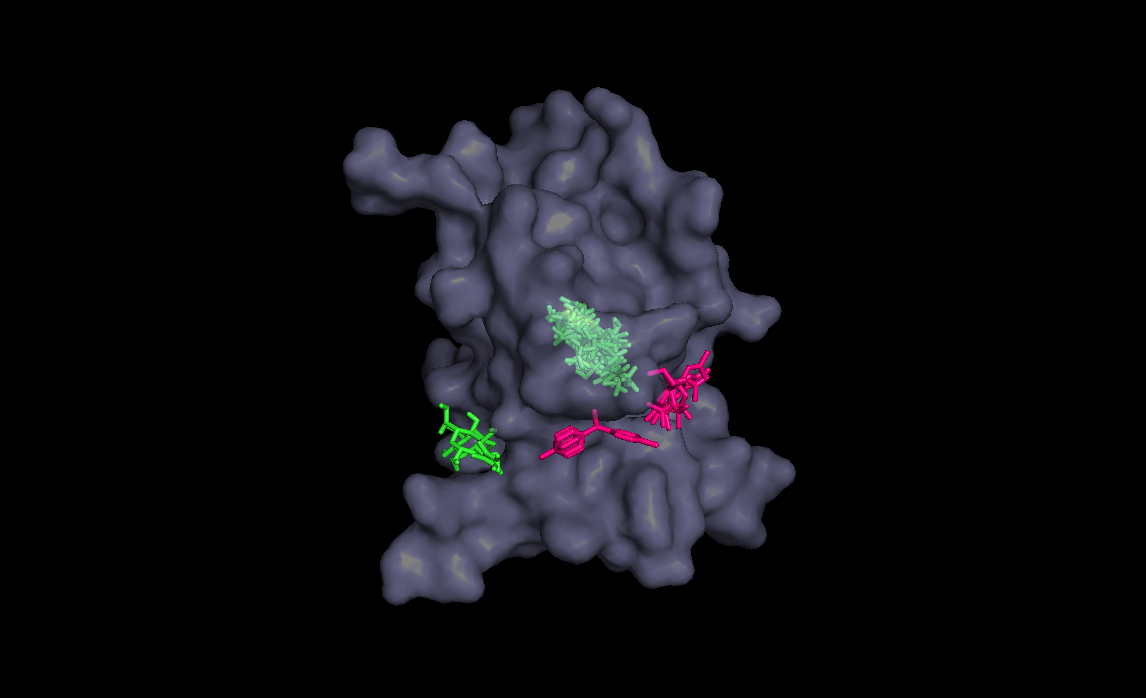
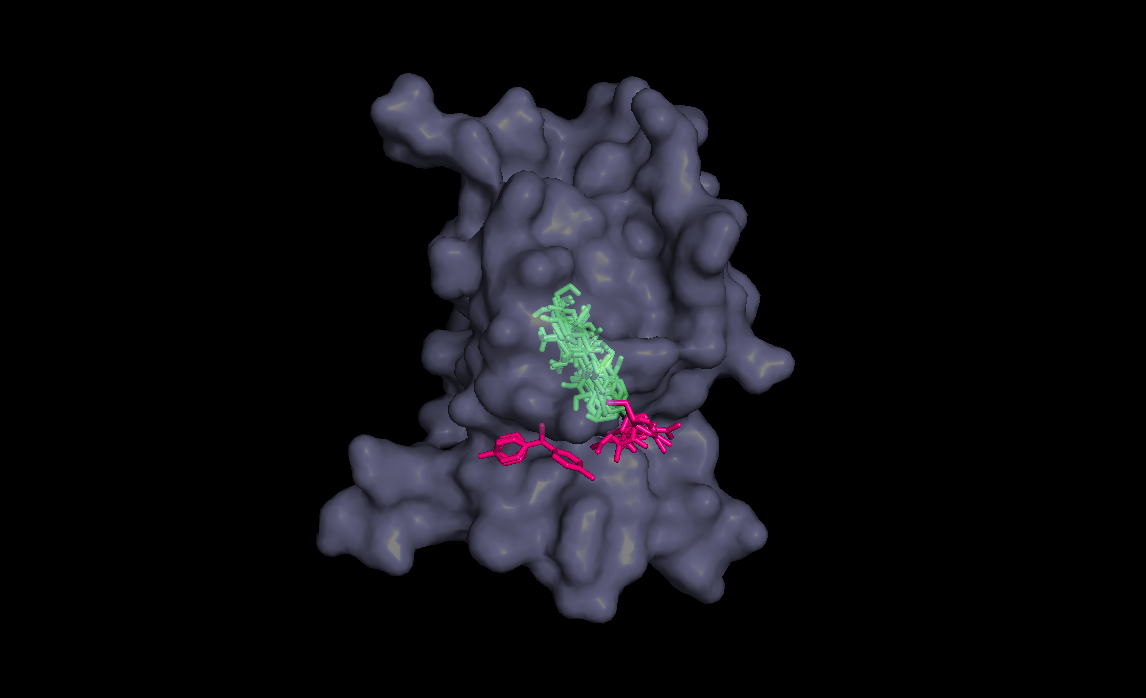
Три лучшие конформации для кадого лиганда :
| конформация |
Радикал |
|||
| H |
OH |
Nh2 |
Ph |
|
|
|
|
|
|
| 1 |
-5.9 |
-6.6 |
-6.7 |
-8.2 |
| 2 |
-5.9 |
-6.5 |
-6.6 |
-8.0 |
| 3 |
-5.9 |
-6.5 |
-6.6 |
-7.5 |
В этой таблице мы видим, что ни один лиганд ни в одном состоянии не встал в нужное место, как и в лучае с немодифицированным радикалом
хотя в случае OH лиганд уже близок к нужному положению, что уже неплохо