Макромолукулярный доккинг
1. Файл амилазы после добавления водородов
2. Амилаза и камелид после модификации mark_sur, который ,по-видимому, добавляет каждой группе заряды из файла uniCHARMM.
3. Доккинг zdock -R .pdb -L lig.pdb
4. Обработка результатов:
create.pl zdock.out
ls complex*pdb >list
zrank list
5. Подсчет квадратов отклонения
я написала срипт, который считает rmsd командой
g_rms -s ../1KXT5.pdb -f complex.39.pdb -o 39
и получила, что самое маленькое значение у комплекса 1826 rmsd=2,393703
Пространственное выравнивание этого комплеса с белком 1KXT
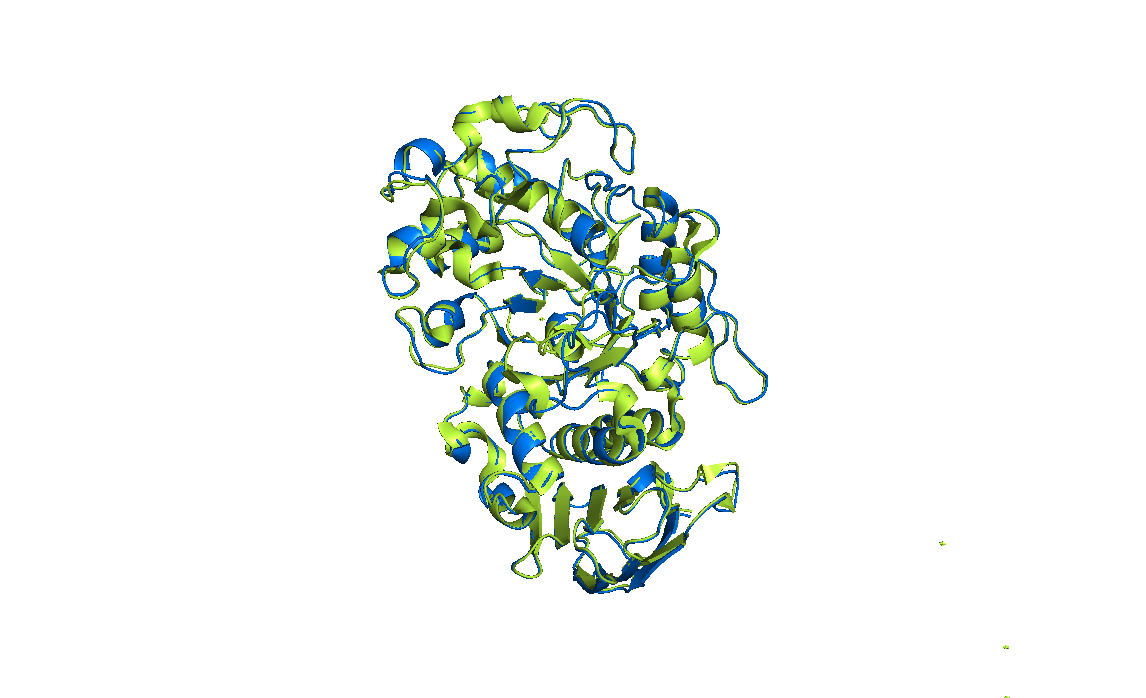
Видно, что не так уж плохо выровнялось, спиральки и листы совпадают почти
© Garanina Irina
