
Для выполнения задания был выбран домен Sugarporin_N (PF11471). Данный домен имеет следующие параметры:
Далее была выбрана доменная архитектура, содержащая 2 домена: Sugarporin_N и Porin_8 (Рис.1). Она встречается у 26 белков.

Затем были скачаны последовательности full в формате fasta. Из них был получен список AC всех белков (full) и белков, содержащих выбранную доменную архитектуру (domain). Далее из full были выбраны последовательности с доменной архитектурой и выровнены в Jalview(ссылка).
Далее я провела ревизию выравнивания: убрала участки до начала первого домена и после второго домена, удалила фрагменты, которые имеют крупные делеции и удалила последовательности, совпадающие более чем на 90%. Получилось выравнивание из 19 последовательностей.
Для построения профиля были использованы следующие команды:
hmm2build HMM revision.fa
hmm2calibrate HMM
hmm2search --cpu=1 HMM Emelyanova-full-105.fasta > search.txt
Получили HMM профиль выбранной архитектуры и файл, содержащий 102 находки. На их основе была получена таблица с информацией о находках. В Excel были построены графики: ROC (Рис.2), распределение весов находок (Рис.3) и F1 (Рис.4).
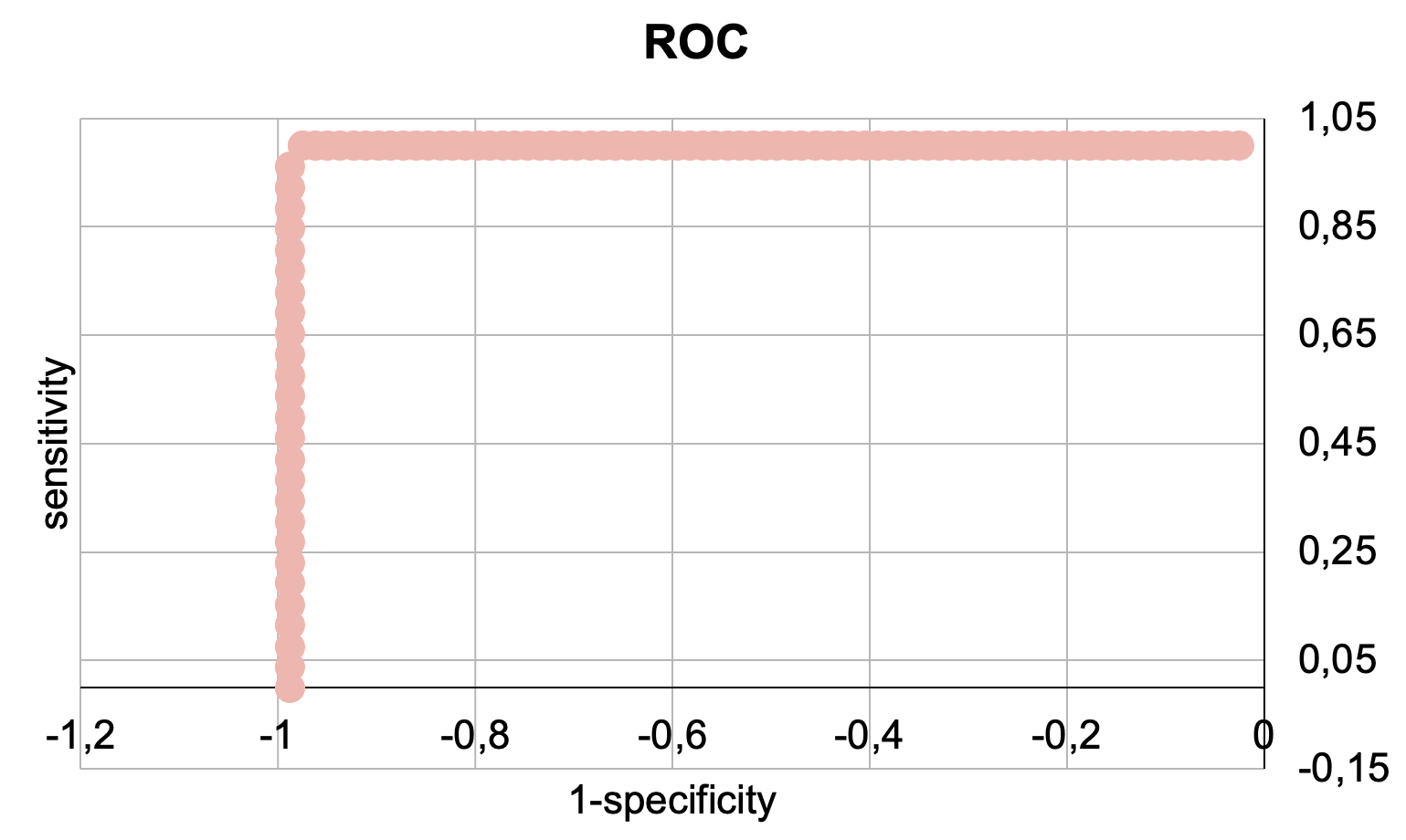
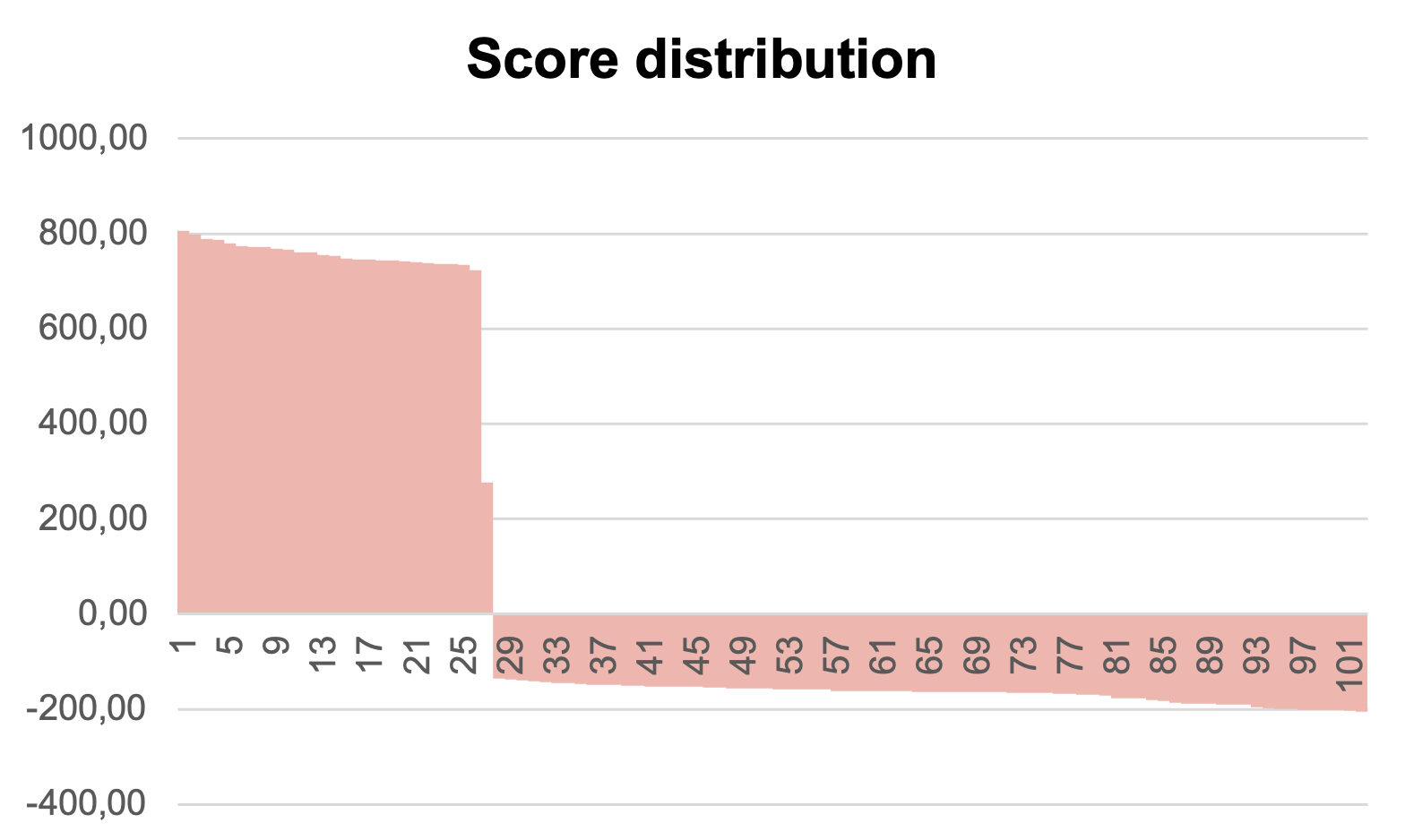
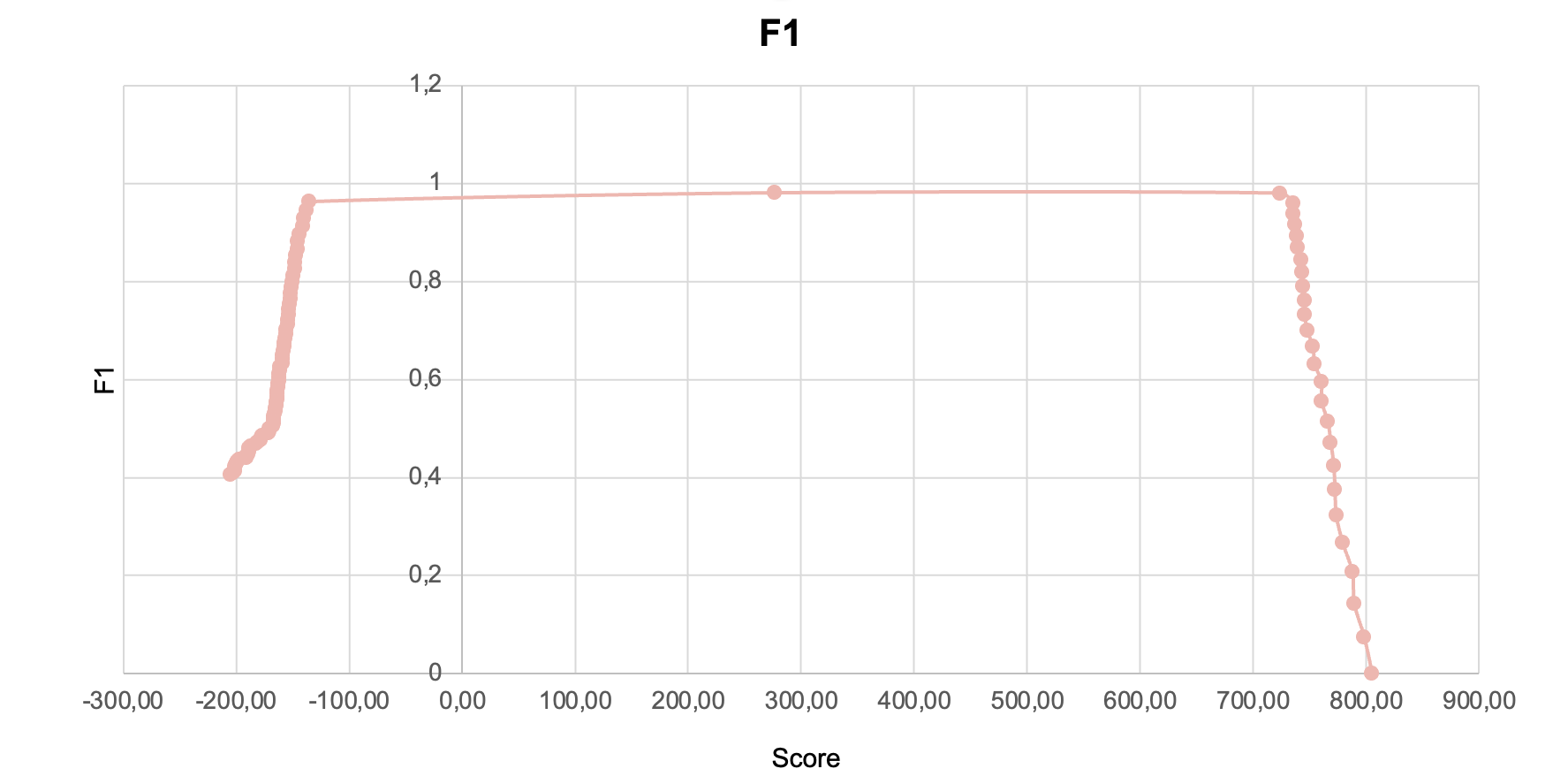
На графиках видно, что получился резкий скачок ROC кривой и весов находок. По графику F1 можно сказать, что порог веса, соответствующий наилучшему соотношению между специфичностью и чувствительностью, равен 723.4, что соответствует весу худшей правильной находки.