Домены и профили
Задание 1. Белки UniProt с заданным составом доменов Pfam.
Были выбраны домены Phage_CI_repr, Peptidase_S24 (PF07022, PF00717). 
PF00717 - Peptidase_S24 - 30760 последовательностей
PF07022 - Phage_CI_repr- 652 последовательности
database:(type:pfam pf07022) database:(type:pfam pf00717) taxonomy:"Bacteria [2]" - запрос в Uniprot.
Таблица с информацией о бактериальных белках с данной доменной архитектурой
Интервал типичных длин белков 234-240 аминокислот.
Задание 2. HMM профиль семейства белков.
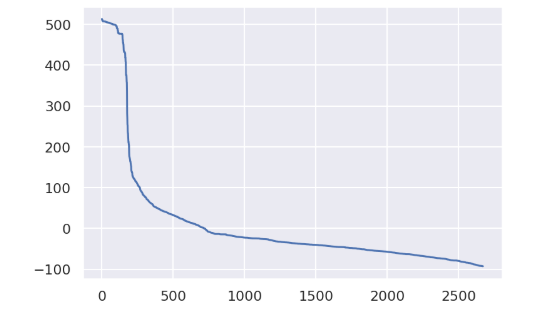
В предыдущем задании была построена гистограмма длин белков, были отобраны 42 белка длиной 234-240 аминокислот(в таблице отмечены оранжевым цветом в колонке selected),
их последовательности в Jalview были выровнены алгоритмом Muscle (with defolts). Выравнивание было отредактировано, я вырезала первые 20 колонок, так как
в них было много инделей в разных последовательностях и выравнивались они плохо, были вырезаны 13 последовательностей, которые выравнивались хуже всего.
Файл с выравниванием.Затем был построен НММ-профиль с помощью команд
hmm2build pr9.hmm alignment.fa
hmm2calibrate pr9.hmm
Файл, полученный в результате построения и калибровки профиля.
Для проверки построенного НММ-профиля были скачаны последовательности всех белков с доменом PF07022 из Uniprot.
hmm2search pr9.hmm uniprot-database_\(type_pfam+pf07022\).fasta - команда для поиска. Результаты поиска с E-value не выше 0.01 импортируем в
таблицу Excel и обрабатываем.
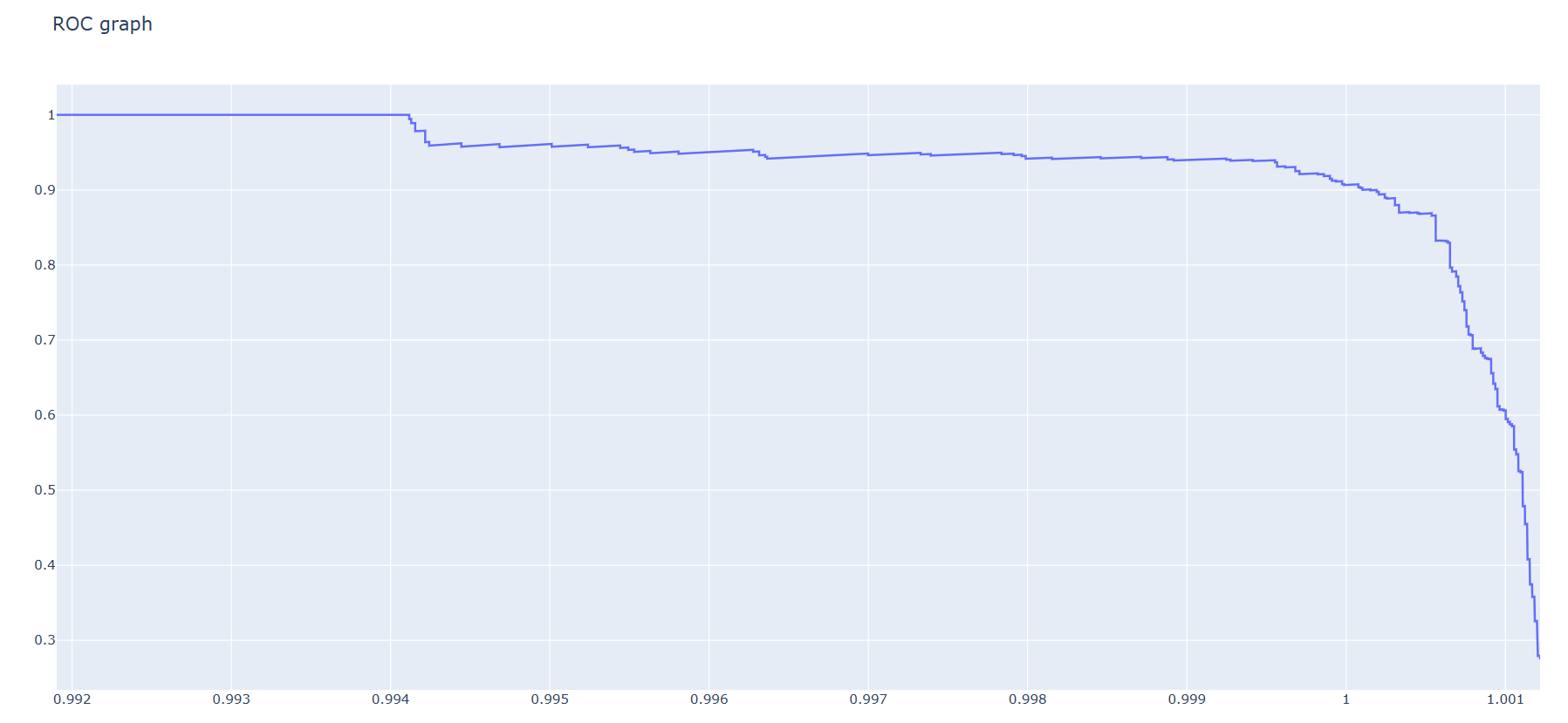
ссылка на таблицу (Лист1, там же выбор порога по F1).
В результате вычисления F1 оказалось, что оптимальный порог - вес -12.3 и e-value 9*10^-5. В таблице ниже указаны характеристики получившегося теста.
| Предсказаны | С нашей архитектурой | С иной архитектурой |
|---|---|---|
| Подтверждено | 673 | 127 |
| Отвергнуто | 45 | 663 |
Чувствительность теста составила 93,7 процентов, а специфичность 87,17 процентов.