
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Выравнивание и гомология. Jalview
Выравнивание последовательностей аминокислотных остатков в белке — оптимальное расположение сравниваемых последовательностей друг под другом, при котором наблюдается:
- максимальное количество совпадений остатков аминокислот;
- сопоставление гомологичных остатков;
- сопоставление остатков в одинаковом пространственном расположении;
- сопоставление остатков, имеющих одинаковую функциональную нагрузку.
В этот раз мы будем работать с множественным выравниванием. Для просмотра и редактирования выравнивания мы использовали програмное обеспечение Jalview. Для следующего множественного выравнивания была выбрана раскраска ClustalX и консервативность >70%. Сначала мы нашли два вертикальных блока (рис. 1 и 2), в которых предполагается гомология между остатками из всех последовательностей. Но при более внимательном рассмотрении рисунка 2 мы можем обратить внимание, что три последовательности: 3,4,8 и 12 сверху, выделяются на фоне всего выравнивания тем, что имеют много меньше совпадений аминокислотных остатков. Проект в формате jar вы можете скачать здесь.

|

|
Рис.1. Вертикальный блок множественного выравнивания. Позиции с 329 по 346. Рисунок получен с помощью Jalview. |
Рис.2. Вертикальный блок множественного выравнивания. Позиции с 365 по 406. Рисунок получен с помощью Jalview. |
Для второго вертикального блока (рис.2.) была составлена таблица консервативности (таблица 1.)
Таблица 1. Консервативность позиций 365-406 из множественного выравнивания
| Параметр | Количество позиций | % |
| Всего позиций | 42 | 100 |
| Абсолютно консервативных позиций | 6 | 14.3 |
| Абсолютно функционально консервативных позиций | 9 | 21.4 |
| Консервативных на 70% позиций | 13 | 31 |
| Консервативных функционально на 70% позиций | 13 | 31 |
Исходя из данной таблицы мы можем сделать вывод, что 97.7% позиций из выбранного блока имеют консервативность выше 70%, а это означает, что оставшиеся 2.7% позиций также гомологичны и сходны между собой.
Два выбранных вертикальных блока располагаются близко: всего 18 позиций между ними (рис.3.), 3 из которых содержат гэпы (16.7%). Проект в формате jar вы можете скачать здесь.

|
Рис.3. Взаимное расположение двух вертикальных блоков множественного выравнивания. Позиции с 329 по 406. Рисунок получен с помощью Jalview.
Далее в выравнивание мы добавили еще одну последовательность и выровнили ее вручную относительно второго вертикального блока (рис.2.). В результате этого выравнивания мы получили 16 (38.1%) совпадающих позиций (рис.4.). Проект в формате jar вы можете скачать здесь.

|
Рис.4. Вертикальный блок множественного выравнивания с добавленной последовательностью. Позиции с 365 по 406. Рисунок получен с помощью Jalview.
Консенсусная последовательность данного выравнивания:
TRPAGDGTFQKWAAVVVPSGEEQRYTCHVEHEGLPEPLTLRW.
Ее графическое представление – LOGO рассматриваемого блока представлено на рисунке 5.

|
Рис.5. LOGO второго блока (позиции с 365 по 406). Рисунок получен с помощью сервиса http://weblogo.berkeley.edu/logo.cgi.
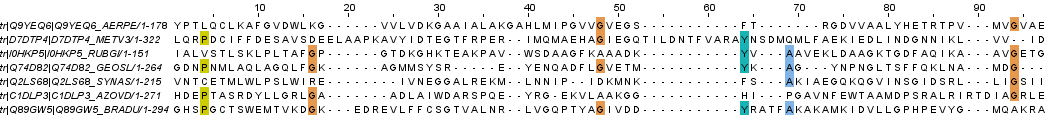
Следующим действием мы построили множественное выравнивание заведомо негомологичных последовательностей. Было выбрано 6 белков со следующими идентификаторами в базе данных Uniprot: Q9YEQ6, D7DTP4, I0HKP5, Q74D82, Q2LS68, C1DLP3, Q89GW5. Далее было построено выравнивание, для которого выбрана раскраска ClustalX и консервативность >50%. Полученное выравнивание состоит из 406 позиций, из которых всего лишь 12 являются консервативными на ≥50% (рис.6.). Они находятся далеко друг от друга и не образуют вертикальных блоков. Также мы можем наблюдать огромное количество гэпов. Именно подобного рода выравнивание говорит о негомологичности исследуемых последовательностей. Проект в формате jar вы можете скачать здесь.
|
Рис.6. Выравнивание негомологичных последовательностей. Позиции с 199 по 295. Рисунок получен с помощью Jalview.
Дата последнего изменения: 09.05.2014