
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
BLAST. Поиск гомологов белка АТФазы VirB4
Программа BLAST (Basic Local Alignment Search Tool) на сайте NCBI служит для поиска последовательностей белков или нуклеиновых кислот, сходных с входной последовательностью, то есть позволяет найти гомологов белка. Для поиска белков со сходными последовательностями используют алгоритм BLASTP (protein-protein BLAST).
С помощью данного алгоритма мною был осуществлен поиск гомолов в базе данных swissprot для белка АТФазы VirB4, полученного из бактерии Thermoanaerobacter pseudethanolicus. Результатом поиска стали 18 последовательностей, для каждой из которых в таблице представлена следующая информация:
- поле Description содержит имя последовательности белка и, возможно, краткое описание, например, из какого организма белок был получен;
- поле Max score и поле Total score содержат нормированный максимальный и общий вес выравнивания соответственно. Если в последовательности ≥ 2 участков локального сходства, то значения весов в данных полях будет различно;
- в поле Query cover в поцентах указано какую часть исходной последовательности (query) покрывает находка;
- поле E-value содержит величину, отражающую количество находок с такими же параметрами в базе данных случайных последовательностей. Чем меньше значение E-value, тем больше значимость находки;
- в поле Ident указан процен совпавших аминокилотных остатков;
- поле Accession содержит идентификатор последовательности.
Была выбрана одна из находок с АС: Р18004, для нее вся вышеуказанная информация представлена в таблице 1.
Таблица 1. Информация о находке с АС: Р18004
| Description | Max score | Total score | Query cover | E-value | Ident | Accession |
| RecName: Full=Protein TraC [Escherichia coli K-12] | 76.6 | 76.6 | 64% | 2e-13 | 23% | gi|136145|sp|P18004.1|TRAC_ECOLI |
Выбранная последовательность длиной 875 а.о. содержит один участок локального сходства, расположенный с 403 по 772 а.о., а для исходной последовательности этот участок находится с 164 по 547 (общая длина последовательности 594 а.о.). На рисунке 1 представлено выравнивание участка query c выбранной находкой, также вы можете скачать его в формате fasta. Вес выравнивания равен 76.6, количество позиций — 406, из которых в 92 (23%) позициях аминокислотные остатки совпадают, 170 (41%) позиций имеют положительное значение в матрице выравнивания, а 58 (14%) содержат гэпы.

|
Рис.1. Выравнивание участка query c выбранной находкой (АС: Р18004). |
Далее была построена карта локального сходства участка query c выбранной находкой (рис.2.). Участок query располагается по оси абцисс, а участок локальньного сходства из выбранной находки по оси ординат. Исходя из полученной карты, можно сделать вывод, что рассматриваемые участки локального сходства практически полностью гомологичны. Имеется 15 точек разрыва линии, а значит в соответствующих местах последовательностей произошли вставки или делеции. В выравнивании данные разрывы отображаются гэпами.
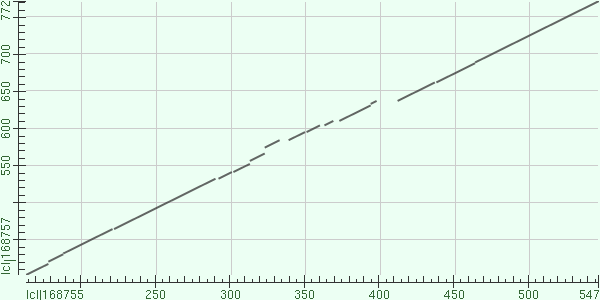
|
Рис.1. Кaрта локального сходства частка query c выбранной находкой (АС: Р18004). Рисунок получен с помощью функции bl2seq на сайте NCBI |
Также для исследуемого белка АТФазы VirB4 из бактерии T. pseudethanolicus были найдены гомологи в двух других доменах. Для этого в окне запуска BLAST в поле Organism было введено соответствующие надцарства: Eukaryota и Аrchaea.
Результатом первого поиска стали 6 последовательностей, из которых только у двух значение E-value было меньше 0.001. Стандартная информация об этих находках представлена в таблице 2. Белок первой находки (белок TRSE) длинной 337 а.о. получен из микроспоридии, а гипотетический белок из второй находки длинной 840 а.о. получен из желтолихорадочного комара.
Таблица 2. Информация о двух эукариотических гомологах для белка АТФазы VirB4
| Description | Max score | Total score | Query cover | E-value | Ident | Accession |
| TRSE protein [Enterocytozoon bieneusi H348] | 62.8 | 62.8 | 51% | 4e-08 | 22% | XP_002652324.1 |
| hypothetical protein AaeL_AAEL015530 [Aedes aegypti] | 58.5 | 58.5 | 48% | 2e-06 | 25% | XP_001647668.1 |
Результатом поиска гомологов в надцарсве Архей стало 100 последовательностей (E-value > 0.001). Из них были выбраны 12 последовательностей, информация о которых представлена в таблице 3, и по которым было построено множественное выравнивание (вы можете скачать выравнивание в формате fasta и проект в формате jar )
Таблица 3. Информация о 12 гомологах белка АТФазы VirB4 из надцарства архей
| Description | Max score | Total score | Query cover | E-value | Ident | Accession |
| hypothetical protein [candidate division DUSEL4 archaeon SCGC AAA011-L22] | 147 | 147 | 78% | 3e-36 | 27% | WP_018203263.1 |
| transfer complex protein [Candidatus Halobonum tyrrellensis] | 140 | 140 | 96% | 7e-34 | 26% | WP_023395986.1 |
| transfer complex protein [Haloferax prahovense] | 123 | 123 | 87% | 2e-28 | 25% | WP_008094506.1 |
| hypothetical protein [Natrialba taiwanensis] | 124 | 124 | 84% | 3e-28 | 25% | WP_006825893.1 |
| hypothetical protein Htur_4995 [Haloterrigena turkmenica DSM 5511] >ref|WP_012945967.1| hypothetical protein [Haloterrigena turkmenica] | 122 | 122 | 84% | 1e-27 | 25% | YP_003406397.1 |
| transfer complex protein [Haloarcula vallismortis] | 117 | 117 | 97% | 3e-26 | 24% | WP_004517778.1 |
| transfer complex protein [Haloarcula marismortui ATCC 43049] >ref|WP_004594535.1| transfer complex protein [Haloarcula] | 117 | 117 | 97% | 4e-26 | 24% | YP_134360.1 |
| Type IV secretory pathway VirB4 protein-like protein [Haloferax mucosum] | 117 | 117 | 94% | 4e-26 | 23% | WP_008319460.1 |
| Type IV secretory pathway VirB4 protein-like protein [Haloarcula hispanica ATCC 33960] >ref|YP_008873754.1| hypothetical protein HISP_16150 [Haloarcula hispanica N601] >ref|WP_014030626.1| Type IV secretory pathway VirB4 protein-like protein [Haloarcula hispanica] | 107 | 107 | 94% | 7e-23 | 22% | YP_004785728.1 |
| putative TrbE protein [Fervidicoccus fontis Kam940] >ref|WP_014557243.1| TrbE protein [Fervidicoccus fontis] | 103 | 103 | 93% | 8e-22 | 23% | YP_005841546.1 |
| transfer complex protein-like protein [Haloferax volcanii DS2] >ref|WP_004042170.1| transfer complex protein-like protein [Haloferax volcanii] | 90.9 | 90.9 | 42% | 7e-18 | 28% | YP_003536305.1 |
| type VI secretion protein [Aciduliprofundum boonei] | 87.8 | 87.8 | 79% | 6e-17 | 24% | WP_008085212.1 |
Дата последнего изменения: 12.05.2014