
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Мотивы. ProSite
Мотив — некий консервативный участок последовательности, сходный у белков, выполняющих одну функцию, который позволяет отличать классы белков друг от друга. Благодаря сервису ProSite возможен поиск мотивов в заданных последовательностях.
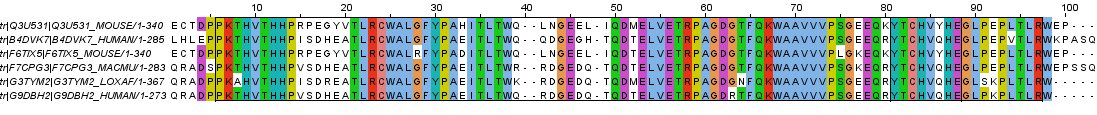
Из множественного выравния практикума "Выравнивание и гомология. Jalview" было выбрано 6 последовательностей со следущими идентификаторами в БД Uniprot: F7CPG3, G3TYM2, G9DBH2, Q3U531, B4DVK7, F6TIX5. С помощью первой опции поиска вышеуказанного сервиса было найдено две группы мотивов в каждой последовательности. Длина мотива из первой группы равна 101, из второго — 8. В множественном выравнивании последовательностей первая группа мотивов располагается с 314 по 405 позицию, вторая — с 389 по 386 позицию (см. рис.1). Проект в формате jar вы можете скачать здесь.
|
Рис.1. Фрагмент множественного выравнивания, содержащий два мотива. Мотивы выделены черными рамочками. Рисунок получен с помощью Jalview.
Для первой группы мотивов были составлены паттерны. Паттерн — общая формула одной группы мотивов, по которой осуществляется поиск гомологичных белков. Паттерны бывают сильными и слабыми. Чем слабее паттерн, тем больше белков находит сервис, но тем ниже вероятность гомологичности этих белков.
Сильный паттерн для первой группы мотивов выглядит следующим образом:
P-K-[TA]-H-V-T-X(0,3)-H(2)-P-[RIV]-[PS]-[ED]-[EY]-[AV]-X(0,1)-T-L-R-C-W-A-L-[GR]-F-Y-P-A-[DEH]-I-T-L-
-T-W-[QKR]-[LQR]-[DN]-G-E-X(0,4)-[DEG]-[QLH]-[IT]-Q-D-[TM]-E-L-V-E-T-R-P-A-G-D-[GR]-[TN]-F-Q-K-
-Q-A(2)-V(3)-P-[SL]-X(0,3)-G-[KE]-E-Q-[KR]-Y-T-C-H-V-[QY]-H-X(0,4)-E-G-L-[PS]-[KE]-P-[LV]-T-X(2)
С помощью второй опции поиска на сервере ProSite было подтверждено, что построенный сильный паттерн подходит для мотивов в избранных последовательностях. Далее был осуществлен поиск гомологичных последовательностей по этому паттерну (использовалась третья опция поиска). В базе данных Swissprot было найдено 112 мотивов в 112 последовательностях. Все найденные белки являются антигенами гистосовместимости различных животных (человека, обезьян, крыс, мышей и др.). В базе данных Tremble нашлось 5 718 мотивов в 5 718 последовательностях. Все белки также являются антигенами, относящимся к главному комплексу гистосовместимости.
Слабый паттерн:
P-K-[TA]-H-V-T-X(0,3)-H(2)-P-X(2)-[ED]-X-[AV]-X(0,1)-T-L-R-C-W-A-L-X-F-Y-P-A-X-I-T-L-T-W-X(3)-G-E-X(0,4)-
-X(3)-Q-D-X-E-L-V-E-T-R-P-A-G-D-X(2)-F-Q-K-Q-A(2)-V(3)-P-X(1,4)-G-X-E-Q-[KR]-Y-T-C-H-V-[QY]-H-X(0,4)-
-
E-G-L-X(2)-P-[LV]-T-X(2)
Для составления слабого паттерна все не очень консервативные позиции были заменены на позицию, подразумевающую любую аминокислоту (Х). Результатом поиска по этому паттерну стали 119 мотивов из 119 последовательностей в Swissprot и 6230 мотивов из 6227 последовательностей в Tremble.
Возможно сильнее ослабить паттерн, если расширить некоторые консервативные позиции. Можно расширить позиции, содержащие алифатические аминокислоты, кислые или основные аминокислоты. Например, позицию, содержащую положительно заряженную аминокислоту аргинин (R), можно расширить добавлением положительно заряженного лизина (K): R → [RK].
Более слабый паттерн:
P-[KR]-[TA]-H-[VILA]-T-X(0,3)-H(2)-P-X(2)-[ED]-X-[AVIL]-X(0,1)-T-[LIVA]-[RK]-C-W-[AILV](2)-X-F-Y-P-[AILV]-
-X-[ILVA]-T-[LIVA]-T-W-X(3)-G-[ED]-X(0,4)-X(3)-[QN]-[DE]-X-[ED]-[LIVA](2)-[ED]-T-[RK]-P-[AILV]-G-[DE]-
-X(2)-F-[QN]-[KR]-[QN]-[AILV](2)-[VAIL](3)-P-X(1,4)-G-X-[ED]-[QN]-[KR]-Y-T-C-H-[VAIL]-[QY]-H-X(0,4)-
-[ED]-G-[LAIV]-X(2)-P-[LVIA]-T-X(2)
По этому паттерну было найдено 125 мотивов в 125 последовательностях в Swissprot и 6362 мотивов в 6359 последовательностях в Tremble.
Аналогично найденным белкам по сильному паттерну, белки, найденные по слабым паттернам, являются антигенами, принадлежищими главному комплексу гистосовместимости.
Дата последнего изменения: 29.05.2014