
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Онлайн BLAST
Программа BLAST (Basic Local Alignment Search Tool) на сайте NCBI служит для поиска последовательностей белков или нуклеиновых кислот, сходных с входной последовательностью. Существует несколько видов этой программы. Одна из которых рассматривалась BLASTP ранее, создана для поиска последовательностей белков гомологичных входной последовательности. Другой вид — BLASTN. И эта программа ищет нуклеиновые последовательности, сходные с входной. Также есть другие виды: BLASTX, TBLASTN, TBLASTX. Программа BLAST реализованна в двух формах: онлайн программа и локальная программа.
Программа BLASTN
В данной работе будет рассмотрена онлайн форма программы BLASTN (работу с локальной программой ищи далее). Данная программа предоставляет следующие возможности:
1. Поиск организма по фрагменту нуклиотидной последовательности
Имея последовательность из неивестного организма, с помощью вышеуказанной программы, можно установить этот организм. Например, был осуществлен поиск с помощью программы BLASTN данной последовательности длинной 300 нк. Поиск проводился со следующими параметрами:
- использование алгоритма megablast (он стоит по умолчанию);
- поиск по банку refseq_genomic;
- поиск по бактериям и археями;
- поиск не по «регионам низкой сложности» (без галочки перед параметром "Low complexity regions") .
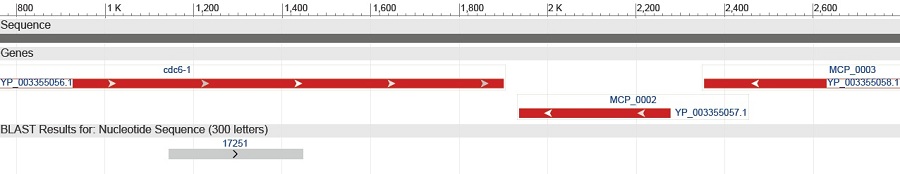
В результате поиска мы установили, что рассматриваемая последовательность взята из генома археи вида Methanocella paludicola (АС записи RefSeq NC_013665.1). А также, что рассматриваемый фрагмент расположен с 1145 по 1444 нуклеотид в геноме археи.
Далее мы определели, что этот фрагмент является частью гена cdc6-1 (см. рис.1), координаты которого — 599-1900. Данный ген кодирует гомолог белка, контролирующий деление клетки.
|
Рис.1. Исследуемый фрагмент и ген cdc6-1 из генома Thermoanaerobacter pseudethanolicus ATCC 33223. Область изображенная на рисунке — от 764 до 2794 нуклеотида. Рисунок получен с помощью геномного браузера на сайте NCBI
2. Поиск гомологов последовательности и поиск некодирующих последовательностей
Программа BLASTN предоставляет возможность найти гомологов входной нуклеотидной последовательности.
Из генома уже много раз рассматриваемой ранее бактерии Thermoanaerobacter pseudethanolicus ATCC 33223 была выбрана последовательность какой-то тРНК, а именно сериновой тРНК, координаты которой 32953..33042. Для выбора тРНК использовался FTP-сервер NCBI, оттуда был скачен файл с раширением .frn, где уже храняться вырезанные тРНК бактерии. Из этого файла по традиции была выбрана первая же последовательность тРНК.
Далее был проведен поиск гомологов данной последовательности по всем бактериям, относящимся к тому же порядку, что и бактерия Thermoanaerobacter pseudethanolicus, а именно к порядку Thermoanaerobacterales.
Поиск был проведиен тремя разными вариантами:
1. с использованием алгоритма megablast: найдено 33 последовательности, из которых все имеют E-value<0.001
2. с использованием алгоритма blastn с параметрами по умолчанию: найдено 69 последовательностей, из которых у 50 E-value<0.001
3. с использованием алгоритма blastn с максимально чувствительными параметрами (длинна слова = 7, match/mismatch = 1/-1):
найдено 80 последовательностей, из которых у 49 E-value<0.001.
Заметим, что, применяя алгоритм megablast, находится меньше гомологичных последовательностей, чем при использовании алгоритма blastn. Из чего можно сделать вывод, что алгоритм megablast является более строгим, чем blastn. При запуске алгоритма blastn с более чувствительными параметрами привело, как и ожидалось, к нахождению большего количества гомологов. Но стоит обратить внимание, что количество находок при использование алгоритма blastn с разными степенями чувствительности с E-value<0.001 сохраняется практически одинаковым, из чего можно сделать вывод, что увеличение чувствительности параметров приводит к увеличению недостоверных последовательностей.
Поиск гомолога белка человека в слоне
Помимо работы с программой BLASTN, была проведена работа по поиску гомологов белка с помощью сайта ENA (это также можно осуществить с помощью программы BLASTP).
Для начала был выбран такой белок человека, для которого идентификатор в Swiss-Prot и моя фамилия начинаются
с максимального количесва одинаковы букв. Для этого была введена следующая команда EMBOSS:
infoseq sw:Х*_human -only -name -desc -out list.txt
на месте Х сначала была подставлена просто буква «k» для того чтобы просто убедиться, что существуют идентификаторы
начинающиеся на эту букву, далее предполагалось пойти по нарастанию количества букв из моей фамилии,
подставляемых вместо Х, но уже поиск с Х=«ko» не дал никах результатов. Поэтому выбор белка производился
среди белков, идентификаторы которых начинаются на «k», а таких белков было найдено 750 штук. Идентификаторы и описание
этих белков сохранены в этом файле. В конечном счете,
был выбран первый же белок из списка (идентификатор KAPCB_HUMAN). Этот белок — бетта субъединица цАМФ-зависимой протеинкиназы.
Далее был получен файл kapcb_human.fasta
с последовательностью выбранного белка , используя команду:
seqret sw:kapcb_human -auto
Затем был проведен непосредственно поиск гомологов выбранного белка в геноме африканского слона. Для это были установлены следующие параметры поиска:
- поиск не отдельных экзонов, а белок полностью (чекбокс "spliced translated nucleotide search")
- поиск по геному африканского слона (в графе "Collection" "Loxodonta_africana") .
В результате поиска было найдено 82 гомолога. Подробнее была рассмотрена лучшая находка:
- E-value — 7E-202;
- длина полученного выравнивания — 351;
- identity полученного выравнивания — 98;
- координаты найденного гена в геноме слона — 6480785..6615981 (обратная цепь);
- количество интронов в данном гене слона — 9 .
Дата последнего изменения: 12.11.2014