
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Восстановление предкового состояния доменной архитектуры
Эволюционные домены – это единицы непрерывной эволюции, то есть эволюции, в процессе которой в аминокислотной последовательности белка от поколения к поколению происходили только незначительные локальные изменения, закрепляемые отбором, такие, как небольшие вставки, делеции или мутации отдельных аминокислотных остатков. Целью данной работы было реконструировать эволюцию доменной архитектуры выбранного домена.
Был выбран домен, встречаемый в доменной архитектуре вместе с доменом ААА_10, который был найден ранее (см. здесь) в белке АТФазы VirB4 из бактерии Thermoanaerobacter pseudethanolicus. Этот домен (AC = PF03135, ID = CagE_TrbE_VirB) относится к семейству CagE, TrbE, VirB и является компонентом секреторной системы IV типа. Данное семейство содержит 1 506 последовательностей, оно представлено в 797 видах и домены семейства входят в 9 различных доменных архитектур (примеры некоторых из них представлены на рис.1). С сайта PFAM было скачано полное выравнивание всех последовательностей домена и открыто в Jalview, покрашено ClustalX By Conservation 10% (скачать проект).
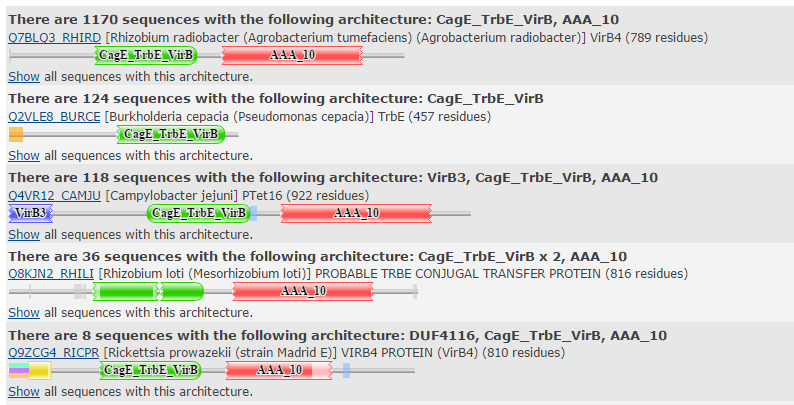
Рис.1. Схемы строения некоторых архитектур, содержащих домен CagE_TrbE_VirB (PF03135). Рисунок получен м помощью сайта PFAM
Далее был получен файл с информацией о доменной структуре каждой последовательности, содержащей выбранный домен (использовался скрипт swisspfam-to-xls.py). Также были получены сведения о таксономии каждой последовательности (использовался скрипт uniprot_to_taxonomy.py). На основе полученных данных была составлена сводная таблица Excel, содержащая список последовательностей с указанием доменной архитектуры, длины домена PF03135 и таксономии.
На ее основе было выбрано 2 доменные архитектуры. Первая из них однодоменная, а вторая является двудоменной, т. е. включает в себя еще один домен: ААА_10 (PF12846), упомянутый раннее, который также участвует в секреторном пути IV типа. Эти архитектуры включают 101 и 592 различную последовательность соответственно.
Также на основе сводной таблицы был выбран тип Proteobacteria из царства Bacteria и два его подтаксона: класс Alphaproteobacteria и класс Gammaproteobacteria. Для каждой архитектуры было выбрано по 12 и 8 представителей каждого подтаксона соответственно (лист «Choisen»). Соответствующие им последовательности были вырезаны из общего выравнивания и открыты в Jalview. С помощью возможностей программы Jalview были удалены небольшие участки с C и N концов, а также столбцы гэпов. Помимо этого, выравнивание было разбито на группы по доменным архитектурам и раскрашено о консервативности внутри групп. Пять последовательностей, принадлежащих первой архитектуре, и четыре– второй, были удалены ввиду их плохого выравнивания с остальными последовательностями. Выравнивание представлено на рисунке 2, проект в формате .jvp можно скачать здесь, выравнивание в формате .fasta можно найти здесь.

|
Рис.2. Выравнивание отобранных последовательностей домена CagE_TrbE_VirB. Выравнивание разбито на группы (выделены черыми рамочками) по доменным архитектурам и раскрашено по консервативности внутри каждой группы. Верхний прямоугольник соответсвует доменной архитектуре 1, включающей в себя домен CagE_TrbE_VirB, нижний - доменной архитектуре 2, в состав которой входят домены ААА и CagE_TrbE_VirB. В названиях последовательностей первая буква А или G соответственно означает пренадлежность подставсону Alphaproteobacteria или Gammaproteobacteria, следующая цифра 1 или 2 указывает на доменную архитектуру 1 или 2 соответственно. Рисунок получен с помощью программы Jalview.
Затем по данному выравниванию в программе MEGA было построено дерево методом Maximum Likelihood (Рис.3).
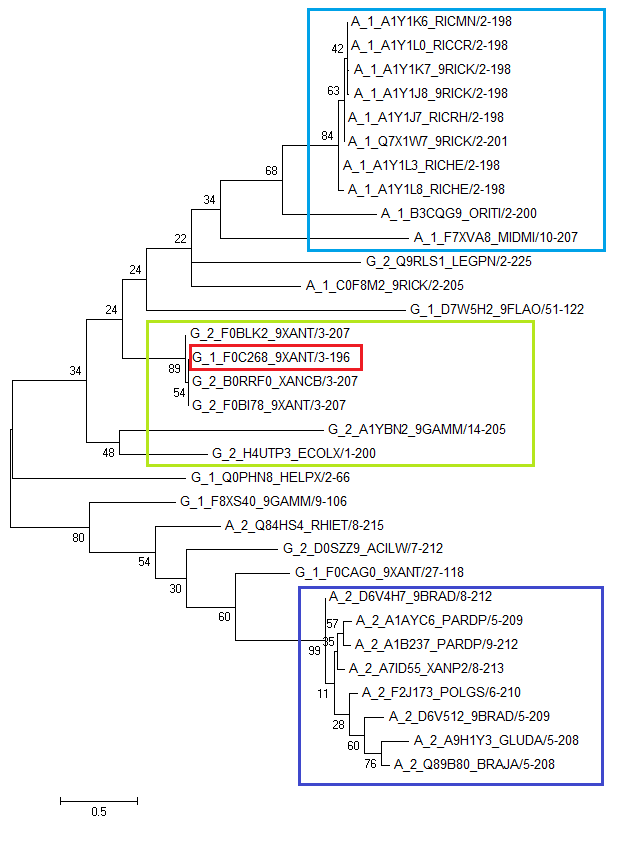
Рис.3. Филогенетическое дерево домена CagE_TrbE_VirB (PF03135). Построено методом Maximum Likelihood с 100 бутстреп репликами. В названиях последовательностей первая буква А или G соответственно означает пренадлежность подставсону Alphaproteobacteria или Gammaproteobacteria, следующая цифра 1 или 2 указывает на доменную архитектуру 1 или 2 соответственно. Синим, голубым и зелеными цветами выделены группы белков, которые можно оособить отдельные клады. Красным цветом выделена последовательность, которая возможно представляет собоой вторую архитектуру.
При анализе построенного древа можно выделить три обособленные клады соответствующие одному из подтаксонов и одной из архитектур. Красной рамочкой выделена последовательность, на которую я обратила внимание и предположила, что она может относиться ко второй архитектуре, что и было подтверждено выравниванием. Сказать оэволюции доменных архитектур по данному дереву невозможно, так как белки первой архитектуры из подтаксона Gammaproteobacteria очень короткие и плохо выровненые, в виду чего они разбросаны по всему дереву.
Дата последнего изменения: 04.09.2015