
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Геномное окружение. База данных STRING
В ходе этой работы было проведено исследование геномного окружения гена, локус которого Teth39_0060, в геноме бактерии Thermoanaerobacter pseudethanolicus (штамма ATCC 33223), и анализ взаимодействия продукта данного гена с другими белками. Напомню, что продукт – это белок АТФазы VirB4, входящий в комплекс секреторного пути IV типа, уже многократно исследовался ранее. В том числе работа по исследованию окружения гена, а также его вхождения в оперон, была уже проведена (см. здесь). В тот раз использовался геномный браузер на сайте NCBI, для этого исследования будет использована база данных STRING. Последняя содержит экспериментально известную и предсказанную информацию о белок-белковых взаимодействиях для более чем 5.2 миллионов белков [1].
Поиск в БД STRING проводился по названию белка – VirB4, и по организму - Thermoanaerobacter pseudethanolicus ATCC 33223. Была найдена пара локусов гена, кодирующего белок VirB4: Teth39_0060 и Teth39_2232. Был выбран первый локус.
При анализе полученной информации было выявлено следующее:
1. На первом уровне близости было обнаружено 5 белков, расположенных рядом с исследуемым. На рисунке 1 представлен граф, узлы которого соответствуют белкам, ребра ярко-зеленого цвета отражают «соседство» каждого белка, ребра синего цвета говорят о совместной встречаемости белков, а салатовые ребра указывают на то, что данное сочетание белков описывалось в литературе. То есть, исходя их данного графа, можно сделать вывод, что каждый из белков, кодируемых в локусах Teth39_0059, Teth39_0060, Teth39_0061, Teth39_0065 и Teth39_0066, находится рядом с друг другом, а белок, кодируемый в локусе Teth39_0052 расположен поблизости только белками из Teth39_0060 и Teth39_0065 локусов. Также есть литературные данные о совместной встречаемости комбинаций белков из локусов Teth39_0052, Teth39_0060, Teth39_0065, а комплекс из белков, расположенный в локусах Teth39_0060, Teth39_0052, Teth39_0065, часто встречается в природе у других организмов.
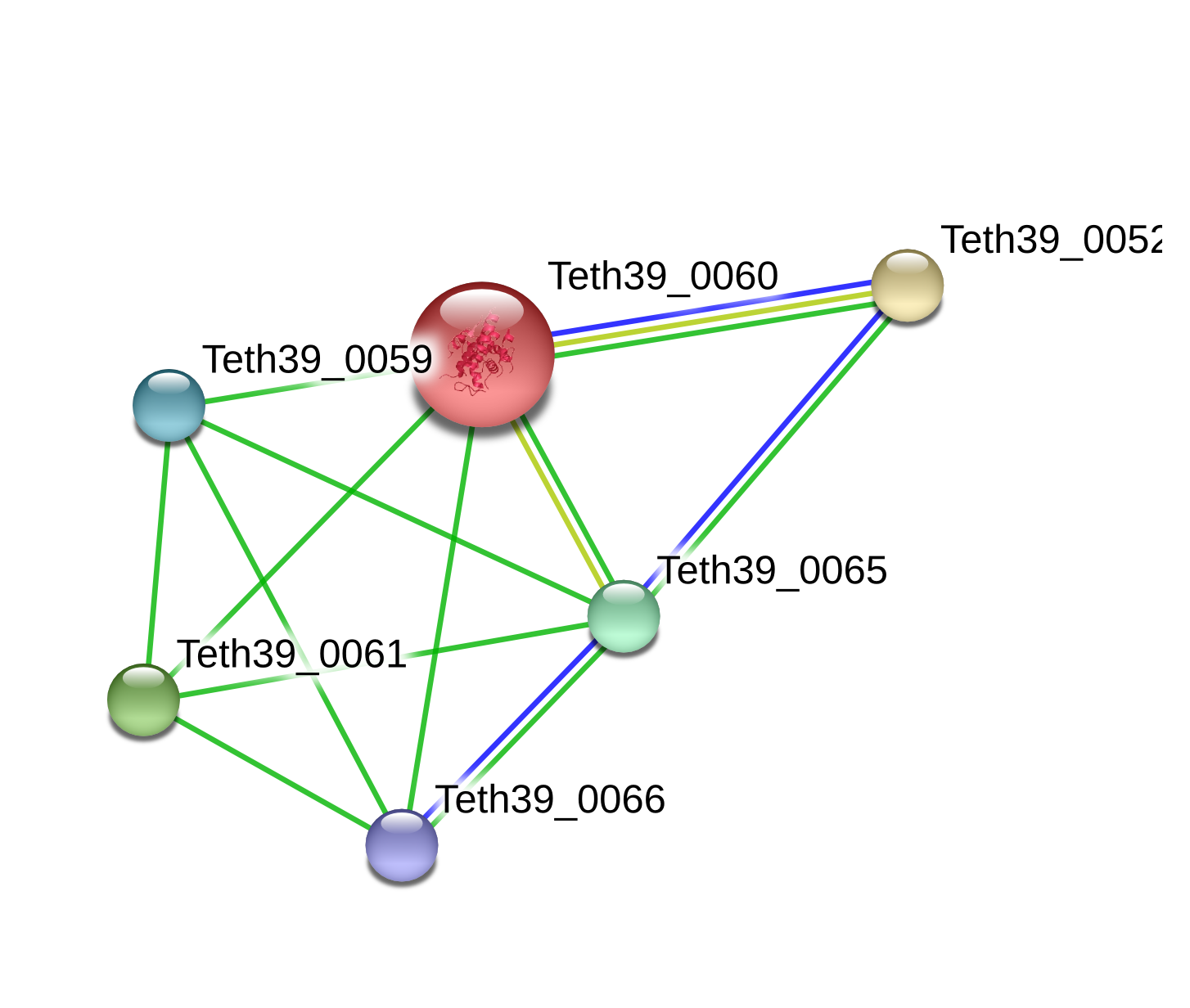
|
Рис.1. Граф Взаимодействия белка АТФазы VirB4 с другими белками. Узлы графа соответствуют белкам: красного цвета – исходный белок АТФазы VirB4 (идентификатор локуса Teth39_0060), желтого цвета – белок из семейства TRAG (Teth39_0052), травянисто-зеленого цвета – белок пептидазы М23В (Teth39_0061), бледно-зеленого цвета – белок Е секреторной системы II типа (Teth39_0065), синего цвета – какой-то гипотетический белок (Teth39_0059), фиолетового – белок секреторной системы II типа (Teth39_0066). Между узлами проходят ребра графов: ярко-зеленого цвета – белки находятся в непосредственной пространственной близости, синего цвета – комбинация белков часто встречается у других организмов, салатового – есть литературное описание комбинации белков. Рисунок получен с помощью БД SRTING.
Сам белок VirB4 имеет отношение к секреторному пути IV типа, есть также литературные данные о принадлежности белка, кодируемого в локусе Teth39_0052 и относящемуся к семейству TRAG, к этому типу секреторного пути [2,3,4]. Этот белок также называют белком VirD4, и есть данные о том, что этот белок совместно с исследуемым являются энергетическими компонентами секреторного пути IV типа [4,5]. Два белка, кодирующиеся в локусах Teth39_0065, Teth39_0066, относятся к секреторному пути II типа, а белок, локус которого Teth39_0061, является пептидазой. Последняя может принимать непосредственной участие в секреции бактерии, так как это фермент расщепляет белковые молекулы, что может способствовать их секреции из клетки. Таким образом, можно подтвердить вывод, сделанный в предыдущей работе, о том, что комплекс из исследуемого белка VirB4 и белков, кодирующиеся в локусах Teth39_0052, Teth39_0059, Teth39_0061, Teth39_0065 и Teth39_0066, выполняет функцию секреции у бактерии Thermoanaerobacter pseudethanolicus.
2. Гены, кодирующие найденные белки, совместно не встречаются в геномах эукариот и архей. Среди бактерий совместное расположение этих генов можно найти в 4 отделах и 2 группах (рис.2):
1) Отдел Протеобактерии. В этом отделе достаточно часто встречается последовательность из трех генов, локусы которых Teth39_0052 (желтый цвет), Teth39_0061 (травянисто-зеленый) и Teth39_0060 (красный). Эти гены соответствуют белкам VirD4, пептидазе и VirB4.
2) Отдел Фирмикуты. Для этого отдела характерно наличие двух генов, кодирующих исследуемый белок VirB4, а также его комбинация с геном белка пептидазы. Причем в геномах некоторых бактерий гены этих белков могут находиться в непосредственной близости друг от друга, то есть на растоянии меньшим, чем 300 нуклеотидов.
3) Отдел Актинобактерии. В геномах бактерий из этого отдела присутствуют только гены 3х белков, а именно белка VirD4, пептидазы и VirB4. Но никакой закономерности в очередности этих генов выявлено не было.
4) Группа BacteroidetesChlorobi. Включает всего 2 бактерии типа Bacteroidetes, которые имеют в своем составе рядом расположенные гены, кодирующие найденные белки, а именно белка VirD4 и белка VirB4. Причем эти гены располагаются на разных цепях.
5) Группа ChlamydiaeVerrucomicrobia. Из данной группы только в геноме одной бактерии Protochlamydia amoebophila (тип Chlamydiae) есть совместно расположенные гены белков VirB4 и VirD4. Но все же эти гены располагаются далеко друг от друга.
6) Отдел Chloroflexi. Также, как и в предыдущем случае, в этом отделе всего одна бактерия, в геноме которой содержаться совместная комбинация генов, найденных белков. Это бактерия Roseiflexus castenholzii, и в ее геноме есть следующая последовательность из генов: дважды ген, кодирующий белок Е секреторной системы II типа (Teth39_0065), ген белка пептидазы и ген белка VirB4.
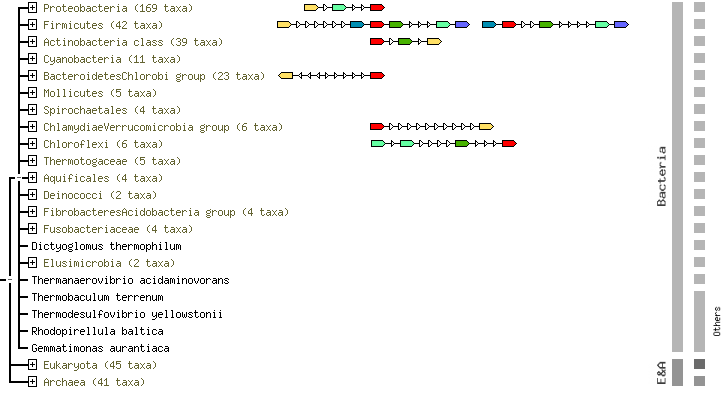
|
Рис.2. Геномное окружение белка VirB4. Дерево организмов с точностью до первого филума. Полностью раскрытое дерево см. здесь. Прямоугольники различного цвета обозначают гены. Гены красного цвета кодируют белок VirB4 (идентификатор локуса Teth39_0060), желтого цвета – белок из семейства TRAG (Teth39_0052), травянисто-зеленого цвета – белок пептидазы М23В (Teth39_0061), бледно-зеленого цвета – белок Е секреторной системы II типа (Teth39_0065), синего цвета – какой-то гипотетический белок (Teth39_0059), фиолетового – белок секреторной системы II типа (Teth39_0066). Треугольник прозрачного цвета также кодирующие гены, но не рассматриваемых белков, не имеющие высокое значение Score. Ориентация прямоугольников соответствует направлению цепи. Черная линия между генами указывает на непосредственное соседство генов, то есть их расположение не далее 300 нуклеотидов друг от друга. Рисунок получен с помощью БД SRTING.
3. Совместная встречаемость белков представлена на рисунке 3. Степень интенсивности цвета квадратика отражает гомологичность последовательности белка: черный цвет – последовательности белков 100% гомологичны. А степень «полноты» фигуры обозначает встречаемость данного белка в представленном таксоне. Например, у не у всех бактерий из отдела Фирмикут, к которому относится бактерия Thermoanaerobacter pseudethanolicus, присутствуют найденный белки, о чем говорят квадратики со скошеными верхними левыми углами. Так же можно сказать, что явной совместной встречаемости этих белков в других таксон не наблюдается.
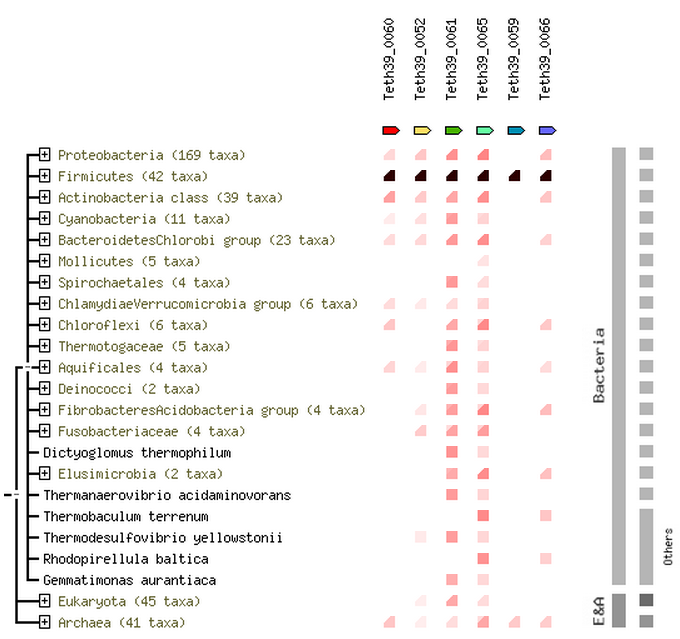
|
Рис.3. Совместная встречаемость белков. Совместная встречаемость белков. Интенсивность цвета квадратика отражает гомологичность последовательности белка, степень «полноты» фигуры обозначает встречаемость данного белка в представленном таксоне. Рисунок получен с помощью БД SRTING.
Источники:
[1] http://en.wikipedia.org/wiki/STRING
[2] Genomes-based phylogeny of the genus Xanthomonas. Rodriguez-R LM, Grajales A, Arrieta-Ortiz ML, Salazar C, Restrepo S, Bernal A BMC Microbiol. 12:43 (2012). PubMed
[3] Proteome of the phytopathogen Xanthomonas citri subsp. citri: a global expression profile. Soares MR, Facincani AP, Ferreira RM, Moreira LM, de Oliveira JC, Ferro JA, Ferro MI, Meneghini R, Gozzo FC Proteome Sci. 8:55 (2010). PubMed
[4] Conjugative Type 4 secretion system of a novel large plasmid from the chemoautotroph Tetrathiobacter kashmirensis and construction of shuttle vectors for Alcaligenaceae. Dam B, Ghosh W, Das Gupta SK Appl Environ Microbiol. 75(13):4362-73 (2009). PubMed
[5] http://www.genome.jp/kegg-bin/show_pathway?tpd03070+Teth39_0052
Дата последнего изменения: 27.03.15