Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Структурная классификация. Сравнение доменов SCOP/SCOPe, ECOD, CATH и Pfam
Пуриновый репрессор (PDB ID: 1wet) является главным регуляторным белком в пути биосинтеза пуриновых нуклеотидов у E.coli. В активном состоянии этот белок является димером и взаимодействует с ДНК. Но далее будет рассматриваться только мономер белка. Согласно четырем различным базам данных по классификациям (SCOPe, ECOD, CATH и Pfam) были найдены границы доменов этого белка:
1) Классификация SCOPe
Для выбранного мономера белка БД SCOPe (Structural Classification of Proteins — extended) нашла 2 домена.
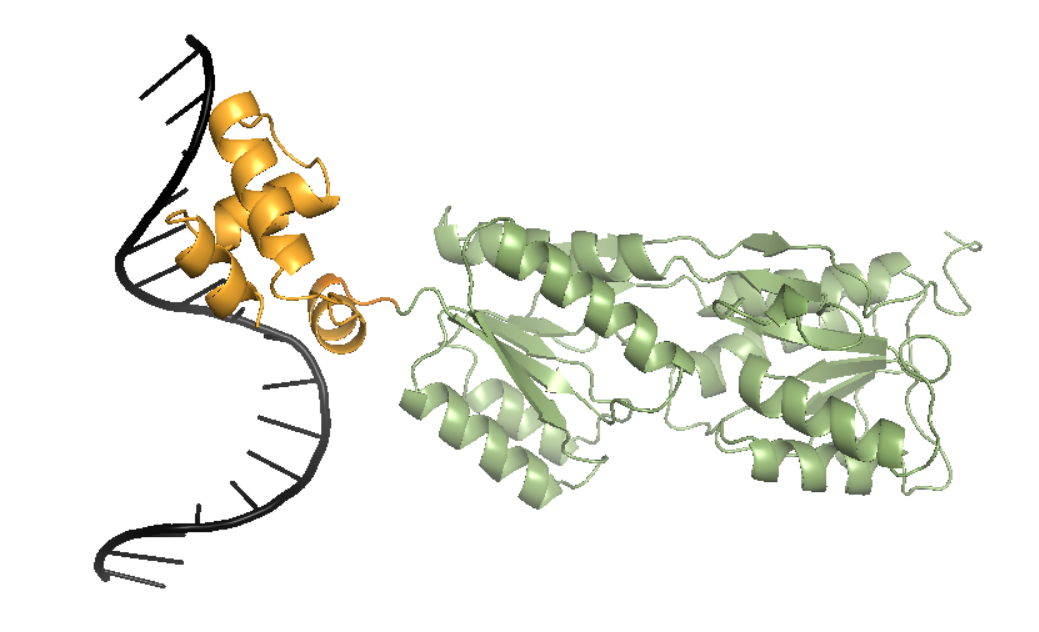
Первый N-терминальный домен (d1weta1) расположен с 3 по 58 ао, он отвечает за взаимодействие с ДНК (рис.1 оранжевый домен).
Второй домен (d1weta2) — С-терминальный и располагается с 59 по 340 ао (рис.1 зеленый домен).
|
|
Рис.1. Два домена пуринового репрессора, определенные БД SCOPe. Домен d1weta1 расположен с 3 по 58 ао, выделен оранжевым цветом. Домен d1weta2 расположен с 59 по 340 ао, выделен зеленым цветом. Черным цветом обозначена молекула ДНК и ао, не входящие в какой-либо домен. Изображение получено с помощью программы pymol. |
2) Классификация ECOD
С помощью этой БД в пуриновои репрессоре было выделено
3 домена.
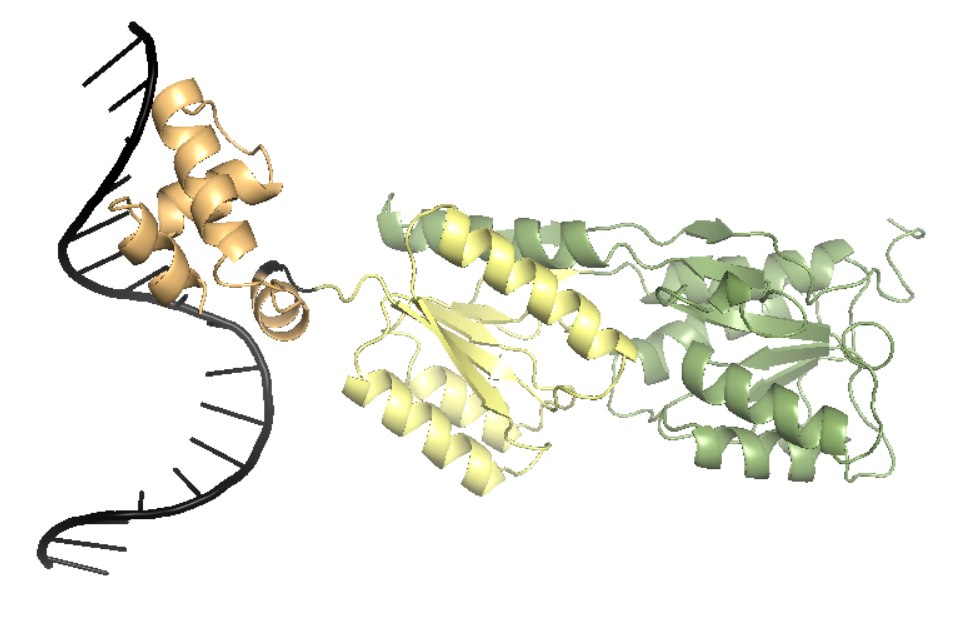
Первый домен (e1wetA4) практически соответствует N-терминальному доменому, классифированному SCOPe (d1weta1). Он располагается с 3 по 55 ао, то есть он не включает 56-58 ао. Эти три остатка располагаются в фрагменте,
который не имеет вторичной структуры и каких-либо других особенностей, поэтому эти различия можно считать
не существенными и будем утверждать, что этот домен SCOPe и ECOD (Evolutionary Classification of Protein Domains) классифицировали одинаково (рис.2 оранжевый домен).
ECOD также предлагает эволюционную классификацию домена на пяти уровнях: архитектура (А), возможный уровень гомологии (Х), уровень гомологии (Н), топология (T), семейство (F).
e1wetA4 домен обладает следующей эволюцинной классификацией:
А: alpha arrays → альфа спирали, в основном расположенные перпендикулярно
X: HTH → мотив спираль-поворот-спираль
H: HTH
T: tetra-helical, lambda repressor-like → четыре спирали по структуре сходные с лябда репрессором
F: LacI → семейство бактериальных регуляторных белков LacI
Следующие два домена, выделенные с помощью ECOD, e1wetA5 (рис.2 желтый домен) и e1wet3 (рис.2 зеленый домен) располагаются с 58 по 159 ао и с 160 по 340 ао соответственно.
Получается, что один домен d1weta2, выделенный с помощью SCOPe, ECOD разделяет на два. Эти два домена имеют одинаковую эволюционную иерархию вплоть до топологии, различаются только семействами:
A: a/b three-layered sandwiches → α/β сэнгвич, приемущественно паралельный β-лист располагается между двух слоев из альфа-спиралей
X: Other Rossmann-like structures with the crossover → структуры похожие на укладку росмана с кроссовером
H: Periplasmic binding protein-like I → сходный с белком связывающимся в периплазме
T: Periplasmic binding protein-like I
F для e1wetA5: EUF05661
F для e1wetА3: EUF05654
|
|
Рис.2. Три домена пуринового репрессора, определенные БД ECOD. Домен e1wetA4 (3-55 ао) покрашен оранжевым цветом, домен e1wetA5 (58-159 ао) — желтым, e1wet3 (160-340) — зеленым. Черным цветом обозначена молекула ДНК и ао, не входящие в какой-либо домен. Изображение получено с помощью программы pymol. |
3) Классификация CATH
БД CATH также определяет наличие 3 доменов
в пуриновом репрессоре (рис.3):
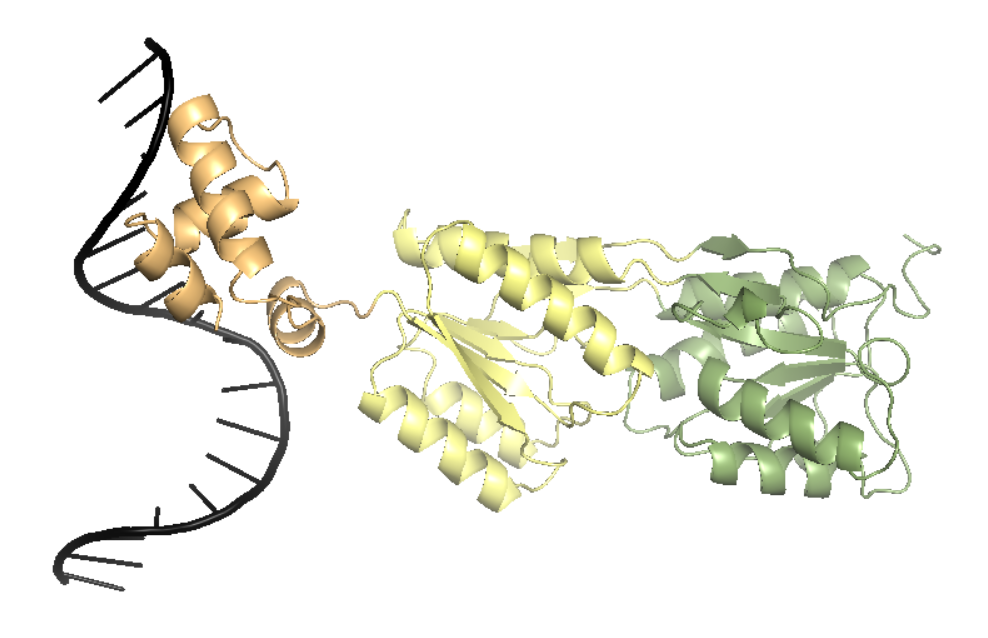
1wetA01 (3-59), 1wetA02 (60-161, 292-323) и 1wetA03 (162-291, 324-340). Первый домен можно считать таким же,
как и N-терминальный SCOPe домен и ECOD домен e1wetA4. А другие два домена отличаются, от e1wetA5 и e1wetA3 доменов,
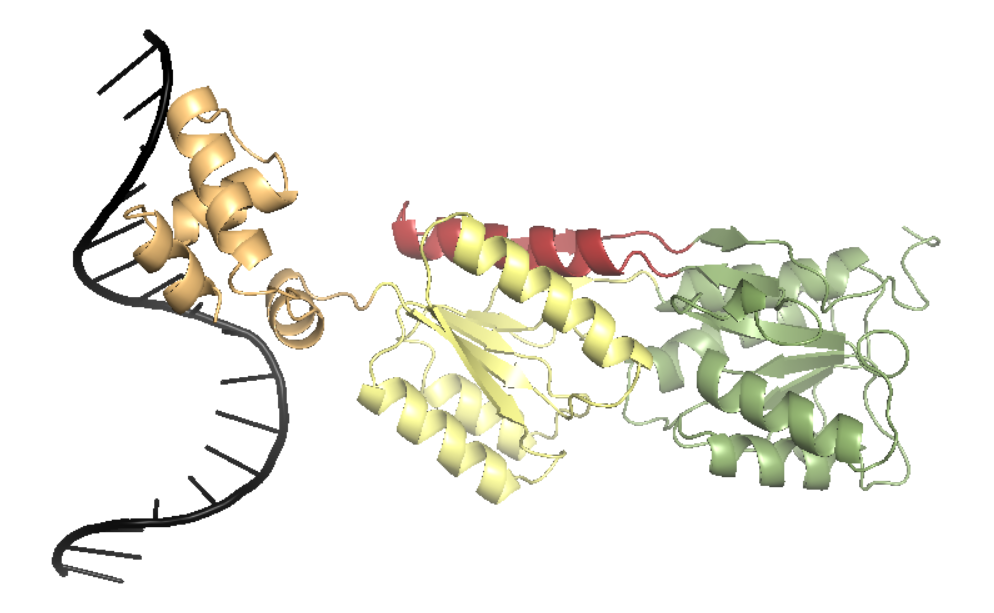
определенных ECOD. Различия заключатся всего лишь в отнесении одной α-спирали (выделена красным цветом на рис.5)
к среднему или С-терминальному домену. CATН также имеет свою классификацию доменов.
C (class): Mainly Alpha
A (Architecture): Orthogonal Bundle
T (Topology): 434 Repressor (Amino-terminal Domain)
H (Homologous Superfamily): lambda repressor-like DNA-binding domains (CATH code 1.10.260.40)
Superfamily: lambda repressor-like DNA-binding domains
Functional family: Transcriptional regulator, LacI family
C: Alpha Beta
A: 3-Layer(aba) Sandwich
T: Rossmann fold
H: CATH code 3.40.50.2300
Superfamily: CATH code 3.40.50.2300
Functional family: не определено для 1wetA02, а для 1wetA03 Transcriptional regulator, LacI family
По классификации домены, определенные ECOD и CATH, очень похожи. Различие состоит в том, что CATH средний и С-концевой домены относит по топологии к росмановской укладке. А ECOD относит к топологии, которая похожа на россмановсую укладку, а не непосредственно к ней. Получение таких доменов, как предлагает CATH возможно, если в ходе эволюции белка происходит перестановка мотивов [1]. То есть, если определенная структура (вторичный элемент), в нашем случае α-спираль, сохраняет свое пространственное расположение, но при этом в последовательности ао меняет свое расположение.
|
|
Рис.3. Три домена пуринового репрессора, определенные БД ECOD. Домен 1wetA01 (3-59) покрашен оранжевым цветом, 1wetA02 (60-161, 292-323) — желтым и 1wetA03 (162-291, 324-340) — зеленым. Черным цветом обозначена молекула ДНК и ао, не входящие в какой-либо домен. Изображение получено с помощью программы pymol. |
Рис.4. Три домена пуринового репрессора, определенные БД ECOD. Домен 1wetA01 (3-59) покрашен оранжевым цветом, 1wetA02 (60-161, 292-323) — желтым и 1wetA03 (162-291, 324-340) — зеленым, без α-спирали, которая отличает CATH C-терминальный домен от ECOD C-терминального домена, покрашена красным цветом. Черным цветом обозначена молекула ДНК и ао, не входящие в какой-либо домен. Изображение получено с помощью программы pymol. |
4) Классификация Pfam
Pfam классифицировал 2 домена в данном пуриновом репрессоре.
Первый домен lacI располагается с 3 по 48 ао (рис.5 оранжевый домен). Этот домен относится к
семейству lacI, и отвечает за связывание с ДНК через
HTH мотив (мотив спираль-поворот-спираль).
Второй домен Peripla_BP_3 располагается на промежутке 170-331 ао (рис.5 зеленый домен). Такой домен характерен для различных
транскриптиционных факторов, его находят в различных переплазма-связывающих белках, но при этом маловероятно нахождение самого
домена в периплазме. Вероятно этот домен связывает
маленькую молекулу лиганда, за которую отвечает ДНК-связывающий домен.
В нашем случае этот домен связывает пуриновое основание — гуанин (рис.5 красный цвет).
Получается, если в E.coli присутствует достаточное или избыточное количество пуриновых оснований,
то пуриновое основание связывается с доменом Peripla_BP_3, в результате чего пуриновый репрессор активируется
и становится способным связаться с ДНК, а это связывание в свою очередь приводит к ингибированию синтеза пуриновых оснований.
Домен lacI отличается от всех ранее найденных N-терминальных доменов, он не включает в себя четвертую α-спираль (рис.5 серый цвет), а состоит только из трех α-спиралей. Домен Peripla_BP_3 также отличается от ранее найденных CATH и ECOD С-терминальных доменов, Peripla_BP_3 не содержить часть одной α-спирали (рис.5 ярко-зеленый цвет). Я думаю, что это несколько неверно и следует включать эту часть α-спирали в С-терминальный домен.
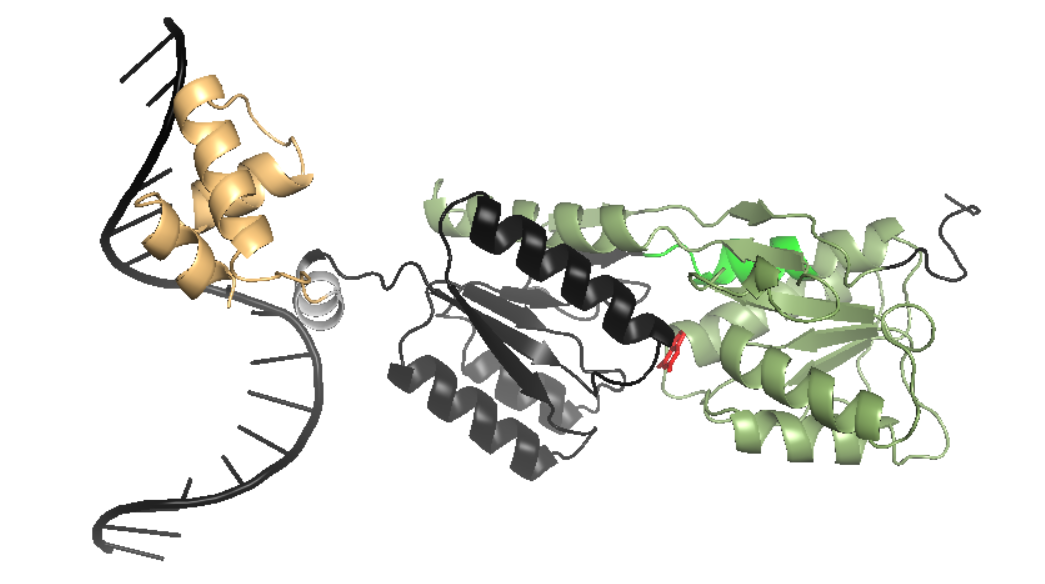
|
|
Рис.5. Два домена пуринового репрессора, определенные БД Pfam. Домен lacI (3-48) покрашен оранжевым цветом, домен Peripla_BP_3 (170-331) — зеленым цветом, красным цветом выделена молекула гуанина. Серым цветом и ярко-зеленым цветом выделены участки, в которых наблюдается отличие доменов, определенных ранее. Черным цветом обозначена молекула ДНК и ао, не входящие в какой-либо домен. Изображение получено с помощью программы pymol. |
Ниже представлена сводная таблица по всем классификациям (табл.1).
Таблица 1. Сводная таблица классификаций доменов для бактериального пуринового репрессора (PDB ID: 1wet)
| База данных структурных классификаций | Количество доменов | Название доменов | Расположение, ао | Краткая характеристика |
| SCOPe | 2 | d1weta1 | 3-58 | N-терминальный домен, отвечает за взаимодействие с ДНК |
| d1weta2 | 59-340 | C-терминальный домен | ||
| ECOD | 3 | e1wetA4 | 3-55 | имеет четыре спирали, по структуре сходные с лябда репрессором, относится к семейству бактериальных регуляторных белков LacI |
| e1wetA5 | 58-159 | обладает α/β-сэнгвич архитектурой, по топологии структура похожа на укладку Росмана |
||
| e1wetA3 | 160-340 | обладает α/β-сэнгвич архитектурой, по топологии структура похожа на укладку Росмана |
||
| CATH | 3 | 1wetA01 | 3-59 | состоит из ортогональных спиралей, по структуре сходные с лябда репрессором, является транскриптиционным фактором, относится к семейству бактериальных регуляторных белков LacI |
| 1wetA02 | 60-161, 292-323 | обладает α/β-сэнгвич архитектурой, по топологии структура является укладкой Росмана |
||
| 1wetA03 | 162-291, 324-340 | обладает α/β-сэнгвич архитектурой, по топологии структура является укладкой Росмана |
||
| Pfam | 2 | lacI | 3-48 | бактериальный регуляторный белок, относится к семейству LacI, отвечает за связывание с ДНК |
| Peripla_BP_3 | 170-331 | характерен для транскриптиционных факторов, связывает маленькую молекулу лиганда, за которую отвечает ДНК-связывающий домен (в данном случае молекулу пуринового основания) |
В результате мы получили, что четыре различных базы данных по структурным классификациям белков дают различный результат в определении границ доменов для бактериального пуринового репрессора (PDB ID: 1wet). Некоторые возможные причины этого обсуждались выше. В общем случае это непосредственно зависит от алгоритма, с помощью которого домены определяются.
На основании проделанной работы, я предположила бы существование трех доменов в исследуемом белке. Первый — располагается на N-конце, отвечает за связывание транскриптиционного фактора с молекулой ДНК и включает 3 или 4 α-спирали. Второй домен — является C-терминальным, связывает молекулу пуринового основания и обладает укладкой Росмана. И последний домен — располагается по середине между первыми двумя доменами и тоже содержит структуру, похожую на укладку Росмана.
[1] Gorbalenya, Alexander E., et al. "The palm subdomain-based active site is internally permuted in viral RNA-dependent RNA polymerases of an ancient lineage." Journal of molecular biology 324.1 (2002): 47-62.
PMID: 12421558
Дата последнего изменения: 18.12.16