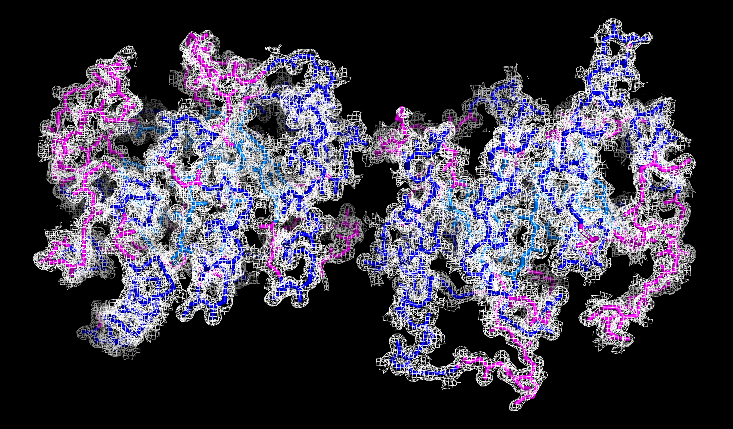
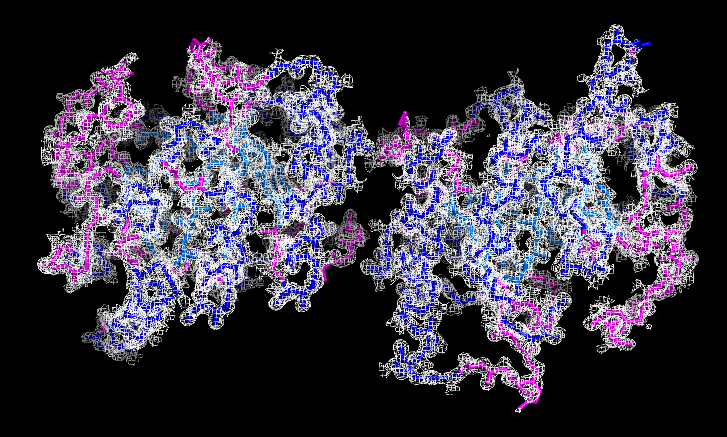
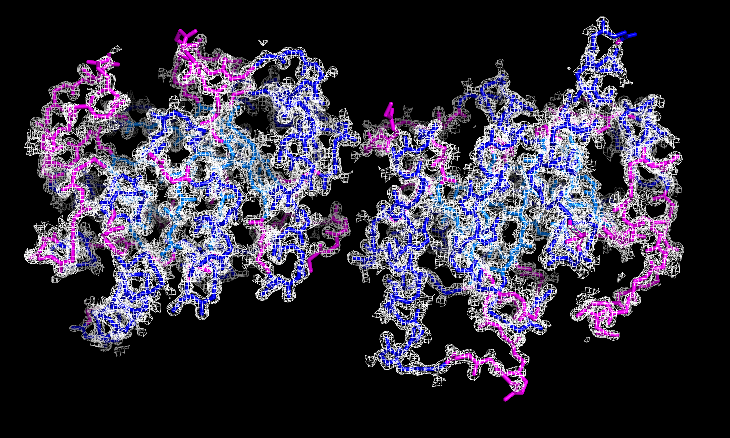
Структура белка тимин-фосфат-синтазы Bacillus subtilis (PDB ID 1G6C, разрешение – 1.40 Å) была выбрана для дальнейшей работы. Было проверено, что структура удовлетворяет следующим требованиям:
Некоторые из найденных гомологов
| Название | RMSD | Nalgn | PDB ID |
| Thiamine phosphate pyrophosphorylase from Pyrococcus furiosus Pfu-1255191-001 | 1.39 | 202 | 1XI3 |
| Crystal Structure of Thiamin Phosphate Synthase from Mycobacterium tuberculosis | 1.69 | 191 | 3O63 |
| Structure of TenI from Bacillus subtilis | 1.88 | 184 | 1YAD |
| Crystal structure of TenI from Bacillus subtilis complexed with product cThz-P | 1.97 | 186 | 3QH2 |
| Mechanism of the Class I KDPG aldolase | 2.16 | 182 | 1WA3 |
Модель структуры, файл mmCIF (macromolecular Crystallographic Information File) 1G6C и файл структурных факторов были загружены из PDB. Каждый структурный фактор расположен в отдельной строке, представляющей собой последовательность чисел, при этом прямого соответствия атомам модели нет. Число атомов в модели можно определить по файлам .pdb или .cif (mmCIF), число отражений (reflections) – по файлу структурных факторов. Поля, приведённые в файлах структурных факторов, могут отличаться от файла к файлу. В файле 1g6c-sf.cif присутствуют следующие данные для каждого отражения (в том числе h, k и l – индексы рефлекса):
| Crystal_ID | Wavelength_ID | Scale_group_code | Index_h | Index_k | Index_l | Status | F_means_au | F_means_sigma_au |
| 1 | 1 | 1 | 0 | 0 | 28 | o | 99.420 | 1.481 |
| 1 | 1 | 1 | 0 | 0 | 32 | o | 164.886 | 8.885 |
| 1 | 1 | 1 | 0 | 0 | 40 | o | 483.905 | 35.549 |
Файл с картой электронной плотности для 1G6C был загружен с сайта EDS.
Для визуализации структуры и электронной плотности была использована программа для визуализации молекулярных данных PyMol.
 |
 |
 |
| Level=1σ, carve=2Å | Level=1.5σ, carve=2Å | Level=2σ, carve=2Å |
Изображения электронной плотности вокруг полипептидной цепи (для уровней изолиний 1.0 σ, 1.5 σ и 2.0 σ на указанных расстояниях от выбранного множества – параметр carve) были получены, в частности, с помощью команд:
| isomesh backbone_map, 1g6c_map, 1, backbone, carve=2 isomesh backbone_map, 1g6c_map, 1.5, backbone, carve=2 isomesh backbone_map, 1g6c_map, 2, backbone, carve=2 |
При уровне подрезки 2σ для некоторых участков молекулы уже пропадает электронная плотность.
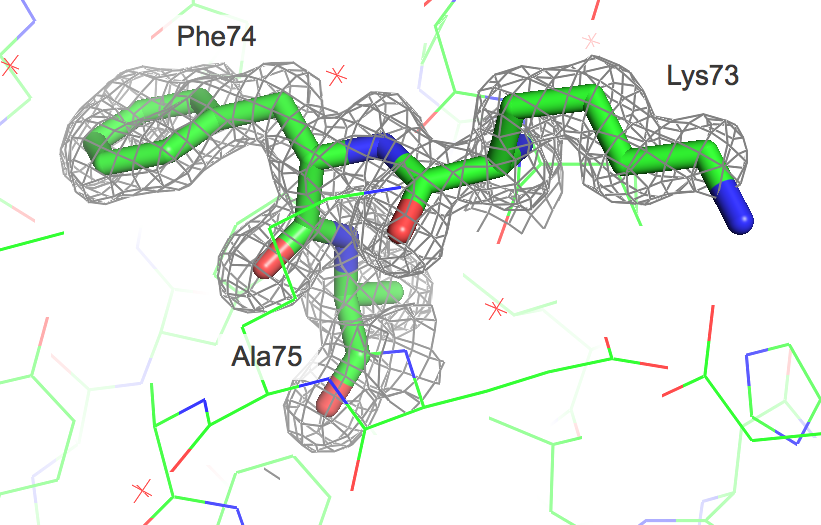
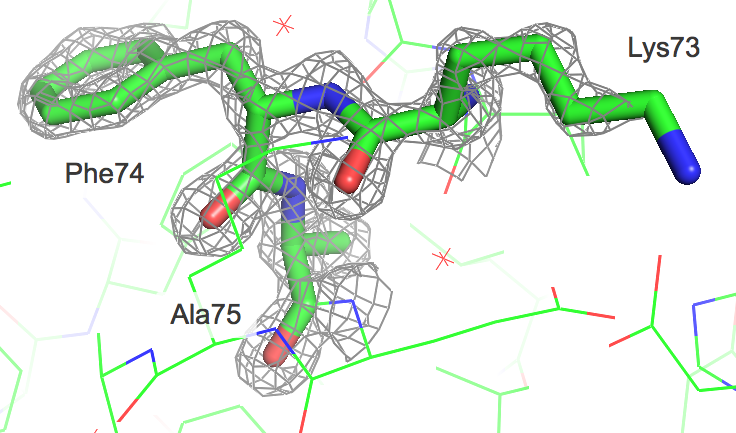
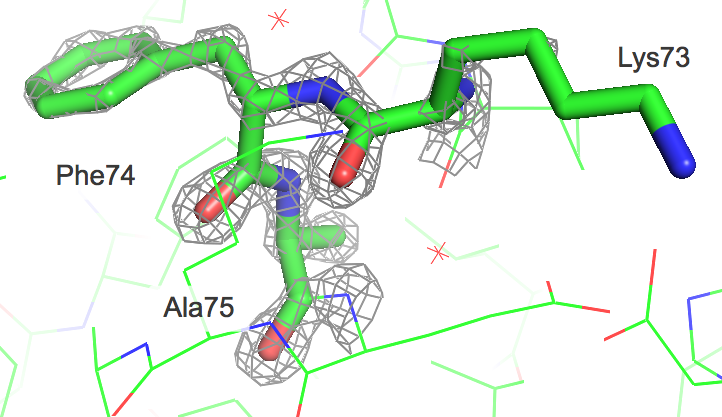
Электронная плотность вокруг трех аминокислотных остатков лизина (Lys73); фенилаланина (Phe74) и аланина (Ala75) для различных уровней подрезки электронной плотности (1.0 σ, 2.0 σ и 3.0 σ) показана на изображениях ниже. По сгущениям электронной плотности видно положение боковых групп аминокислот, однако наблюдаются и случаи несоответствия, например, по сгущению электронной плотности нельзя определить положение атомов радикала лизина Lys73.
 |
 |
 |
| Level=1σ, carve=2Å | Level=2σ, carve=2Å | Level=3σ, carve=2Å |
| Электронная плотность для участка структуры Lys73 + Phe74 + Ala75 |
Фрагмент кода, с помощью которого были получены изображения электронной плотности вокруг аминокислотных остатков:
| select lys_phe_ala, resi 73-75 show sticks, lys_phe_ala center lys_phe_ala zoom lys_phe_ala isomesh map, 1g6c_map, 1.0, lys_phe_ala, carve=2 isomesh map, 1g6c_map, 2.0, lys_phe_ala, carve=2 isomesh map, 1g6c_map, 3.0, lys_phe_ala, carve=2 |
На основании полученных данных можно сделать вывод, что разрешение структуры 1G6C не позволяет оценивать отдельные атомы структуры, однако в большинстве случаев есть возможность проследить движение полипептидной цепи, хотя на "граничных" амикислотных остатках наблюдается ухудшение точности визуализации.