BLAST
Определение последовательности
Для поиска был использован обычный blastn (так как про последовательность ничего не известно) в Nucleotide collection.
После приведенных выше данных можно утверждать, что последовательность представляет собой первую субъединицу митохондриальной цитохром-оксидазы, четвертого - и последнего - белка цепи переноса электронов, и с большой долей уверенности предположить, что она принадлежит Polycirrus medusa - виду из класса полихет (неожиданно! почему медуза?), который обитает в северных морях. Подозрительно... неужели его привезли с беломорской биологической станции?
В принципе все последовательности достаточно похожи, но так как этот белок должен быть очень сильно консервативен - он участвует в клеточном дыхании! - количество накопившихся snp между первыми четырьмя образцами (все принадлежат интересующему нас организму) и остальными шестью (разные представители того же рода) намекаeт на то, что все-таки это белок из вышеупомянутой Polycirrus medusa.
Сравнение разных вариантов blast
Из-за высокой консервативности последовательности из первого задания сложно было добиться сильно различающихся результатов. Если даже ограничиться семейством Terebillidae без нужного рода, то blastn выдает очень много результатов вне зависимости от настроек. Megablast можно ограничить до любой степени, включая полное отсутствие результатов, но хотелось все-таки сравнить способности обычного алгоритма. В результате поиск был проведен по роду за исключением организма Polycirrus medusa.
| Алгоритм | Megablast | Blastn | Blastn |
|---|---|---|---|
| Параметры | По умолчанию | Word size: 15 Match/Mismatch Scores: 4,-5 |
|
| Область поиска | Polycirrus кроме Polycirrus medusa | ||
| Количество находок | 16 | 51 | 44 |
| Различия | Совпадают (за исключением одной элементарной транспозиции) с началом blastn. Polycirrus sp. 11BIOAK-1243 и Polycirrus sp. 11BIOAK-1496. | Семь дополнительных находок, все 28S ribosomal RNA gene, partial sequence. | Транспозиция аналогичная Megablast. Еще две между Polycirrus eximius voucher HUNTSPOL0084 и Polycirrus sp. BOLD:AAY2881 voucher FTP_0553,
Polycirrus sp. CMC06 voucher BIOUG |
Как и ожидалось, обычный blastn находит больше всего, с подкрученными параметрами - чуть поменьше, а megablast выделяет самые похожие последовательности.
С вирусом вышла обратная ситуация. Область поиска ограничивать не было смысла. Megablast на настройках по умолчанию выдавал ноль совпадений, если не считать исходную последовательность вируса.
| Алгоритм | Megablast | Blastn | Blastn |
|---|---|---|---|
| Параметры | Word size: 20 | По умолчанию | Word size: 15 Match/Mismatch Scores: 4,-5 |
| Количество находок | 5 | 64 | 22 |
| Различия | Первые два результата совпадают с таковыми в blastn. Следующие обнаруживаются в самом конце списка blastn, а последний - в середине. | Очень. Много. Всего. Из интересного: участки хромосом колей, предсказанные белки Aedes albopictus Apis mellifera и Bombus impatiens, скэффолды и даже полные геномы других видов | Одиннадцать (!) уникальных результатов: восемь предсказанных белков CPEB3 Gopherus evgoodei, мРНК Apis cerana, участок хромосомы Crenobacter cavernae и мРНК гипотетического белка Branchiostoma floridae. |
Что происходит в табличке выше? Почему накрученный blastn выдает результаты, которых нет в обычном? Неясно.
Поиск гомологов
Все используемые последовательности были скачаны ручками из базы данных Uniprot после поиска соответствующих белков в подходящих (как показалось автору - в таксоне Fungi) организмах и принадлежат Saccharomyces cerevisiae. Blast был запущен следующими командами:
makeblastdb -in X5.fasta -dbtype nucl
tblastn -query P36604.fasta -db X5.fasta > bip.out
| Белок | Функция | Лучшая находка | Результат |
|---|---|---|---|
| Binding immunoglobulin protein (BiP) | Белок теплового шока, шаперон, отвечающий за связывание только что синтезированных белков на ЭПР и поддержание их в нужном для фолдинга состоянии, а также транспорт некоторых белков через мембрану ЭПР. | Scaffold-96 Score: 790 E-value: 0.0 Покрытие: 0.95 |
Последовательности гомологичны. Только один длинный (около десяти нуклеотидов) гэп. Есть еще две находки с таким же E-value, причем одна из них стоит всего лишь на три очка меньше. |
| Histone H1 | Линкерный гистон, который связывается со внешней стороной нуклеосомы, фиксируя на ней ДНК. | Scaffold-423 Score: 38.1 E-value: 0.003 Покрытие: 0.17 |
Гомологичных последовательностей в геноме не обнаружено. Это чрезвычайно удивительно. Действительно, зачем паразиту гистоны? |
| DNA topoisomerase 2 | Фермент, релаксирующий сверхспирализованные молекулы ДНК, осуществляя АТФ-зависимое расщепление обеих цепей с последующим переносом их через разрыв и лигированием. | Scaffold-693 Score: 1133 E-value: 0.0 Покрытие: 0.81 |
Последовательности гомологичны. Длинные участки без гэпов, высокий вес. Есть еще одна похожая находка (разница в четыре очка). |
Не знаю, зачем здесь нужны полные результаты blast, но пусть будут:
Поиск генов
Оказалось, что у вороны из предыдущего практикума еще не аннотированы гены. Для поиска был взят тот же скэффолд, что предлагался к скачиванию на вышеупомянутой страничке. Однако когда через полчаса чуда не произошло, было решено использовать что-то покороче. Был найден самый маленький скэффолд той же многострадальной вороны и пропущен через blastx.
Почему? Почему эта ворона оказалась больна? Разумеется, ниже нашлись и приличные белки, но рядом с подавляющим большинством из них красовалось PREDICTED, поэтому было решено повторить blastx по другой базе данных - чтобы предсказывать не по другим предсказаниям, а по проверенным фактам.
Объектом интереса является самый первый результат в выдаче. PAR-3 - рецептор, активируемый протеазой, белок из класса трансмембранных рецепторов, сопряженных с G-белками. Он является кофактором и посредником гидролиза фосфоинозитида, вызываемого тромбином. К сожалению, выравнивание выглядит не слишком привлекательно.
Оно затрагивает только вторую половину протеина, и туда не попадает сайт расщепления тромбином. Но что если повторить алгоритм на сервере? Действия выполнялись аналогично третьему заданию, файл с последовательностью человеческого белка был скачан из результатов blastx.
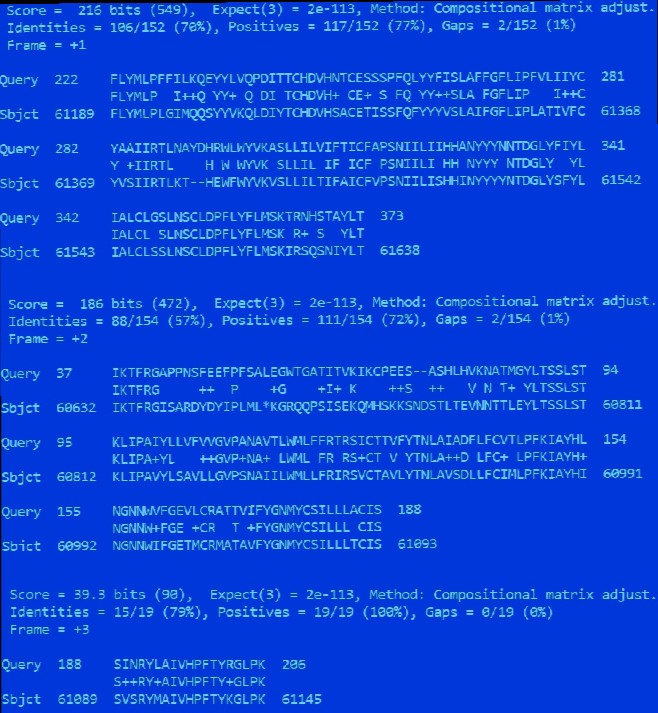
И вторая часть белка волшебным образом материализуется из воздуха! Вместе с третьей! Сайт расщепления (38-39 остатки) на месте, и вообще три выравнивания почти полностью покрывают протеин, кроме участка 207-221 (15 остатков), чье исчезновение не до конца понятно, и 3'-конца. Можно выдвинуть гипотезу, что первые аминокислотные остатки в этом белке намного менее консервативны, так как отрезаются при активации рецептора тромбином, и фактически не участвуют в его дальнейшей работе. Таким образом, мутация в этом участке - если она конечно не нонсенс - не повлияет на функцию кофермента. Три этих участка разделены... интронами? Если приглядеться, можно заметить, что границы выравнивания в скэффолде выглядят немного подозрительно, но когда это предложение было написано, было слишком поздно, чтобы пытаться разгадать еще одну загадку.