Домены и профили
Pfam

Таких последовательностей в базе данных всего 132853. Представить сложно, когда столько отсеквенировали...
| AC | ID | название | число бактериальных последовательностей |
|---|---|---|---|
| PF00270 | DEAD | DEAD/DEAH box helicase | 65256 |
| PF01966 | HD | HD domain | 43279 |
Поиск в Uniprot был произведен следующим образом: taxonomy: "Bacteria [2]" database:(type:pfam pf00270) database:(type:pfam pf01966).
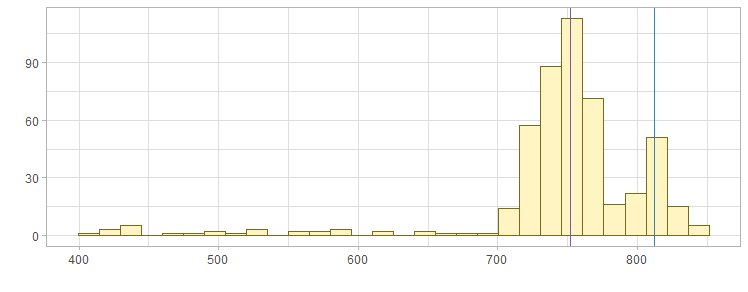
На графике фиолетовая линия обозначает медиану (752), голубая - моду (812).
Разочаровывающе длинную таблицу можно посмотреть вот тут. Так как на гистограмме два пика, длина белка считалась характерной, если находилась между ними (+- несколько остатков) - таким образом у половины всех белков наблюдается характерный размер.
Самое время прерваться на любование толстым енотом, который выглядит прямо как невыспавшаяся я на карантине:

HMM
Выравнивание выглядит довольно неплохо. Было вырезано достаточное количество белков,
чтобы консервативные блоки выглядели прилично, но результат до сих пор внушает опасения... Список выбранных
последовательностей можно посмотреть выше в таблице,
отфильтровав в экселе строчки по цвету последнего столбца. Их длины варьируются от 750 до 816. Кажется, изначально интервал был больше,
но рандомное тыканье на красивые имена из разных семейств сделало свое дело.
Для построения профиля были использованы следующие команды:
hmm2build -g profile.txt aligned.fa
hmm2calibrate profile.txt
hmm2search profile.txt HD.fasta > results.txt
В процессе этого выяснилось, что у HD-домена в Uniprot очень много записей, а именно 186049, и если вы думаете, что это много, просто попытайтесь запустить поиск по хеликазной штуковине. Если вы тут надеетесь увидеть таблицу с результатами, вас ждет разочарование: этот скрипт разбирается с входным файлом, выводит примитивную таблицу проверки предсказаний для порогового значения e-value 0.05 и записывает статистику для ROS-кривой в отдельный файл, который потом использует этот скрипт для построения красивых графиков, потому что рисовалки в питоне - зло.
Теперь о результатах. На обоих графиках бирюзовые прямые отсекают на осях координат значения, соответствующие выбранному порогу.
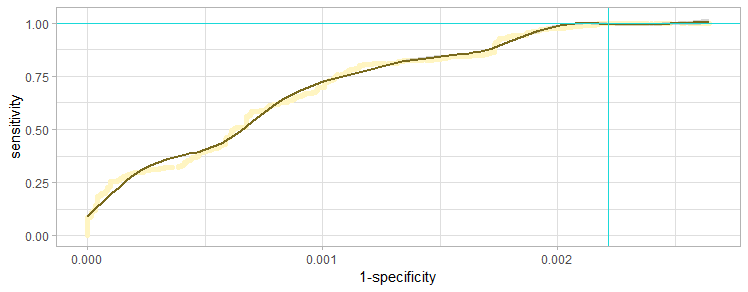
Алгоритм выдает результат всего лишь для 1360 последовательностей, e-value которых меньше 10. Именно поэтому в качестве итогового порога брался не вес, а обычное e-value в 0.05 - специфичность всегда получается крайне высокой, а чувствительность так и не дорастает до единицы, так как один белок алгоритм почему-то пропустил.
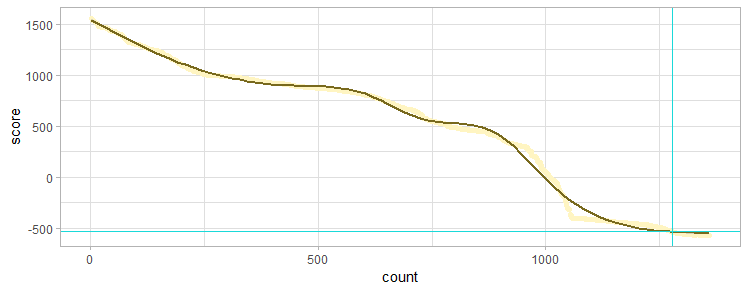
Этот график был построен немного неправильно: брались не номера, а количество находок с таким же весом или больше. Это казалось хорошей идеей, так как весы повторяются, и лестница все равно выглядит похоже.
| выборка Uniprot | |||
|---|---|---|---|
| Positive | Negative | ||
| поиск HMM | Positive | 861 | 399 |
| Negative | 8 | 185632 | |
Самое время прерваться на любование толстым енотом, который выглядит прямо как пытающаяся остатками продолговатого мозга написать скрипт в три ночи после двух стаканов виски я на карантине:

Специфичность у профиля действительно высока, чувствительность - тоже. В целом он работает довольно клево! Обилие найденных им белков - следствие того, что выбранная архитектура может быть окружена другими доменами. Несколько быстрых запросов в Uniprot показывают, что даже белки с довольно высоким e-value зачастую оказываются CRISPR-ассоциированными эндонуклеазами/хеликазами.
А что насчет одинокого белка из Uniprot, который HMM полностью проигнорировал? Это CRISPR-ассоциированные эндонуклеаза Cas3 из Listeria monocytogenes, патогенной бактерии, которая вызывает инфекцию, поражующую ЦНС и уничтожающую мозг... Миленько. Не она ли питается мозгом авторки этого практикума прямо сейчас?
На самом деле, для объяснения феномена достаточно взглянуть на выравнивание этого белка с теми, которые использовались для построения профиля. Его длина существенно короче, а N-концевой блок выравнивается примерно никак.
А вдруг N-концевой блок белка из нашей премилой Listeria monocytogenes выравнялся с остальными где-то раньше? Это было проверено ручками - нет там его. Ну и ладно, главное, CRISPR работает.