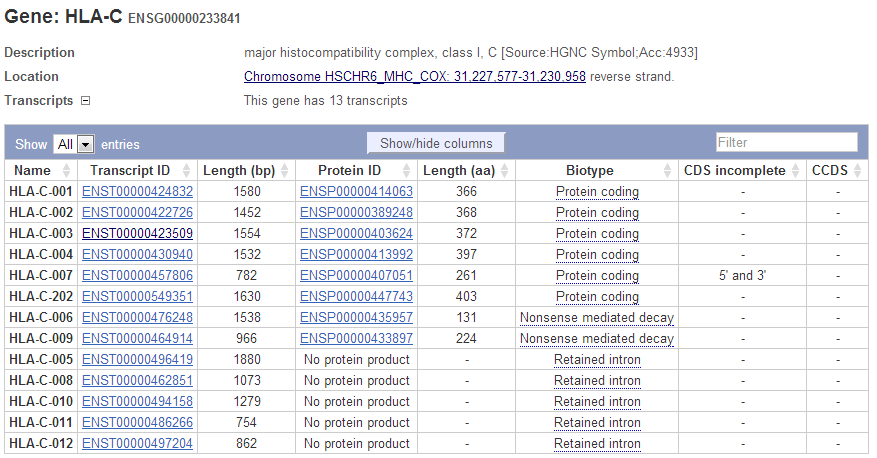
Данные ген - HLA-C, основная информация по нему описана в предыдущих работах.
Беру одну из последовательностей Задания 3
Занятия 2.1
(точнее - самый длинный экзон из данной мне последовательности этого задания).
Далее, провожу поиск BLAT по Genomic Sequence.
На странице с результатами представлены:
локация найденных выравниваний в человеческом геноме,
графическое отображение множественного выравнивания,
summary выравнивания -
представляет собой таблицу находок, формат и сортировку которой можно регулировать.
Первоначальная выдача такая:
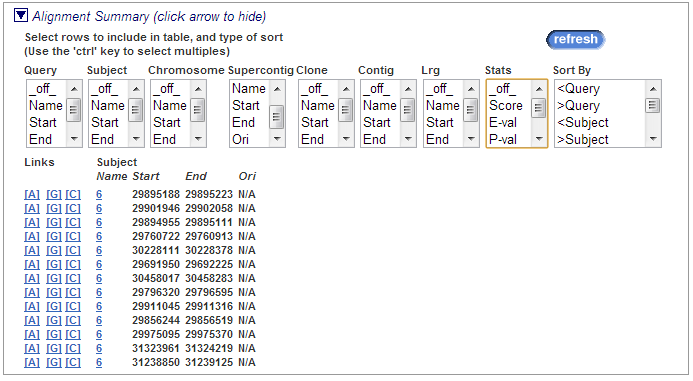
Я хочу посмотреть, в какой последовательности находки
располагаются на хромосоме, и формирую выдачу таким образом:
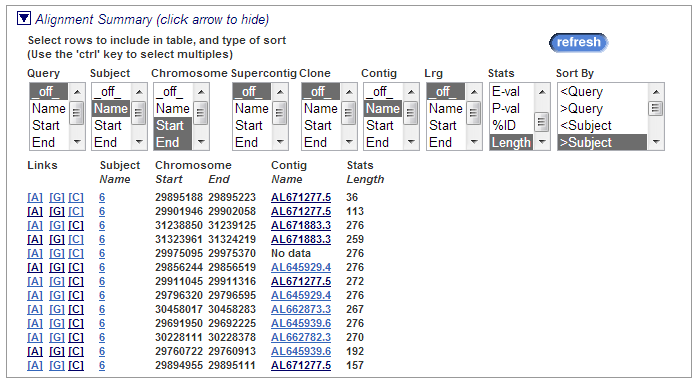
Ссылки:
[A] - посмотреть выравнивание с находкой,
[G] - последовательность находки с размеченными экзонами,
[C] - посмотреть данный контиг во всех подробностях.
Выбираю контиг AL671883.3, перехожу по ссылке [C].
Три картинки:
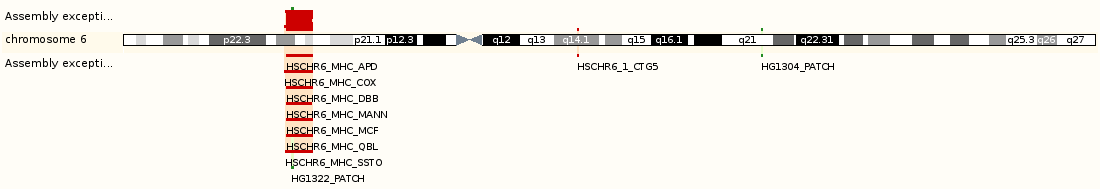
расположение на хромосоме, но не совсем понятно, чего именно. Возможно, разметка гаплотипа?
HSCHR6_MHC - локация HLA-C на участке хромосомы p22.3.

детальная картина участка генома, со всеми расположенными генами белков и РНК,
псевдогенами и процессируемыми транскриптами.

подробная картинка считываемых и процессируемых РНК. Подписаны и выделены те, с которых
итоге считывается белок, а также подвергающиеся nonsense madiated decay.
Ниже представлены: консенсусная кодирующая последовательность,
выравнивания с кДНК, график процентного GC-содержания и Assembly exceptions (не очень понятно, что это).
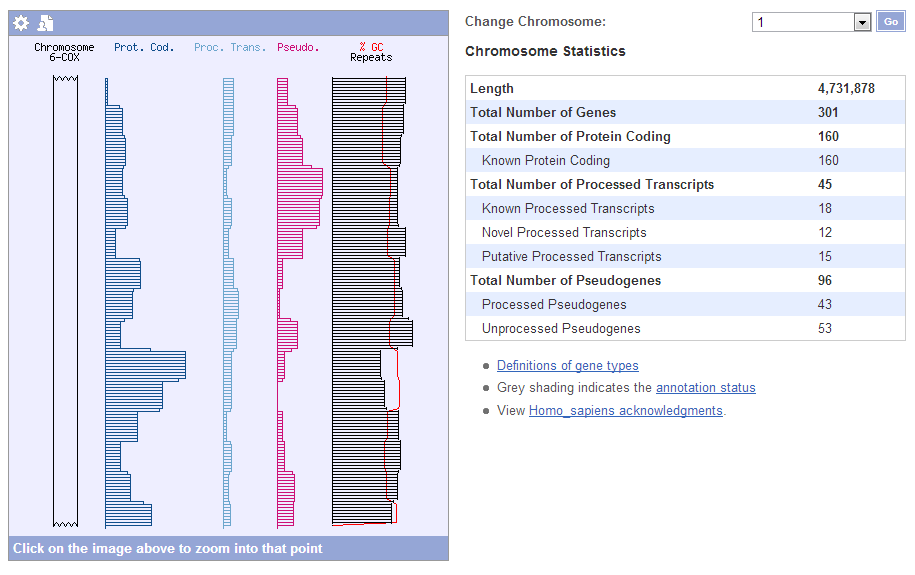
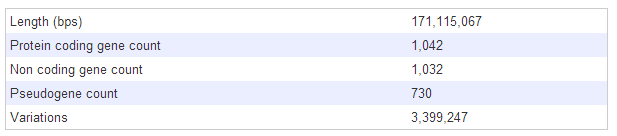
Интересным кажется пункт меню Chromosome summary.
Здесь приведена статистика по хромосоме (в моем случае - по 6-ой):
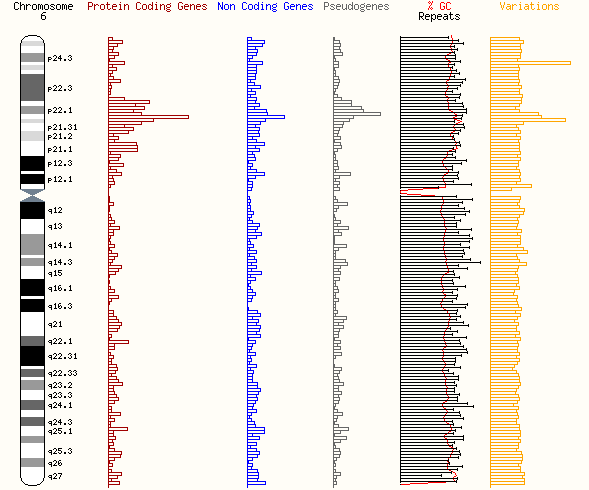
А также интересный и наглядный обощенный график распределений кодирующих участков, некодирующих участков,
псеводгенов, GC-повторов и вариативности:
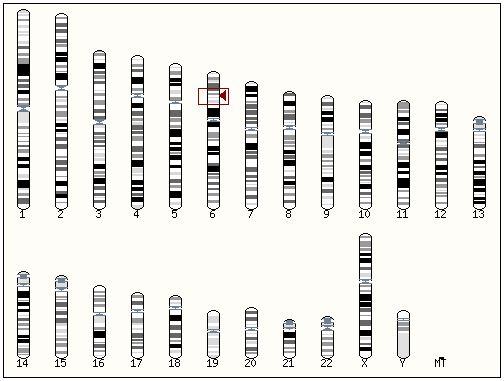
Перехожу в пункт Whole genome.
Здесь представлена разметка всего генома человека - 22 аутосомы,
X и Y половые хромосомы и митохондриальная хромосома.
Ниже - summary по геному, информация о последнем обновлении, версии, количестве нуклеотидов, генов, псевдогенов, экзонов,
транскриптов и много другой общей информации.
Интересный пункт меню - Synteny.
Здесь можно найти связи между хромосомами и генами человека и других организмов.
Почему-то, несмотря на то, что рассматриваемая хромосома - шестая,
браузер выдает информацию о синтении седьмой (возможно, баг сервера в подписи? на картинке отмечена шестая хромосома.)
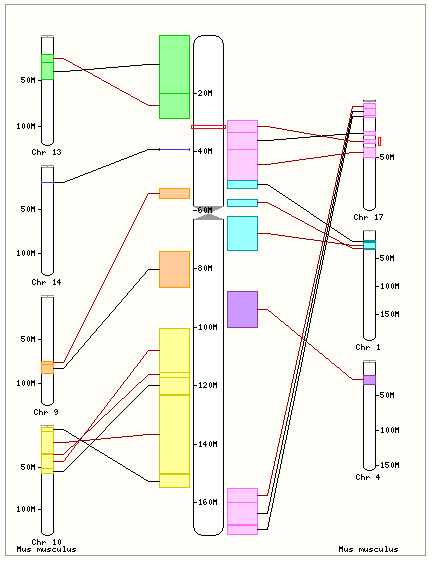
Гомологов гена HLA-C в мыши он не находит.
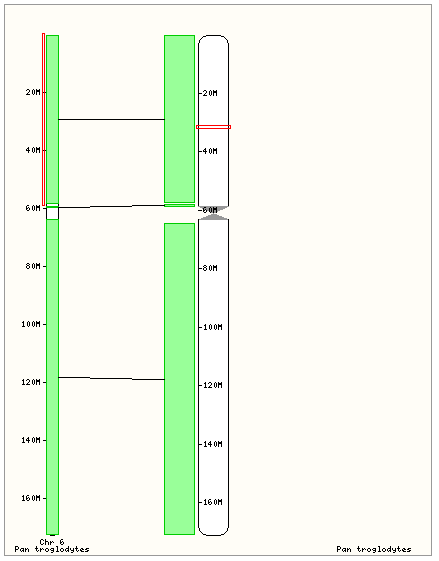
С шимпанзе всё гораздо лучше: находится гомолог Q198H2_PANTR (ENSPTRG00000041261).
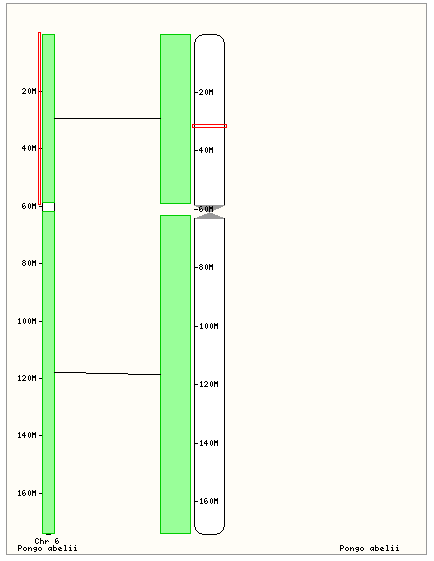
С орангутаном: гомолог HLA-C (ENSPPYG00000016427).
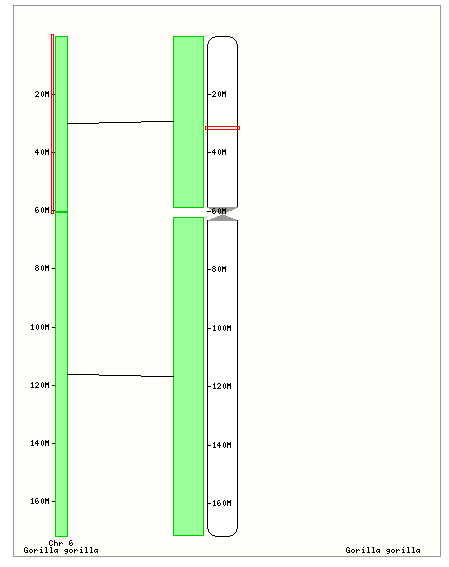
С гориллой: гомолог HLA-C (ENSGGOG00000006624).
Ссылки на другие браузеры.
UCSC
Выдает выравнивания данного участка хромосомы с различными последовательностями.
Работа с ним будет описана ниже. Картинка для участка данного контига (можно слдить за альтернативным спайсингом):

NCBI
Выдает мелкую, трудночитаемую картинку:

Ввожу в поиске на главной странице название выданного мне гена: HLA-C.
Результаты находятся для человека (28 генов, 1 фенотип, 6 соматических мутаций, 120 транскриптов, 30 вариаций),
шимпанзе (1 ген, 1 транскрипт), гориллы и орангутанга (по 1 гену).
Перехожу к списку генов HLA-C человека. Все они расположены в регионе HSCHR6_MHC (то есть участок MHC 6-ой хромосомы).
Причины такой множественности объяснимы: гены HLA формируют гаплотип (совокупность аллелей в одном геноме) на локусе p21.3 хромосомы 6.
Выбираю первый ген в списке: идентификатор ENSG00000233841, локация HSCHR6_MHC_COX:31227577-31230958:-1.
Локация:

Считываемые и процесированные мРНК:

Синим отмечены процессированные мРНК, красным - дающие разные варианты белков.
Информация и описание транскриптов гена:
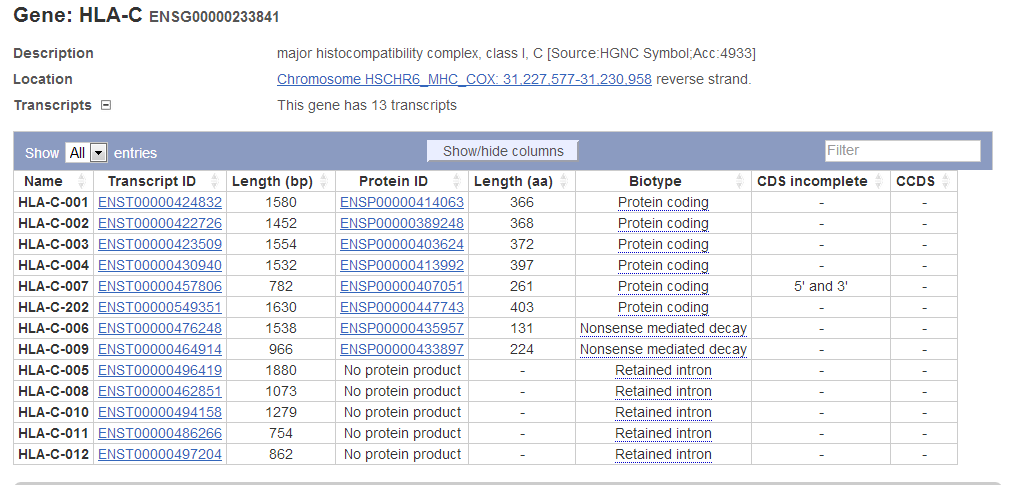
Меню Splice variants (13)

Меню Supporting evidence
Позволяет посмотреть картинку выравнивания
одного из предполагаемых транскриптов гена с
реальными белками, EST и кДНК.
Меню Sequence
Экзон-интронная структура на выравнивании:
>chromosome:GRCh37:HSCHR6_MHC_COX:31226977:31231558:-1
AGAATCTCTACCTGGTGCTTTCAGACAAAACTTCACCAGGTTTAAAGAGAAAACTCCTGA
CTCTACACGTCCATTCCCAGGGCGAGCTCACTGTCTGGCATCAAGTTCCCCATGGTGAGT
TTCCCTGTACAAGAGTCCAAGGGGAGAGGTAAGTGTCCTTTATTTTGCTGGATGTAGTTT
AATATTACCTGAGGTGAGGTAAGGTAAGGCAAAGGGTGGGAGGCAGGGAGTCCAGTTCAG
GGACGGGGATTCCAGGAGGAGAAGTGAAGGGGAAGGGGCTGGGCGCAGCCTTGGGGTCTC
TCCCTGGTTTCCACAGACAGATCCTTGTCCAGGACTCAGGCACACAGTGTGACAAAGATG
CTTGGTGTAGGAGAAGAGGGATCAGGACGAAGTCCCAGGTCCCGGGCGGGGCTCTCAGGG
TCTCAGGCTCCAAGGGCCGTGTCTGCATTGGGGAGGCGCCGCGTTGGGGATTCTCCACTC
CCCTGAGTTTCACTTCTCCCAACCTGCGTCGGGTCCTTCTTCCTGAATACTCATGACGCG
TCCCCAATTCCCACTCCCATTGGGTGTCGGGTTCTAGAGAAGCCAATCAGCGTCTCCGCA
GTCCCGGTTCTAAAGTCCCCAGTCACCCACCCGGACTCACATTCTCCCCAGAGGCCGAGA
TGCGGGTCATGGCGCCCCGAGCCCTCCTCCTGCTGCTCTCGGGAGGCCTGGCCCTGACCG
AGACCTGGGCCTGTGAGTGCGGGGTTGGGAGGGAAGCGGCCTCTGCGGAGAGGAGCGAGG
GGCCCGCCCGGCGAGGGCGCAGGACCCGGGGAGCCGCGCAGGGAGGTGGGTCGGGCGGGT
CTCAGCCCCTCCTCGCCCCCAGGCTCCCACTCCATGAGGTATTTCGACACCGCCGTGTCC
CGGCCCGGCCGCGGAGAGCCCCGCTTCATCTCAGTGGGCTACGTGGACGACACGCAGTTC
GTGCGGTTCGACAGCGACGCCGCGAGTCCGAGAGGGGAGCCGCGGGCGCCGTGGGTGGAG
CAGGAGGGGCCGGAGTATTGGGACCGGGAGACACAGAACTACAAGCGCCAGGCACAGGCT
GACCGAGTGAGCCTGCGGAACCTGCGCGGCTACTACAACCAGAGCGAGGACGGTGAGTGA
CCCCGGCCCGGGGCGCAGGTCACGACCCCTCCCCATCCCCCACGGACGGCCCGGGTCGCC
CCGAGTCTCCCCGTCTGAGATCCACCCCAAGGTGGATCTGCGGAACCCGCCCAGACCCTC
GACCGGAGAGAGCCCCAGTCGCCTTTACCCGGTTTCATTTTCGGTTTAGGCCAAAATCCC
CGCGGGTTGGTCGGGGCGGGGCGGGGCTCGGGGGACTGGGCTGACCGCGGGGGCGGGGCC
AGGGTCTCACACCCTCCAGAGGATGTATGGCTGCGACCTGGGGCCCGACGGGCGCCTCCT
CCGCGGGTATGACCAGTCCGCCTACGACGGCAAGGATTACATCGCCCTGAACGAGGACCT
GCGCTCCTGGACCGCCGCGGACACCGCGGCTCAGATCACCCAGCGCAAGTTGGAGGCGGC
CCGTGCGGCGGAGCAGCTGAGAGCCTACCTGGAGGGCACGTGCGTGGAGTGGCTCCGCAG
ATACCTGGAGAACGGGAAGGAGACGCTGCAGCGCGCAGGTACCAGGGGCAGTGGGGAGCC
TTCCCCATCTCCTATAGATCTCCCGGGATGGCCTCCCACGAGGAGGGGAGGAAAATGGGA
TCAGCACTGGAATATCGCCCTCCCTTGAATGGAGAATGGCATGAGTTTTCCTGAGTTTCC
TCTGAGGGCCCCCTCTGCTCTCTAGGACAATTAAGGGATGAAGTCTCTGAGGAAATGGAG
GGGAAGACAGTCCCTGGAATACTGATCAGGGGTCTCCTTTGACCACTTTGACCACTGCAG
CAGCTGTGGTCAGGCTGCTGACCTTTCTCTCAGGCCTTGTTCTCTGCCTCACACTCAATG
TGTCTGAAGGTTTGATTCCAGCTTTTCTGAGTCCTGCAGCCTCCACTCAGGTCAGGACCA
GAAGTCGCTGTTCCTCCCTCAGAGACTAGAACTTTCCAATGAATAGGAGATTATCCCAGG
TGCCTGTGTCCAGGCTGGCGTCTGGGTTCTGTGCCGCCTTCCCCACCCCAGGTGTCCTGT
CCATTCTCAGGATGGTCACATGGGCGCTGCTGGAGTGTCCCAAGAGAGATGCAAAGTGTC
TGAATTTTCTGACTCTTCCCGTCAGAACCCCCAAAGACACACGTGACCCACCACCCCCTC
TCTGACCATGAGGCCACCCTGAGGTGCTGGGCCCTGGGCTTCTACCCTGCGGAGATCACA
CTGACCTGGCAGCGGGATGGGGAGGACCAGACCCAGGACACCGAGCTTGTGGAGACCAGG
CCAGCAGGAGATGGAACCTTCCAGAAGTGGGCAGCTGTGGTGGTGCCTTCTGGACAAGAG
CAGAGATACACGTGCCATATGCAGCACGAGGGGCTGCAAGAGCCCCTCACCCTGAGCTGG
GGTAAGGAGGGGAATGGGGGGTCACATCTCTTATCAGAGAAAGCAGAAGTCCTTCTGGAG
CCCTTCAGCCGGGTCAGGGCTGAGGCTTGGGGGTCAGGGCCCCTCACCTTCTCCTCCTTT
CCCAGAGCCATCTTCCCAGCCCACCATCCCCATCATGGGCATCGTTGCTGGCCTGGCTGT
CCTGGTTGTCCTAGCTGTCCTTGGAGCTGTGGTCACCGCTATGATGTGTAGGAGGAAGAG
CTCAGGTAGGGAAGGGGTGAAGAGCGGGGTCTGGGTTTTCTTGTCCCACTGGGAGTTTCA
AGCCCCAGGTAGAAGTGTGCCCCGCCTTGTTACTGGAAGCACCATCCACACATGGGCCAT
CCCAGCCTGGGACCCTGTGTGCCAGCACTTACTCTTTTGTGAAGCACATGTGACAATGAA
GGACGGATGTATCACCTTGATGATTATGGTGTTGGGGTCCTGATTCCAGCATTCATGAGT
CAGGGGAAGGTCCCTGCTAAGGACAGACCTTAGGAGGGCAGTTGGTCCAGAACCCACAAC
TGCTTTCCCCATGTTTCCTGATCCTGCCCTGGGTCTGCAGTCGTAGTTCTGGAAACTTCT
CTTGGGTCCAAGACTAGGAGGTTCCCCTAAGATCACATGGCCCTGCCTCCTCCCAGTCCC
CTCATAGGGCATTTTCTTCCCACAGGTGGAAAAGGAGGGAGCTGCTCTCAGGCTGCGTGT
AAGTGATGGCGGCGGGCGTGTGGAGGAGCTCACCTACTCCATAATTCCTCTTGTCCCACA
TCTCCTGCGGGCTCTGACCAGGTCTTTTTTTTTGTTCTACCCCAGGCAGCAACAGTGCCC
AGGGCTCTGATGAGTCTCTCATCACTTGTAAAGGTGAGATTCTGGGGAGCTGAAGTGGTC
GGGGGTGGGGCAGAGGGAAAAGGCCTGGGTAATGGGGATTCTTTGATTGGGACGTTTCGA
GTGTGTGGTGGGCCGTTCAGAGTGTCATCACTTACCATGACTGACCTGAATTTGTTCATG
ACTATTGTGTTCTGTAGCCTGAGACAGCTGCCTGTGTGGGACTGAGATGCAGGATTTCTT
CACACCTCTCCTTTGTGACTTCAAGAGCCTCTGGCATCTCTTTCTGCAAAGGCGTCTGAA
TGTGTCTGCGTTCCTGTTAGCATAATGTGAGGAGGTGGAGAGACAGCCCACCCCCGTGTC
CACCGTGACCCCTGTCCCCACACTGACCTGTGTTCCCTCCCCGATCATCTTTCCTGTTCC
AGAGAGGTGGGGCTGGATGTCTCCATCTCTGTCTCAAATTCATGGTGCACTGAGCTGCAA
CTTCTTACTTCCCTAATGAAGTTAAGAACCTGAATATAAATTTGTGTTCTCAAATATTTG
CTATGAAGCGTTGATGGATTAATTAAATAAGTCAATTCCTAGAAGTTGAGAGAGCAAATA
AAGACCTGAGAACCTTCCAGAATTTGCATGTTCGCTGTGCTGAGTCTGTTGCAGGTGGGG
GTGGGGAAGGCTGTGAGGAGCCGAGTGTGGACGGGGCCTGTGCCTAGTTGCTGTTCAGTT
CTTCATGGGCTTTATGTGGTCAGTCCTCAGCTGGGTCACCTTCACTGCTCCATTGTCCTT
GTCCCTTCAGTGGAAACTTGTCCAGCGGAAGCTGTGACCACAGAGGCTCACCCATCGCCC
AGGGCAGCCCCTGCACACGGGAGTCCCTGTGCTTTCTGAGACAAATTTTCAGACCCATTC
AGCTCCTGCCCTCCTTCTAGGGCTCCTCTTCTGCTTTGGTCTCCTGCCCTCTCTCCCTTC
CCTGATTCCAGTGATCTTCGTGCTGACTCCAATCCCAACTCATGAATCTAAAGCAGAGCC
TAATTTAGATTTGTATTTGTTTGTAAAATTGGGTCCATAGTCTAGAATTGTTCCTTCCTG
AAGAGAGAAACCTGATCGTGTGCTGCAGTGTGCGGGGCGGTTGGTGTGGGAGGAGGGATA
GGGGAGGGAGGACACACAAGCAGCCCTGCTGAGAAAAGTACAGGCGGCCTCGGTGTCAGT
GTGAGGGGACCTTGTGCTGCAG
Меню External References
Позволяет найти ссылки о гене на другие базы данных. Здесь можно выяснить другие названия гена.
Например, я выясняю, что выбранная мною аллель гена: CW*08.
Меню Regulation
Теоретически позволяет посмотреть регуляротные сайты. Но для моего гена они отсутствуют.
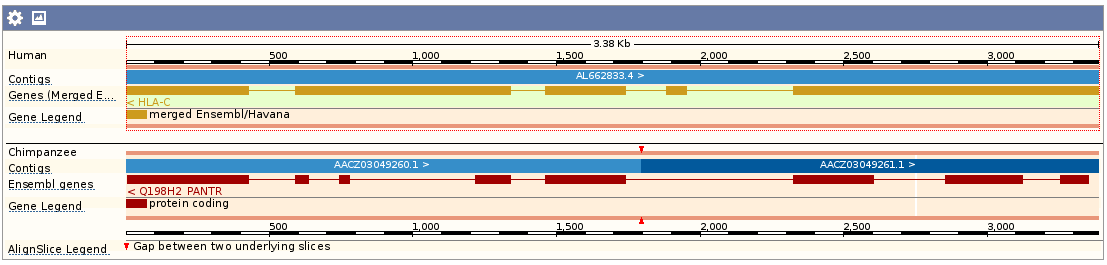
Меню Comparative Genomics
Позволяет выравнивать, искать паралоги, ортологи, сторить деревья, находить семейства белков для гена.
Для моего гена доступны Genomic Alignments, Protein families.
Меню Genomic alignments
Позволяет выровнять последовательность гена с другими организмами. Доступно в текстовом формате и в формате картинки.
Я рассматривала выравнивания с шимпанзе (хорошее, два фрагмента) и с мышью (не такое хорошее, больше фрагментов).
Не понятно, что значит пометки lastz / blastz рядом с названиями организмов, предлагаемых для выравнивания.
Целое выравнивание приводить бессмысленно. Пример заголовка выравнивания:
Homo sapiens › chromosome:GRCh37:HSCHR6_MHC_COX:31226977:31231558:-1
Pan troglodytes › chromosome:CHIMP2.1.4:6:31639239:31641419:-1
chromosome:CHIMP2.1.4:6:31636478:31638865:-1
Protein families
Меню Phenotype
Здесь можно найти список фенотипических изменений, связанных с вариациями гена.
Есть список ссылок на аннотации фенотипов, в которых упоминается данный ген.
У моего гена в первой таблице прописано "Предрасположенность к псориазу". Перехожу по ссылке
и читаю, что с псориазом больше всего связана аллель HLA-Cw6. Здесь же
узнаю, что такое псориаз - хронический дерматоз, встречающийся у 2% человеческой популяции.
Меню Genetic Variation
Позволяет подробнее узнать о различных экзон-интронных структурах гена.
Варианты меню Location
Ничего особенного и нового не нахожу для данного региона.
Проверяю генетические вариации.
Genetic Variation, Resequencing
Здесь можно проверить, "хорошо" ли было с данным локусом у Вентера и Уотсона. В гене HLA-C, в их генотипах нет никаких мутаций.
Немного информации из UCSC.
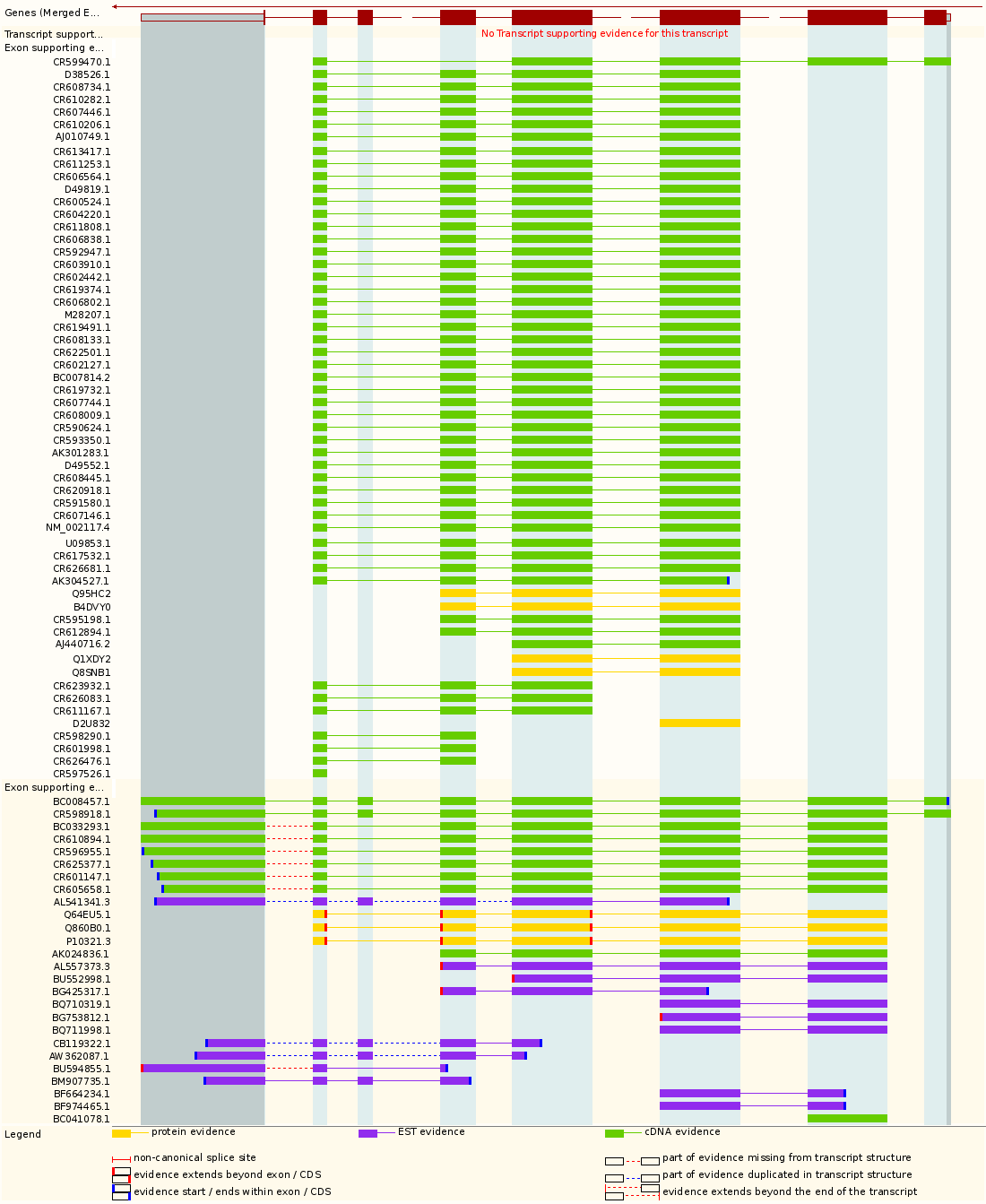
Выдает выравнивание данного участка хромосомы с различными существующими последовательностями.
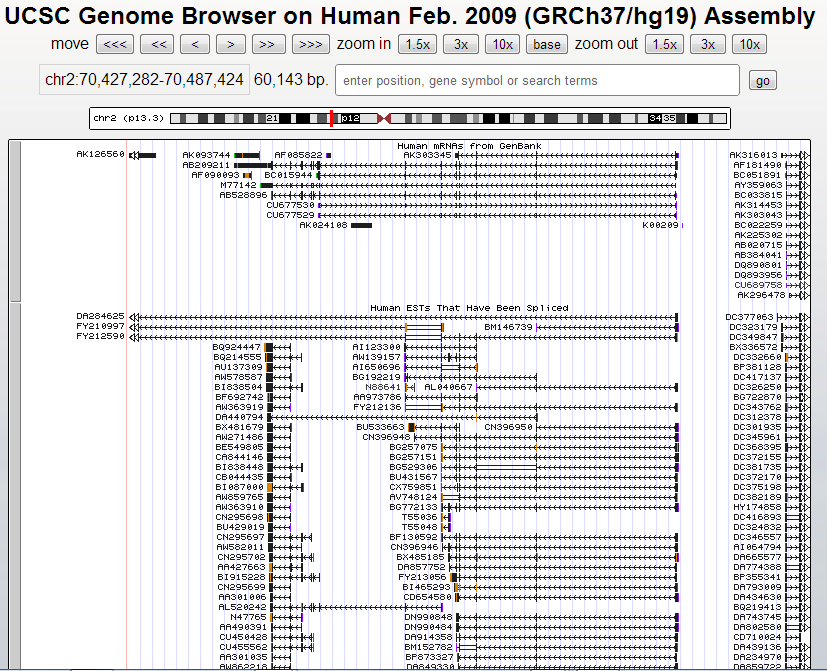
Примечательно, что такие свойства отображения, как тип (hide - спрятать, dense, squish, pack, full), отображаемые выравнивания (Human mRNA, EST), регулируются и меняются, в зависимости от
требований. Например, здесь поставлено Human mRNA - pack, Humans ESTs - pack.
Учень удобный наглядный сервис. Можно отслеживать расположение генов, альтернативный сплайсинг, nonsense mediated decay.
Из Vega:
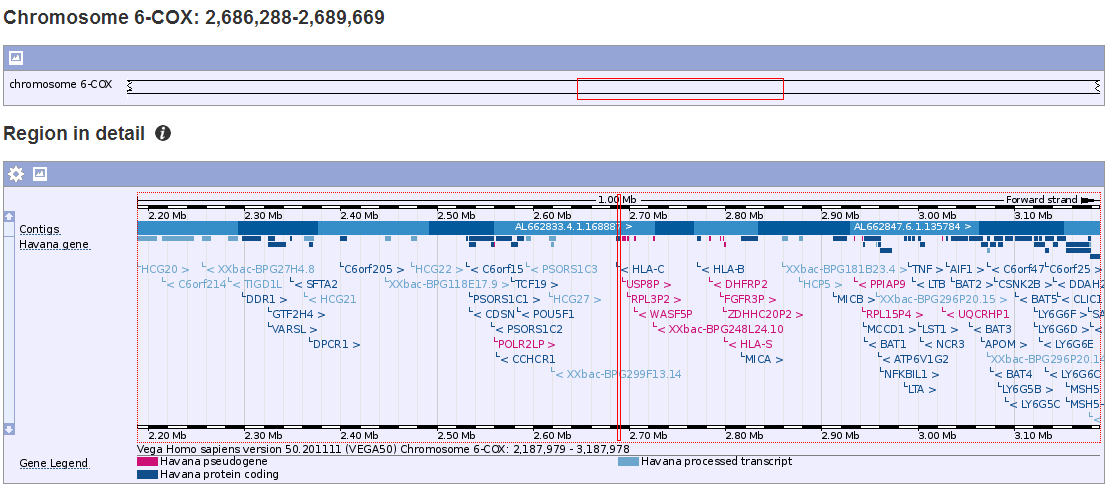
Сразу бросается в глаза другая, сглаженная цветовая гамма.
Непривычно ориентироваться по сравнению с резкими различиями цветов в ensembl.
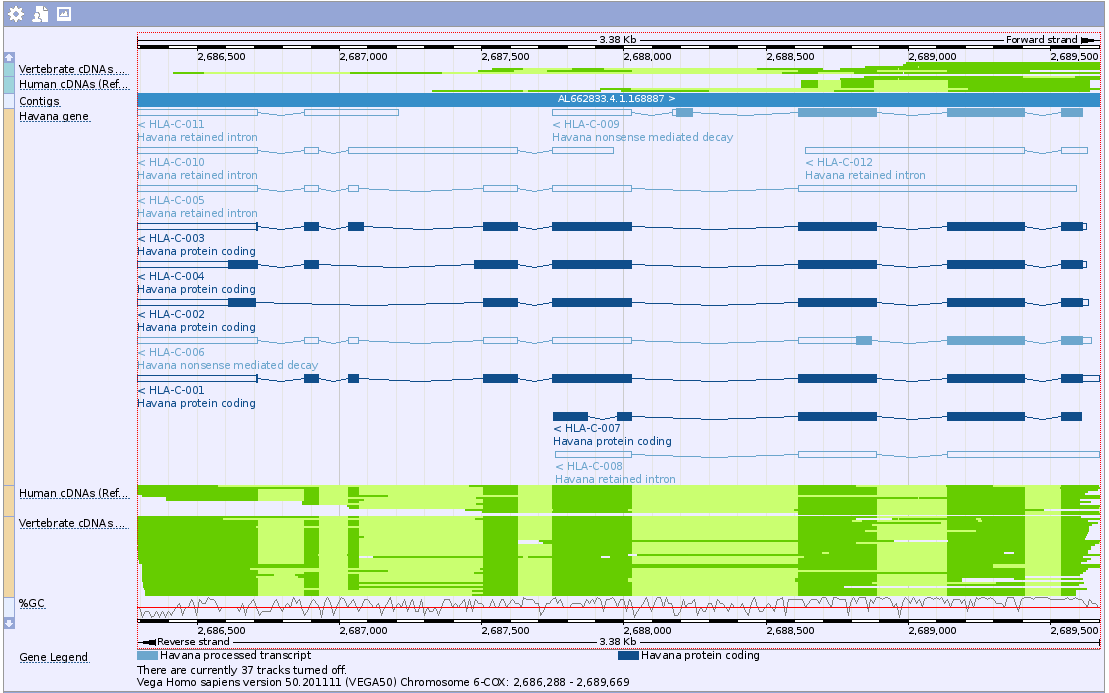
Любопытное представление Chromosome summary, на мой взгляд горадзо более наглядное, чем в ensembl: