Киотская энциклопедия генов. Задание 6.
1. Метаболические пути с участием фермента MECR_HUMAN
Название фермента: MECR_HUMAN
KEGG: hsa:51102
("hsa" означает Homo sapiens).
Адрес KEGG: http://www.genome.jp/kegg/
Поиск по идентификатору привел на такую страничку:
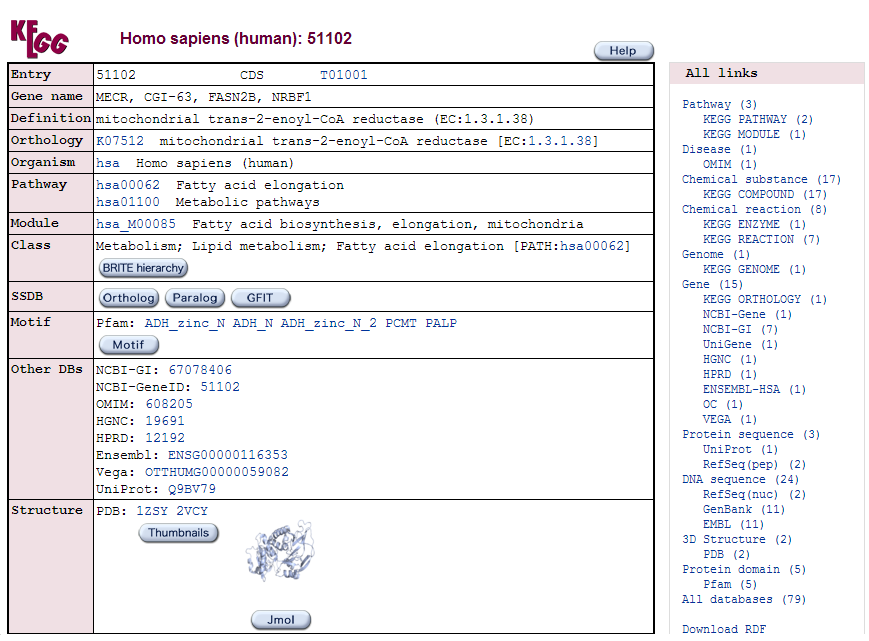
На страничке - таблица, где можно найти: имя гена, кодируемый фермент с
EC-шником, организм, задействованные метаболические пути,
мотивы белка, ссылки на другие базы данных, структуру, позицию
в хромосоме, аминокислотную и нуклеотидную соответствующие
последовательности.
Сейчас интерес представляет строка "Pathway" с ссылками на карты метаболических путей, ассоциированных с геном.
Вторая ссылка "hsa01100 Metabolic pathways" ("Метаболические пути") - не представляет практического
интереса, она приводит к огромной общей карте всех метаболических путей,
описанных в KEGG. Цветом выделены разные группы путой, при достаточном
увеличении можно различить отдельные циклы, ссылки.
На всякий случай ссылка: hsa01100 Metabolic pathways
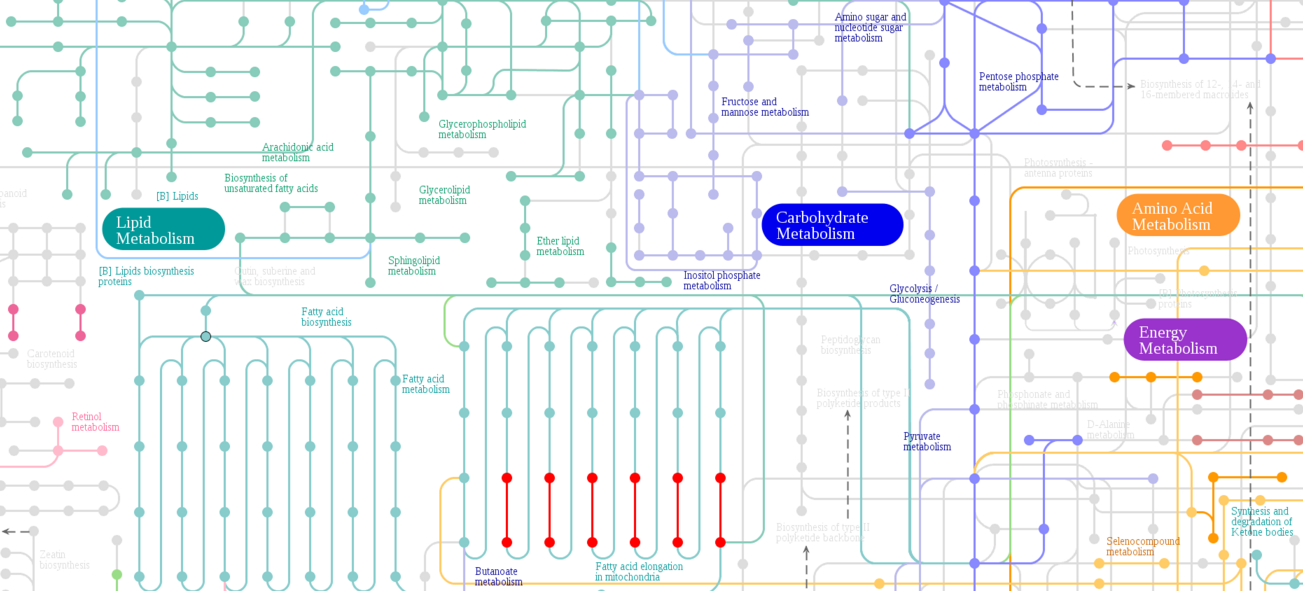
Например, вот область карты, о путях которой будет идти речь дальше:
Отдельно путь элонгации жирных кислот:
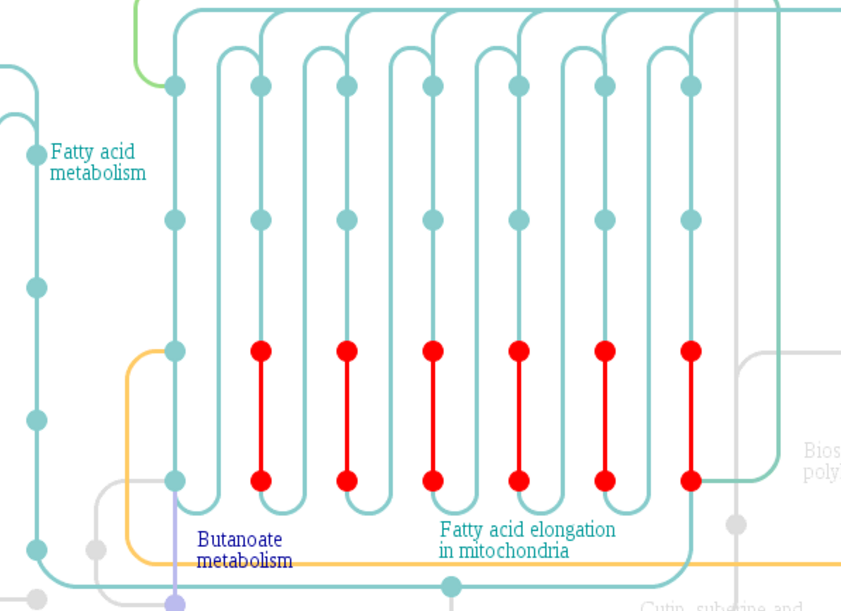
Как видно, он сопряжен с путями биосинтеза и метаболизма жирных кислот
("налево" по схеме), метаболизмом бутаноата, гликолизом и метаболизмом
пирувата ("направо" по схеме).
Теперь о первой ссылке: "hsa00062 Fatty acid elongation" ("Элонгация (процессинг) жирных кислот")
Карта этого пути, много более понятная, чем в предыдущем описании:
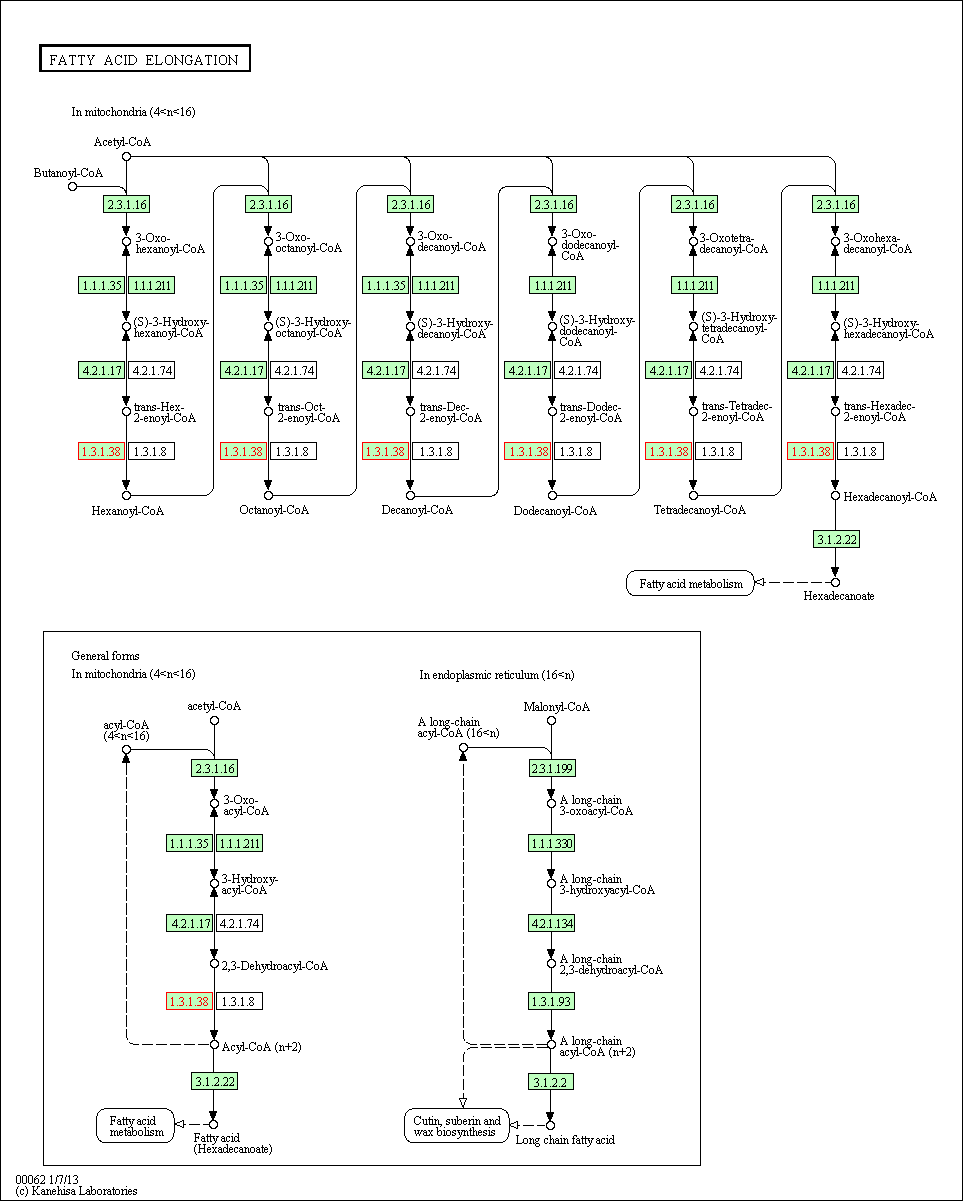
Ссылка: hsa00062 Fatty acid elongation
Мой фермент описан в митохондиальным пути элонгации (или процессинга) жирных кислот,
на каждом этапе удлиннения цепи кислоты осуществляет последний
этап перед очередным удлинненнием - гидрирование.
Например, судя по схеме, мой фермент необратимо осуществляет превращение
2-транс-додеканоил-КоА (C03221) в додеканол-КоА (C01832):
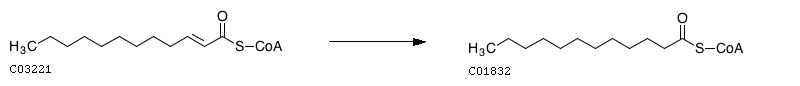
Примечательно, что в EC 1.3.1.38описана
обратная реакция - дегидратация. (также см. предыдущее занятие)
Теперь важным кажется замечание, что фермент работает с транс-формами дегидроацил-КоА (еноил-КоА),
в отличие от активности фермента 1.3.1.37, работающего с цис-формой. Этот факт подчеркивается в каждом описании EC 1.3.38, как в SRS, так и в KEGG.
Большая работа по рассмотрению гена проделана в пункте 5.1 текущей работы.
2. Структурные формулы заданных соединений
Требуется последовательно перейти по ссылкам с главной страницы и далее:
"KEGG2" ("KEGG Table of Contents") -> "KEGG LIGAND".
Вещества и требуемая информация о них:
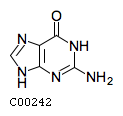
гуанин - guanine - C00242
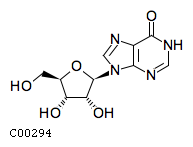
инозин - inosine - C00294
3. Метаболический путь от гуанина к инозину
С главной страницы требуется перейти: "KEGG PATHWAY" ->
"Search&Color Pathway" (сразу в разделе "Pathway Mapping").
Поиск по "Reference pathway". В поле ввожу:
C00242 red
C00294 green
Выдача поиска:
ko00230 Purine metabolism (2)
ko01100 Metabolic pathways (2)
Перехожу по первой, значимой ссылке.
Карта, на которой действительно присутствует окрашенными и гуанин, и инозин (окраска для акцентирования внимания):
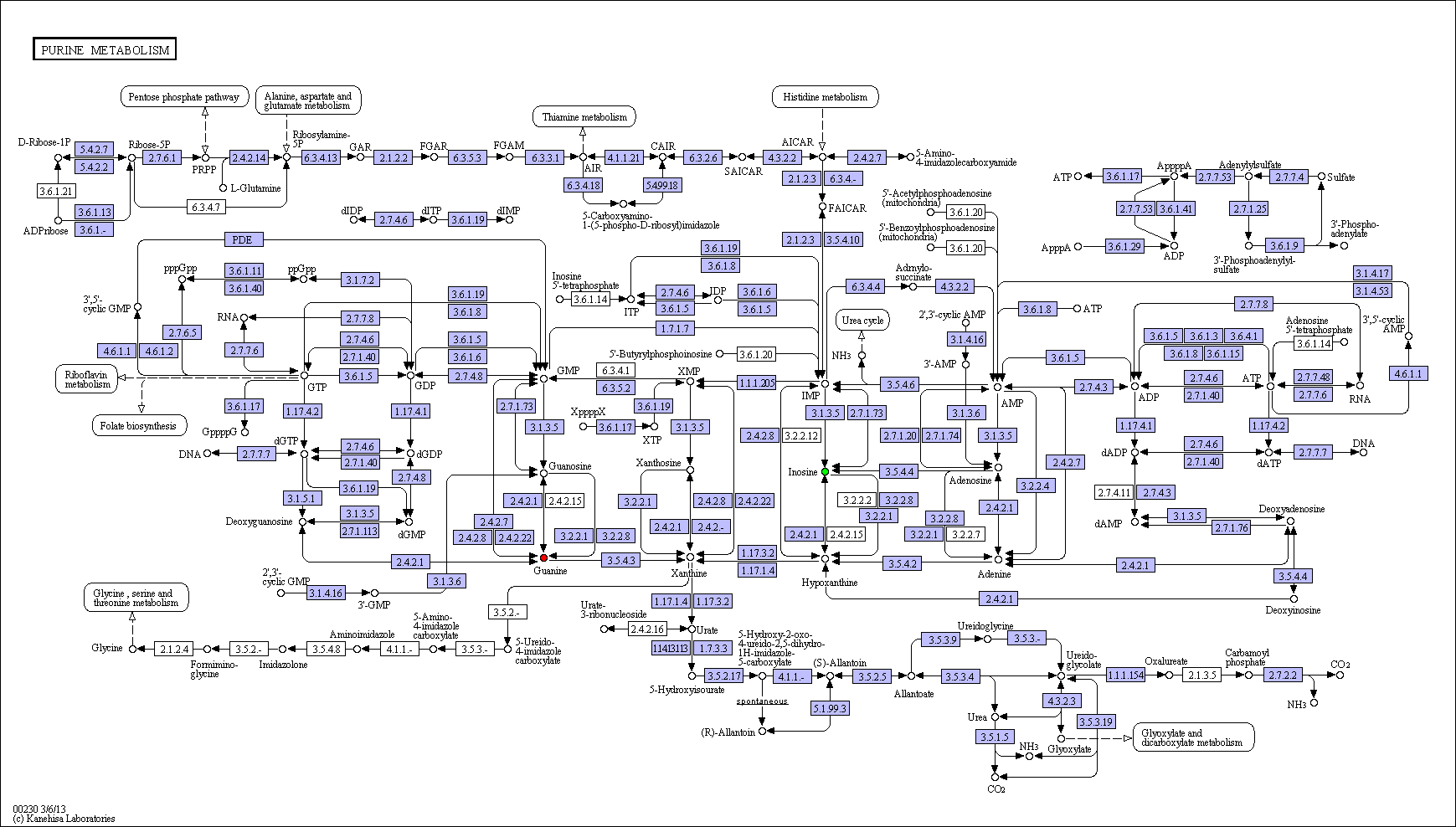
Выбираю кратчайший путь, метаболически соединяющий данные мне вещества, запрашиваю новую картинку с окрашенными интермедиатами.
Карта: метаболизм пуринов
Путь: инозин (С00294) -> гуанин (С00242)
Путь с интермедиатами:
инозин (С00294) <-> IMP (C00130 инозинмонофосфат)
-> XMP (C00655 ксантинмонофосфат)
-> GMP (C00144 гуанозинмонофосфат)
<-> гуанин (С00242)
Выбранная цепочка необратима, следовательно, инозин как
исходное соединение - выделено красным, интермедиаты - желтым, гуанин как конечное - зеленым.
Новый запрос:
C00294 red
C00130 yellow
C00655 yellow
C00144 yellow
C00242 green
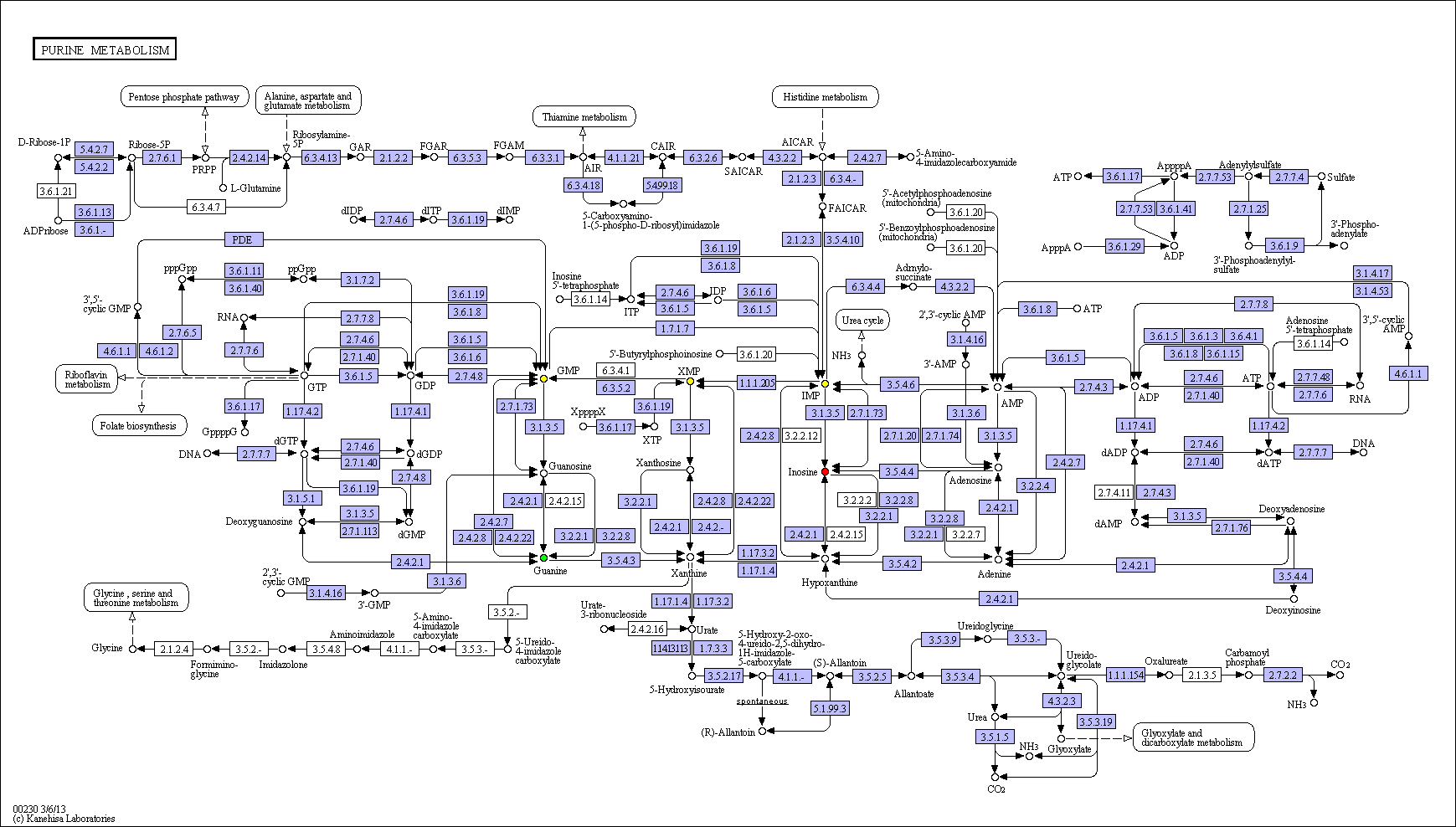
Ссылка: Полученная карта с требуемой раскраской
4. Сравнение выбранного метаболического пути у разных организмов
Возможность выбранной цепочки ферментативных реакций в разных организмах
| Организм |
Цепочка реакций возможна
(да/нет/неизвестно) |
Обоснование |
| Bacillus subtilis |
Нет выбранной прямой цепочки (неизвестно?) |
Неизвестен фермент 2.7.1.73, осуществляющий прямое
превращение инозин->ИМФ, но возможен обходной путь через гипоксантин.
Известны 5 из 7 ферментов. |
| Archaeoglobus fulgidus |
Неизвестно |
Ничего не известно про метаболизм гуанина,
нет данных о превращении инозина в ИМФ. Известны 2 из 7 ферментов. |
| Arabidopsis thaliana |
Нет (неизвестно?) |
Неизвестны пути превращения инозина в ИМФ.
Известны 4 из 7 ферментов. |
| Homo sapiens |
Нет выбранной прямой цепочки (неизвестно?) |
Неизвестен фермент 2.7.1.73, осуществляющий прямое превращение
инозин->ИМФ, но возможен обходной путь через гипоксантин.
Известны 4 из 7 ферментов. |
Картинка для арабидопсиса (Arabidopsis thaliana):
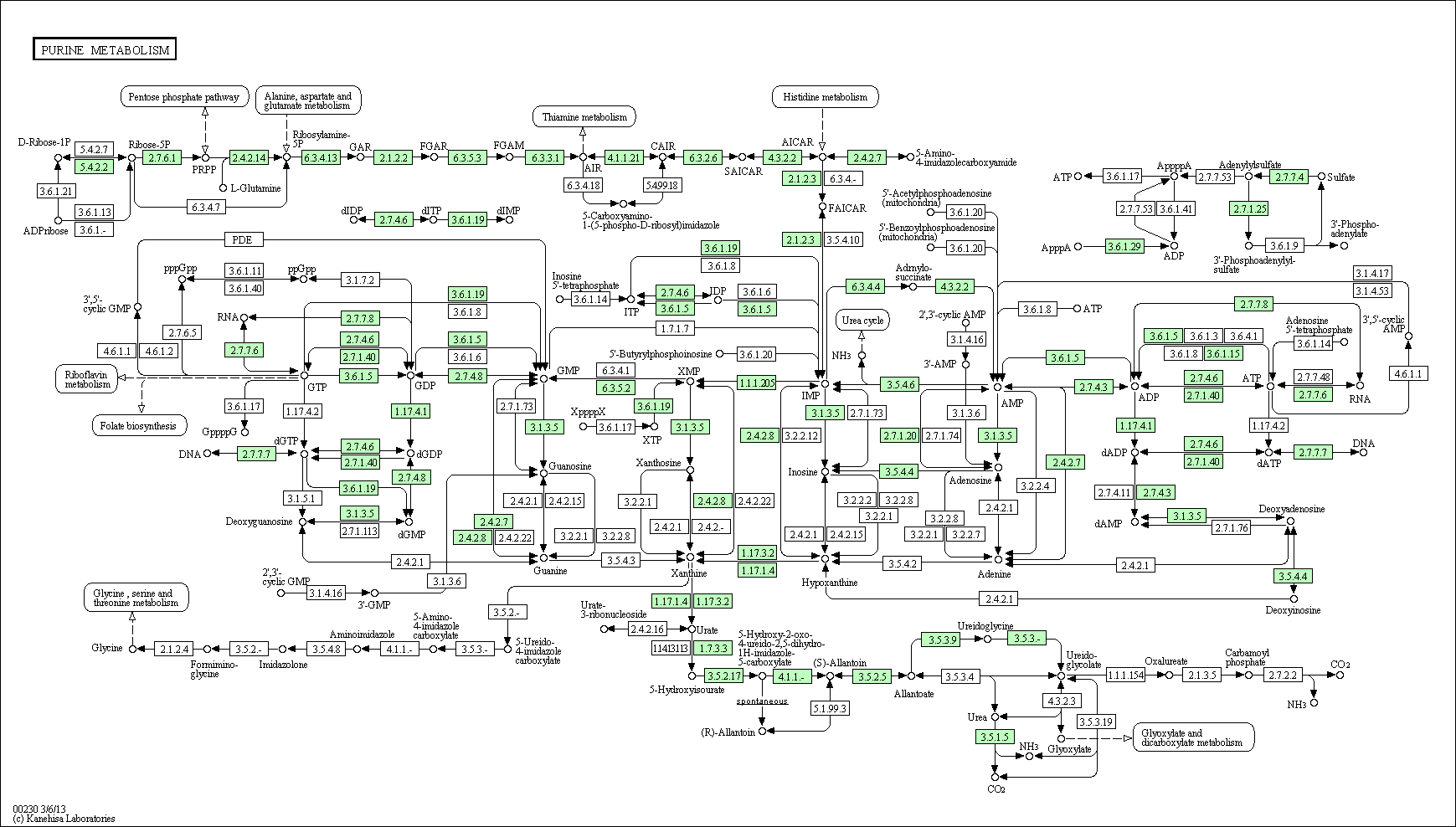
5. Дополнительное задание
5.1. К заданию 1. Что ещё можно узнать о гене.
Хочется подробнее рассмотреть страничку о гене:
http://www.genome.jp/dbget-bin/www_bget?hsa:51102
В строке Entry приведена ссылка на статью о полном геноме организма, из которого взят данный ген.
Эта статья - http://www.genome.jp/dbget-bin/www_bget?gn:T01001
имеет свои ссылки на другие базы данных; описание таксономии организма;
статистику по количеству белок-кодирующих и РНК-кодирующих генов; ссылки на статьи, в которых использовался данный геном.
Снова возвращаюсь на страничку о гене.
Далее следует всякая поезная информация об имени гена,
функции кодируемого белка, участие в метаболизме, ссылки
на список ортологов и паралогов, ссылки на другие базы данных, PDB.
Картиночка белка, приведенная здесь:
5.2. KEGG API.
KEGG предоставляет API (application programming interface) для
настраиваемого пользования энциклопедией. Для биоинформатика это дает возможность
автоматизировать и упростить поиск.
Ссылка на
мануал
по KEGG API, описано, например, как добраться до KEGG с помощью Perl или Python.
Мне, интересно, как сформулировать запрос к KEGG в форме URL:
описано здесь
Например, для первого задания, запрос http://rest.genome.jp/link/hsa:51102
выдает список перекрестных ссылок с другими базами данных.
На запрос http://rest.genome.jp/get/cpd:C00242+cpd:G00294 будут выданы полные статьи о
веществах из задания 2, разделенные "//".
5.3. Другие возможности KEGG.
"KEGG Organisms: Complete Genomes" - содержит полную таблицу всех организмов, описанных в KEGG.
Количество описанных орагнизмов: 185 эукариот, 2151 бактерия, 149 архей.
В таблице приведена некая классификация, сокращенное и полное название, а также ссылки на полные геномы. Каждый элемент талицы представляет собой гиперссылку, так что интересующий таксон можно рассмотреть отдельно от других, например, и увидеть, для каких организмов имеются полные геномы.
"KEGG BRITE" - иерархические классификации, охватывают данные из геномики, транскриптомики, протеомики и метаболомики
"KEGG DISEASE" - заболевания человека
"KEGG DRUG" - классификация лекарств
Страница семестра
© Галицына Александра, 2013










