Практикум 8
Задание 1
К сожалению сборки контигов содержащие информации больше чем последовательность ориджина не вышло.Поэтому я взяла участок хромосомы А1 кодирующий ген GJB2,дефекты которого приводят к глухоте или тугоухости.[1]
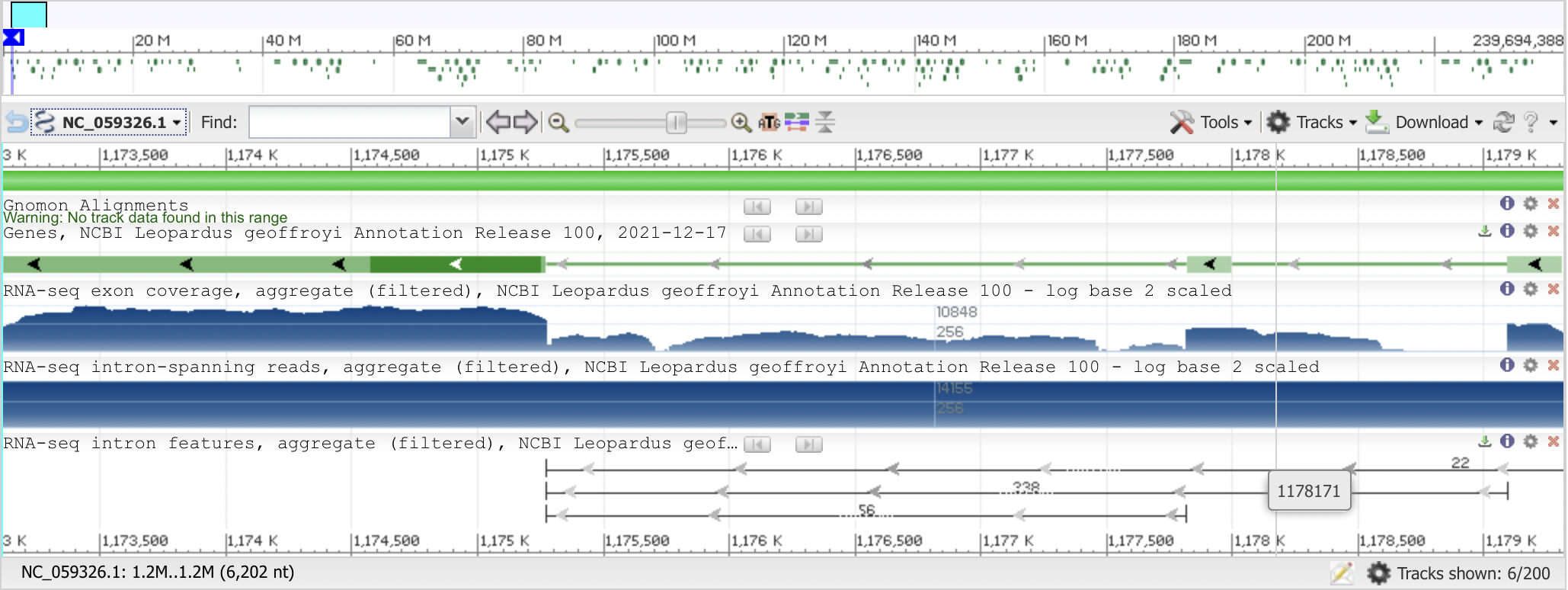
Скачать последовательность можно по ссылке.
Далее будем работать с этой последовательностью прогоняя по ней BLAST исключая хордовых.
В поиске по blastn было найдено большое количество синтезированных генов, что неудивительно для гена важным с медицинской точки зрения, но на удивление были найдены гомологи среди малярийных комаров Anopheles(притом с E-value 0.0 и Identites 626/710(88%)) и некоторые актиний.Всего находок выдало 93 штуки.
При прогонке через megablast количество находок резко снизилось,их всего 11,в основном те же искусственно синтезированные, но последовательность принадлежащая малярийному комару также сохраняется(данные те же что и представленые выше), хотя актинии пропали, вероятно из-за малого процента идентичности.
Blastx выдал 71 последовательность.Что неудивительно так как, если я правильно выяснила все функции белка, он также зашивает гэпы.По идее ничего особо нового он не выдал, также синтезированные белки и малярийный комар(E-value 5e-144,Identities 219/226(97%)) .Однако надо отметить откуда-то взявшуюся последовательность Klebsiella pneumoniae (E-value 2e-3,Identities 46/69(67%)) и представителя семейства энтерококков.Откуда появились прокариоты я так и не поняла, но успокаивает то что процент идентичности не превышает 40%.
Tblastx я не дождалась, он не смог ничего найти на протяжении 4 минут.Попробовав изменить параметры и ничего не получив поняла что занятие бесполезно для данной последовательности.
Так blastn подходит для поиска гомологов некодирующих генов,а megablast работает быстрее и подходит для картирование на геном и поиска почти идентичных последовательностей.Blastx чаще всегог используется когда последовательность кодирует белок,также с его помощью можно определять аннотируемые белки.Tblast подходит для поиска непредсказанной белковой последовательности.
Задание 2
Для начала пакет blast+ был загружен на компьютер.Стоить сказать что команды будут выглядеть не совсем стандартно, так как система безопасности моего компьютера отказывалась открывать файлы "скаченные из интернета" и пришлось ее обходить.
первым делом я проиндексировала последовательность своего генома
/Users/karina/Documents/ncbi-blast-2.13.0+/bin/makeblastdb -in GCF_018350155.1_O.geoffroyi_Oge1_pat1.0_genomic.fna -dbtype nucl
Далее будет использоваться blastn так как мы ищем сходство нуклеотидной последовательности с рРНК.
/Users/karina/Documents/ncbi-blast-2.13.0+/bin/blastn -task blastn -query 16S_rRNA_ecoli.fasta -db GCF_018350155.1_O.geoffroyi_Oge1_pat1.0_genomic.fna -out bl16s.txt
/Users/karina/Documents/ncbi-blast-2.13.0+/bin/blastn -task blastn -query 23S_rRNA_ecoli.fasta -db GCF_018350155.1_O.geoffroyi_Oge1_pat1.0_genomic.fna -out bl23s.txt
Находок для 16S РНК было 8, но так как в сборке была также и митохондриальная хромосома,находка была и там,но с E-value 6 что точно не является гомологом.С удовлетворительным E-value (8e-05) была только одна последовательность из E1 хромосомы.
Находок для 23S РНК было 9,с E-value от 0,007 6 находок.С лучшим E-value равным 5e-17 была находка в той же хромосоме Е1,что и для 16S РНК.Тут ксати находка в митохондриальной с E-value 0.022,что точно сильно лучше 6.
Результаты по поиску с 16S РНК можно найти тут,а для 23S РНК тут.