Реконструкция эволюции доменной архитектуры
1. Выравнивание представителей домена Pfam белков с разной доменной архитектурой
Для работы было выбрано семейство с ID M16C_assoc(PF08367), домен, встречающийся у эукариот и бактерий около С-конца металлопротеазы М16С.
Были выбраны архитектуры:
1. Peptidase_M16, Peptidase_M16_C, M16C_assoc - 991 последовательностей, из них только 20 фрагменты
2. Peptidase_M16, Peptidase_M16_C, M16C_assoc, Peptidase_M16_C - 357 последовательностей, из них только 26 фрагменты

Рисунок 1. Полученное программой MEGA дерево (Maximum Likelihood) (Названия листьев: e (синий) - Эукариоты, b (красный) - Бактерии, 1 - архитектура №1, 2 - архитектура №2).
Как видно из полученного дерева (Рис. 1), архитектуры четко разделяются, а так же содержатся в последовательностях видов обоих таксонов. Таким образом, можно сделать вывод, что они возникли до разделения таксонов, при этом мы ничего не можем сказать об их взаимной эволюции.
2. Построение профиля подсемейства
В качестве выборки для построения профиля была выбрана клада с последовательностями A0A094AZ10, A0A094D3Y7, A0A094DGN8, A0A094F6Q5; в качестве подсемейства: все предстваители e2, кроме A0A024TAG9 и A0A075AR14, так как подереву они слишком отстоят от остальных представителей e2.
По выравниванию выборки был построен профиль пакетом HMM. По построенному и откаллиброванному профилю был произведен поиск по всем последовательностям uniprot имеющим домен PF08367. Результаты и анализ приведены в таблице excel.
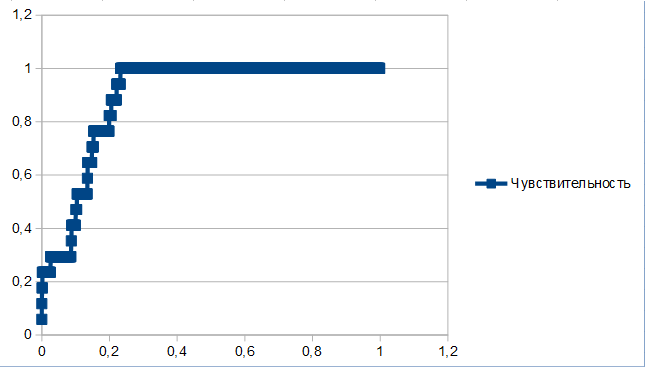
Рисунок 2. ROC кривая.
Порог e-value: 3E-26
Таблица 1. Таблица с результатами.
| На самом деле | Принадлежит семейству | Не принадлежит | Сумма |
| Выше порога по профилю | 17 | 294 | 311 |
| Ниже порога | 0 | 1466 | 1466 |
| Сумма | 17 | 1760 | 1777 |
