Поиск сигналов. Теория
1. Биологическая роль определенного транскрипционного фактора в бактерии.
С помощью сервера MEME был найден мотив связывания. Лого приведено по синей ссылке. E-value: 1.9e-020. PWM.
С помощью сервера TOMTOM был найден похожий мотив по базе RegTransBase. ТФ называется "Putative iron-sulfur cluster assembly transcriptional regulator IscR" (Транскрипционный регулятор сборки пируват железо-серного кластера IscR) с именем выравнивания "IscR_Rhodospirillales_Sphingomonadales". E-value = 3.03. По RegTransBase PWM для найденного мотива выглядит так. E-value больший единицы позволяет заключить, что скорее всего находка лишь отдаленно напоминает искомый мотив, что и видно из сравнения лого мотивов.
C помощью программы FIMO нашел мотив в геноме бактерии. Выдача MEME. Мотив нужно искать только в upstream region, так как именно там вероятность найти мотив максимальна. Если бы мы искали мотив не только в этих областях, скорее всего, мы бы нашли много лишнего. В таблице 1 приведены лучшие находки (порог E-value = 10E-8).
Таблица 1. Лучшие находки.
| ID белка | Цепь | Координаты (относительно гена) | Координаты гена | Координаты мотива в геноме | p-value | Совпавший участок |
|---|---|---|---|---|---|---|
| AAC75480 | + | 45..59 | 2544752..2545609 | 2544797..2544811 | 2.36E-09 | GGAATCATATATCCC |
| AAC75481 | - | 105..119 | 2545773..2546669 | 2545878..2545892 | 2.36E-09 | GGAATCATATATCCC |
| AAT48160 | + | 49..63 | 3157650..3158576 | 3157699..3157713 | 1.66E-08 | GAAATCATTTATTCC |
| AAC75993 | - | 86..100 | 3098558..3099565 | 3098644..3098658 | 1.66E-08 | GGAATAATTTATTCC |
| AAC74891 | + | 147..161 | 1905688..1906254 | 1905835..1905849 | 2.48E-08 | GAAATAATATATTCC |
Ниже (в таблице 2) приведены функции белков, синтезируемых с генов, в upstream участках которых находятся сигналы.
Таблица 3. Функции белков, синтезируемых с генов, в upstream участках которых находятся сигналы.
| AAC75480 | MurR — Репрессор гена murPQ. Репрессирует экспрессию оперона murPQ, ответственного за синтез и деградацию N-ацетилмурамной кислоты. |
|---|---|
| AAC75481 | MurQ — Эстераза N-ацетил-6-фосфат-мурамной кислоты. |
| AAT48160 | Функция неизвестна |
| AAC75993 | Функция неизвестна |
| AAC74891 | Белок внутренней части плазматической мембраны. Предполагается, что функционирует как магниевый насос. |
2. Метилирование
Были рассмотренны две находки с высокими p-value: mot1.fa, mot2.fa. Программа fuzznuc пакета EMBOSS предназначена для поиска паттернов в заданной последовательности. Эта программа использовалась для поиска сайтов метилирования, пересекающихся с найденными FIMO в Задании 1 мотивами (были взяты сами мотивы и участки ± 50 нуклеотидов по бокам от них, чтобы не упустить сайты, пересекающиеся с мотивом не полностью). В участках искались сайты из файла MT_sites.txt, содержащего сайты метилирования:
fuzznuc -sequence motX.fa -pattern @MT_sites.txt -outfile fuzznucX.out
В результате были получены файлы: fuzznuc1.out, fuzznuc2.out.
Таблица 3. Число находок паттернов.
| Идентификатор | Число находок |
|---|---|
| AAC75480 | 59 |
| AAT48160 | 82 |
Число находок сайтов метилирования впечатляет, однако большинство из них не пересекаются с мотивом, а те, которые пересекаются, как правило содержат много N.
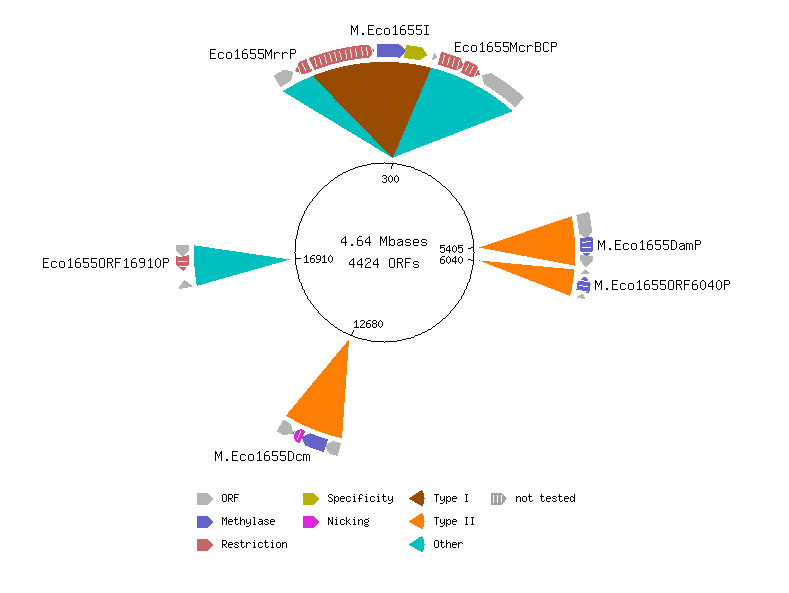
Рисунок 1. Выдача REBASE.
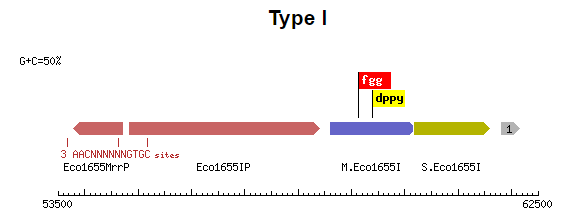
Рисунок 2. Выдача REBASE.
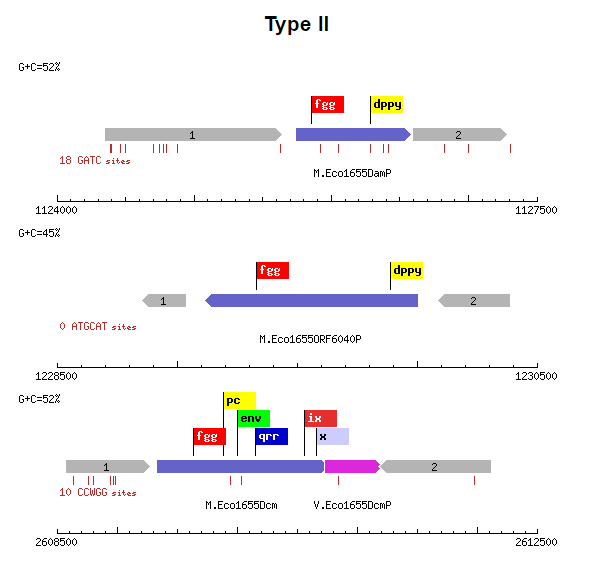
Рисунок 3. Выдача REBASE.
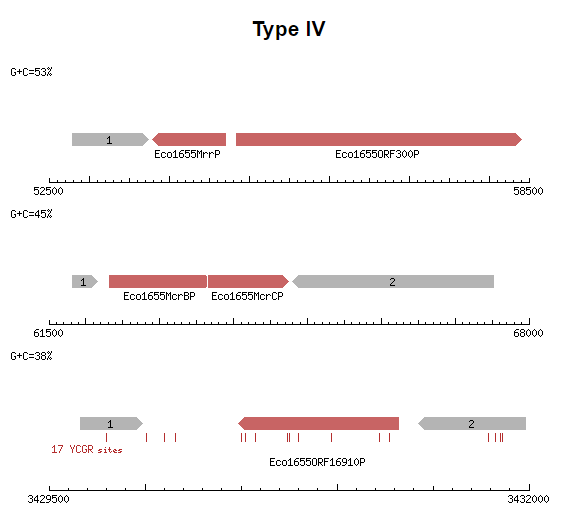
Рисунок 4. Выдача REBASE.
Ни один из паттернов метилтрансфераз данной бактерии не пересекается с обоими мотивами, так что мы можем вполне уверенно утвержать, что метилирование не влияет на связывание ТФ со своим сайтом.
