A- и В- формы ДНК. Структура РНК.
задание №1. Построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
С помощью Рutty в папке Term3 создадим директорию Practice2:
mkdir Term3/Practice2
Введем следующие команды, чтобы указать путь к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
С помощью программы fiber пакета 3DNA построим A-, B- и Z-формы дуплекса ДНК,
последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc".
Для ввода последовательности ДНК используем клавиатуру.
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb
Получены файлы
gatc-a.pdb,
gatc-b.pdb,
gatc-z.pdb.
Задание №2. Средства RasMol для работы со структурами нуклеиновых кислот.
Упражнение 1.
Научиться выделять разные атомы и химические группировки, используя предопределенные множества RasMol.
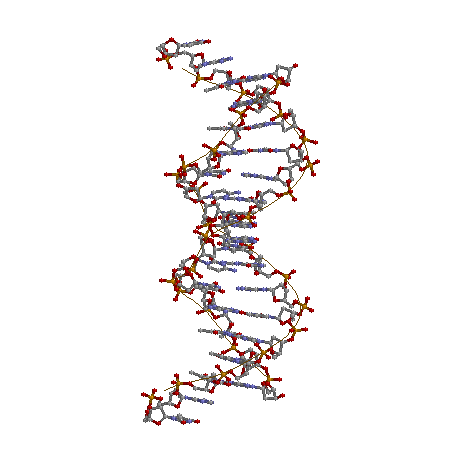
- сахарофосфатный остов ДНК (А-форма)
|
 Рис.1 Рис.1 |
|
|
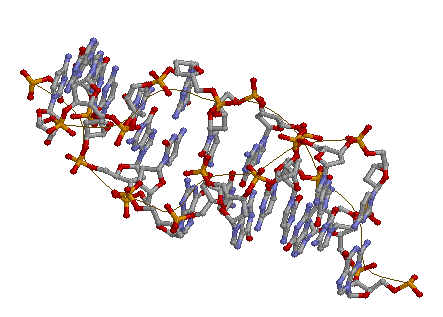 Рис.2 Рис.2 |
|
|
 Рис.3 Рис.3 |
- атом N7 во всех гуанинах и только в первом по последовательности (красный)
(B-форма)
|
 Рис.4 Рис.4 |
Упражнение 2.
Получить файлы PDB ДНК- и РНК-содержащих структур.
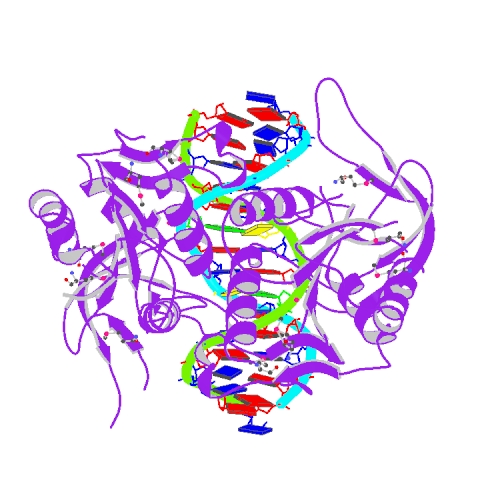
1dfm , endonuclease complexed with DNA
|
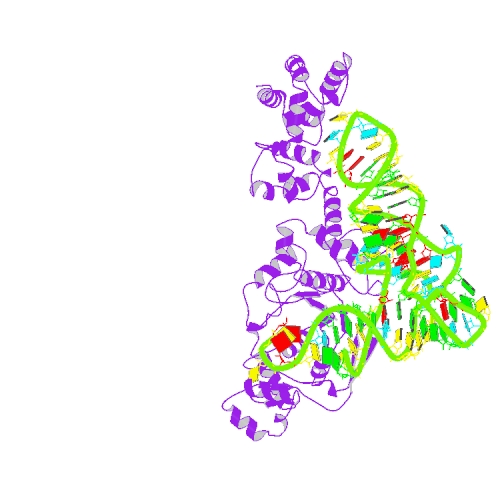
2cv0 , Glutamyl-tRNA synthetase in complex with tRNA(Glu) and L-glutamate
|
Упражнение 3.
Проверить заданные структуры ДНК и РНК на наличие разрывов.
Сохраните координаты атомов только ДНК и РНК в отдельных файлах для дальнейшей работы.
Проверено. Разрывов нет.
Задание №3. Сравнение структур 3-х форм ДНК с помощью средств RasMol.
Упражнение 1.
Откроем файл gatc-b.pdb (B-форма ДНК) из задания 1.
Визуально определим большую и малую бороздку.
Выберем заданное азотистое основание (ADE 10).
Определим, какие атомы основания явно обращены в сторону большой бороздки, а какие - в сторону малой.
С помощью ChemSketch получим изображение основания, выделим красным цветом атомы,
смотрящие в сторону большой бороздки, синим - в сторону малой.
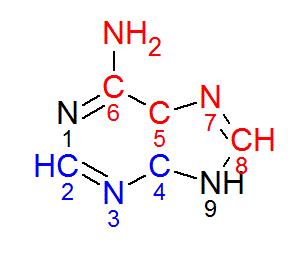
- В сторону большой бороздки обращены атомы a10.N6, a10.C5, a10.C6, a10.N7, a10.C8.
- В сторону малой бороздки обращены атомы a10.C2, a10.N3, a10.C4.
- Остальные атомы основания a10.N1 и a10.N9 лежат на условной линии границы малой и большой бороздок.
В A-форме ДНК (gatc-a.pdb) атомы распределятся с точностью наоборот:
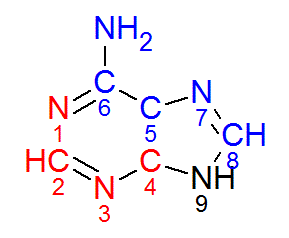
- В сторону большой бороздки обращены атомы a10.N1, a10.C2, a10.N3, a10.C4.
- В сторону малой бороздки обращены атомы a10.N6, a10.C5, a10.C6, a10.N7, a10.C8.
- Aтом a10.N9 лежит на условной линии границы малой и большой бороздок.
В Z-форме ДНК (gatc-z.pdb) Аденин не представлен.
Упражнение 2.
Сравнить основные спиральные параметры разных форм ДНК.
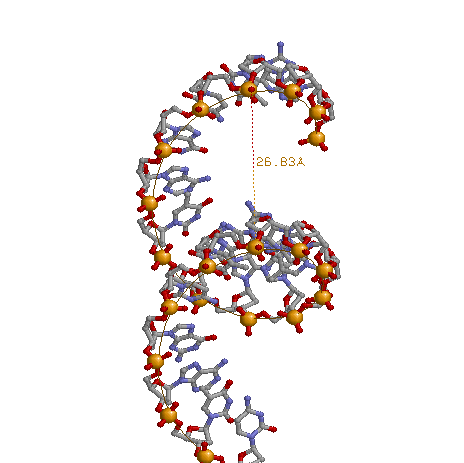
(например расстояние между витками)
| |
A-форма |
B-форма |
Z-форма |
| Тип спирали (правая или левая) |
правая |
левая |
левая |
| Шаг спирали (A) |
26.83 |
33.75 |
43.50 |
| Число оснований на виток |
11 |
10 |
12 |
| Ширина большой бороздки |
16.81 |
17.21 |
18.30 |
| Ширина малой бороздки |
7.98 |
11.69 |
9.86 |
Упражнение 3.
Сравнить торсионные углы в структурах А- и В-форм.
| угол |
α |
β |
γ |
δ |
ε |
ξ |
χ |
| A-форма (презентация) |
-62 |
173 |
52 |
88 или 3 |
178 |
-50 |
-160 |
| A-форма (RasMol) |
-51.71 |
174.80 |
41.72 |
79.04 |
-147.75 |
-75.11 |
-157,22 |
| B-форма (презентация) |
-63 |
171 |
54 |
123 или 131 |
155 |
-90 |
-117 |
| B-форма (RasMol) |
-29.91 |
136.33 |
31.16 |
143.34 |
-140.78 |
-160.49 |
-98.03 |
Задание №4. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Для файлов из Упражнения 1. gatc-a.pdb, gatc-b.pdb и gatc-z.pdb выполним команду:
find_pair -t gatc-x.pdb stdout | analyze
В результате для каждой структуры создан ряд файлов с описанием разных её параметров.
В файлах
gatc-a.out ,
gatc-b.out ,
gatc-z.out
можно найти значения торсионных углов.
Вынесем полезную информацию в файл gatc_torsion.txt.
Из сравнения торсионных углов в структурах A-, B- и Z-форм ДНК
видно, что в наибольшей степени различаются значения следующих углов:
в A- и B-формах - δ и χ
в A- и Z-формах - α
в B- и Z-формах - α, ξ и χ.
Так как пакет 3DNA работает только со старым форматом .pdb, используем программу remediator для перевода
файлов 1DFM.pdb и 2CV0.pdb в старый формат:
remediator --old "XXXX.pdb" > "XXXX_old.pdb"
И применим команды find_pair и analyze.
Данные о торсионных углах в структуре тРНК 2CV0_old.pdb
вынесем в отдельный файл 2cv0_torsion.txt .
Данная структура больше всего похожа на A-форму ДНК, если рассматривать значения торсионных углов.
Исследование т-РНК
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между
азотистыми основаниями (файлы
2cv0_pairs.txt и
2cv0_old.out ).
Найдем стебли в последовательности т-РНК.
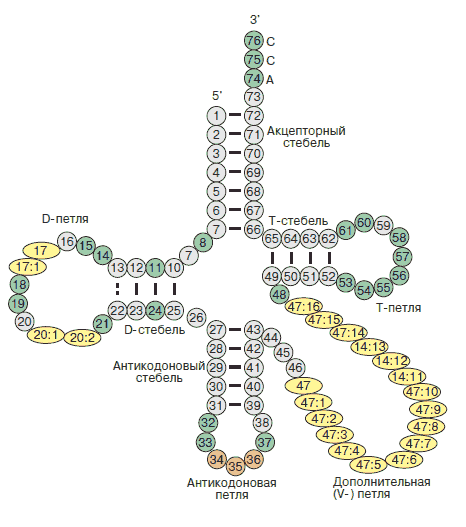
В соответствии с полученными данными:
- акцепторный стебель состоит из участка 501 - 507 и комплементарного ему участка 566 - 572 (на рисунке окрашен в красный);
- Т-стебель состоит из участка 549 - 553 и комплементарного ему участка 561 - 565 (зеленый);
- D-стебель состоит из участка 510 - 513 и комплементарного ему участка 522 - 525 (синий);
- антикодоновый стебель состоит из участка 526 - 532 и комплементарного ему участка 538 - 544 (оранжевый).
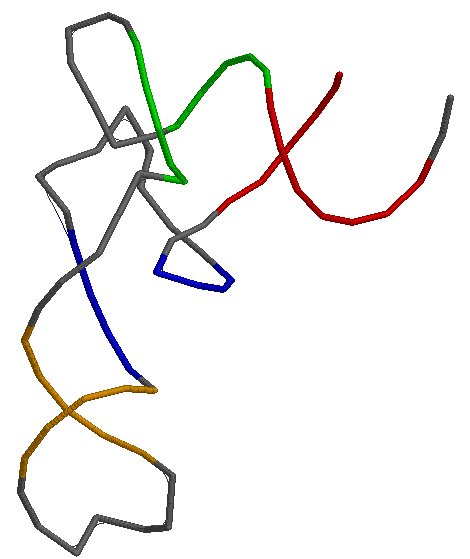
Как видно из все того же файла 2cv0_pairs.txt в структуре РНК
содержится 8 неканонических пар:
| неканонические пары нуклеотидов |
пример взаимодействия A538--C532 |
- G502--U571
- U554--A558
- U555--G529
- A538--C532
- A544--G526
- U513--G522
- A514--U508
- G515--C548
|
|
Благодаря все тому же файлу 2cv0_pairs.txt заметим, что в молекуле есть 5 пар нуклеотидов, не вошедшие в стебли.
Внимательно рассмотрев структуру РНК можно обнаружить, что пары нуклеотидов 554-558 и 555-518 дополнительно стабилизируют Т-петлю,
а пары 514-508, 515-548 и 519-556 образуются на пересечении цепи РНК в пространстве.
Научиться находить возможные стекинг-взаимодействия.
Сперва с помощью файла stecking.pdb и файла 2cv0_pairs.txt
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/UC 1.18( 0.12) 0.00( 0.00) 0.00( 0.00) 0.91( 0.00) 2.09( 0.12)
2 GC/GU 7.14( 4.30) 0.00( 0.00) 0.00( 0.00) 6.63( 4.10) 13.76( 8.40)
3 CC/GG 0.16( 0.01) 0.00( 0.00) 0.30( 0.00) 2.78( 1.27) 3.24( 1.28)
4 CC/GG 0.00( 0.00) 0.00( 0.00) 0.16( 0.00) 3.82( 2.48) 3.98( 2.48)
5 CC/GG 0.18( 0.01) 0.00( 0.00) 0.31( 0.00) 2.91( 1.49) 3.40( 1.49)
6 CA/UG 0.05( 0.00) 0.00( 0.00) 2.00( 0.48) 0.56( 0.14) 2.62( 0.62)
7 AG/CU 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.91( 1.00) 2.91( 1.00)
8 GG/CC 2.16( 0.71) 0.00( 0.00) 0.24( 0.00) 0.62( 0.19) 3.02( 0.89)
9 GG/CC 4.21( 2.85) 0.00( 0.00) 0.01( 0.00) 0.52( 0.00) 4.74( 2.85)
10 GG/CC 3.70( 2.34) 0.00( 0.00) 0.22( 0.00) 0.00( 0.00) 3.92( 2.34)
11 GG/CC 4.13( 2.67) 0.00( 0.00) 0.48( 0.00) 0.00( 0.00) 4.61( 2.67)
12 GU/AC 5.77( 2.83) 0.00( 0.00) 0.00( 0.00) 1.12( 0.02) 6.89( 2.85)
13 UU/GA 5.05( 2.64) 0.00( 0.00) 0.00( 0.00) 3.74( 1.24) 8.78( 3.88)
14 UA/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 AG/CC 3.93( 2.16) 0.00( 0.00) 0.00( 0.00) 5.20( 2.36) 9.13( 4.52)
16 GG/CC 2.92( 1.44) 0.00( 0.00) 0.51( 0.00) 0.38( 0.00) 3.81( 1.44)
17 GC/GC 6.19( 3.17) 0.00( 0.00) 0.00( 0.00) 6.51( 3.37) 12.69( 6.53)
18 CC/GG 0.20( 0.01) 0.00( 0.00) 0.69( 0.00) 2.95( 1.47) 3.84( 1.48)
19 CG/CG 0.00( 0.00) 0.00( 0.00) 5.02( 2.28) 0.00( 0.00) 5.02( 2.28)
20 GA/GC 1.66( 0.00) 0.00( 0.00) 0.00( 0.00) 4.93( 2.24) 6.59( 2.24)
21 AG/CG 0.00( 0.00) 0.00( 0.00) 0.47( 0.00) 1.50( 0.92) 1.97( 0.92)
22 GU/AC 5.06( 2.37) 0.00( 0.00) 0.00( 0.00) 3.41( 2.04) 8.47( 4.41)
23 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 3.87( 2.43) 3.87( 2.43)
24 CU/GG 1.30( 0.05) 0.00( 0.00) 0.00( 0.00) 2.71( 1.19) 4.01( 1.24)
25 UA/UG 0.00( 0.00) 2.33( 0.10) 5.78( 4.47) 0.00( 0.00) 8.11( 4.57)
26 AG/CU 3.49( 1.30) 0.00( 0.00) 0.04( 0.00) 0.00( 0.00) 3.53( 1.30)
27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
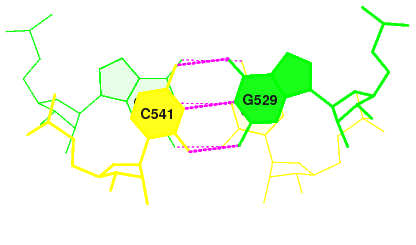
найдем, номера нуклеотидов с наибольшей суммой перекрывания.
Это нуклеотиды 501, 502, 571, 572 и 540, 530 ,541, 523.
С помощью команды
ex_str -17 stacking.pdb step17.pdb
выведем координаты взаимодействующих атомов в отдельные файлы step17.pdb, step1.pdb.
Построим изображение с помощью команды
stack2img -cdolt step8.pdb step8.ps
Конвертируем картинку в подходящий формат...
На страницу 3 семестра
© Aleshin Vasily
 Рис.1
Рис.1  Рис.2
Рис.2  Рис.3
Рис.3  Рис.4
Рис.4  Рис.1
Рис.1  Рис.2
Рис.2  Рис.3
Рис.3  Рис.4
Рис.4