Филогенетические деревья. Занятие 4.
Задание 1. Построение дерева по нуклеотидным последовательностям.
С помощью SRS отыщем геномы бактерий по белку "RL2_*****".
Затем найдем 16S rRNA в геноме.
Например командой
seqret EMBL:AP008971 -sask
Использованные для этого данные занесем в таблицу:
| бактерия |
EMBL AC |
координаты |
цепь |
| BACAN |
AE016879 |
9335-10841 |
normal |
| BACSU |
AL009126 |
9810-11364 |
normal |
| CLOB1 |
CP000726 |
9282-10783 |
normal |
| CLOTE |
AE015927 |
41801-43309 |
complement |
| ENTFA |
AE016830 |
248466-249987 |
normal |
| FINM2 |
AP008971 |
197837-199361 |
normal |
| GEOKA |
BA000043 |
10421-11973 |
normal |
Полученные fasta-файлы с последовательностями 16S РНК из геномов бактерий
были объединены в один и выровнены в Jalview с помощью QlustalO.
Затем, сохраненный новый файл был открыт в MEGA, проанализирован c Neibour-joining как некодирующее нуклеотидное
выравнивание. Использован Бутстреп.
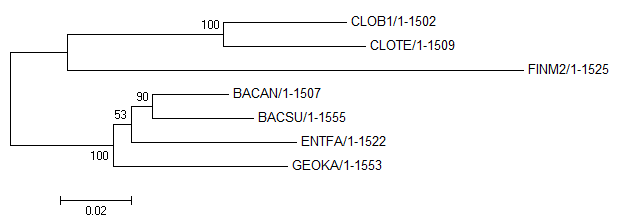 |
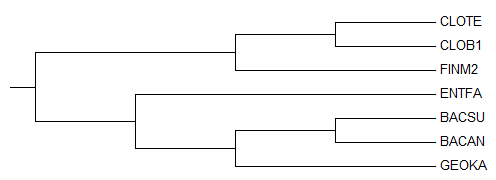 |
| Неукорененное дерево, полученное по 16S-РНК |
Исходное дерево, принятое за образец |
По картинкам можно заметить, что метод "neighbor-joining" привел неверное филогенетичеткое дерево
(отсутствует ветвь (BACSU,BACAN,GEOKA vs CLOTE,CLOB1,FINM2,ENTFA), вместо нее есть ветвь (BACSU,BACAN,ENTFA vs CLOTE,CLOB1,FINM2,GEOKA).
Это ровно столько же, сколько дало аналогичное выравнивание по белкам, поэтому, к сожалению, метод в данном случае не
проявил свое превосходство.
Задание 2.
Для поиска ортологов и паралогов используем файл с белками организмов на диске P и blastp.
Сперва проиндексируем файл:
makeblastdb -in proteo.fasta -out prot -dbtype prot
Затем по сформированной таким образом базе прогоним blastp, выравнивая белки с заготовленной заранее fasta белка CLPX_BACSU.
blastp -query CLPX.fasta -db prot -out prot_CLPX.txt -evalue 0.001
В результате получим набор белков (prot_CLPX.txt без обработки).
Выберем из них только белки семи описываемых бактерий,
выравнивания приводить ни к чему.
Используя ID белков, с помощью Jalview выровняем их. Выравнивание точно показывает
консервативные и неконсервативные области.
По данному выравниванию построим дерево в MEGA. Используем, например, Minimum-evolution.
Приведем несколько пар ортологов и паралогов, считая, что дерево получено правильно, как это сказано в задании.
Указание. Два гомологичных белка будем называть ортологами, если они а) из разных организмов; б) разделение
их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из
одного организма будем называть паралогами.
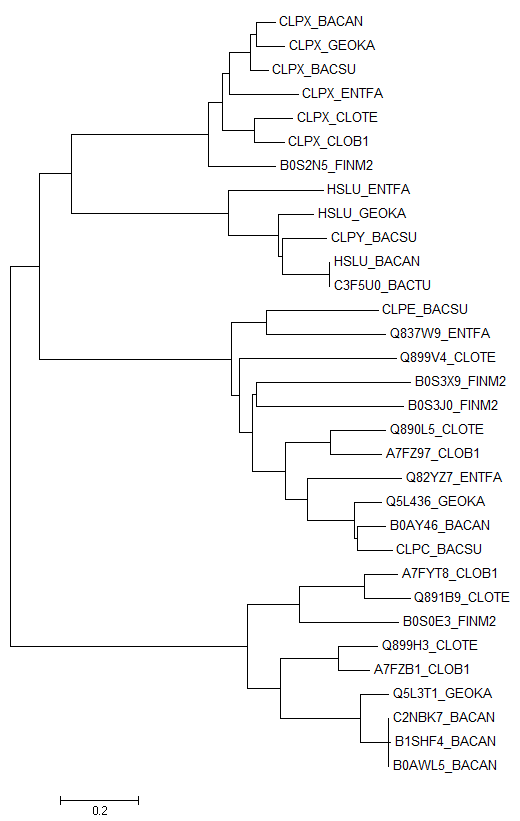 |
Паралоги: CLPX_BACSU & CLPY_BACSU; HSLU_ENTFA & CLPY_ENTFA; HSLU_GEOKA & CLPY_GEOKA; B0S3J0_FINM2 & B0S3X9_FINM2;...
Ортологи: CLPX_BACAN & CLPX_GEOKA; CLPX_CLOTE & CLPX_CLOB1; CLPX_BACSU & CLPX_BACAN; HSLU_ENTFA & HSLU_GEOKA;...
|
|
На страницу 4 семестра
© Aleshin Vasily




