Укоренение в среднюю точку
Укоренение в среднюю точку филогенетического дерева, построенного в практикуме 2 алгоритмом Neighbour joining tree using blosum62, было сделано программой retree пакета PHYLIP. Полученное дерево было визуализировано программой MEGA. Оно совпадает с исходным деревом. Его нельзя считать правильным по тем же причинам, что указаны в практикуме 2. Изображение представлено на рисунке 1.

Рисунок 1. Филогенетическое дерево построенное алгоритмом Neighbour joining и переукорененное в среднюю точку программой retree.
Укоренение с использованием внешней группы
Деревья, построенные методом максимальной экономии ("Maximum parsimony") невозможно укоренить в среднюю точку, потому что этот метод не учитывает длины ветвей. В данном можно воспользоваться укоренением с помощью внешней группы. Для создания дерева методом максимальной экономии программой Muscle было сделано выравнивание последовательностей из практикума 2 вместе с последовательностью из E.coli, которая будет выступать в роли внешней группы. Затем, согласно инструкции, в программе MEGA было построено дерево методом максимальной экономии ("Maximum parsimony") (рисунок 2). Затем в качестве внешей группы была выбрана последовательность из E.coli. Окончательное дерево показано на рисунке 3. Его можно считать правильным, так как оно совпадает с реальным филогенетическим деревом, данных бактерий.
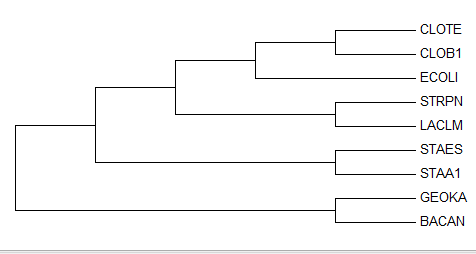
Рисунок 2. Неукорененное филогенетическое дерево построенное алгоритмом Maximum parsimony.
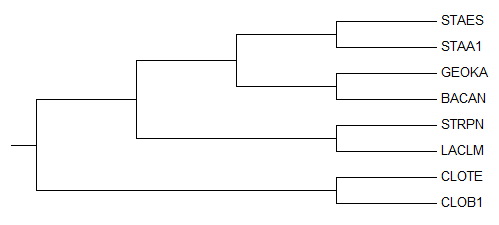
Рисунок 3. Филогенетическое дерево построенное алгоритмом Maximum parsimony, укоренненное с помощью внешней группы.
Bootstrap анализ
Для рассматриваемых последовательностей был произведен бутстрэп анализ. При выполнении данного анализа выравнивание разбивается на определенное количество реплик. Каждая реплика получается в результате удаления половины случайно выбранных столбцов из выравнивания. Удаленные столбцы заменяются копиями других случайно выбранных столбцов. Затем по полученным репликам составляется дерево, а полученные деревья сравниваются. Бутстрэп анализ был выполнен с помощью программы MEGA ("Bootstrap method" в меню "Test of Phylogeny"). Число реплик 100. Было получено два дерева "Original tree" и "Bootstrap consensus tree". Они представлены на рисунках 4 и 5. Правильное дерево показано на рисунке 6.
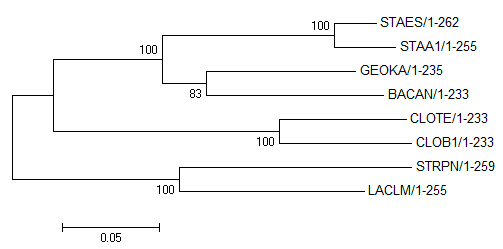
Рисунок 4. Филогенетическое дерево "Original tree" построенное с помощью Neighbour joining.
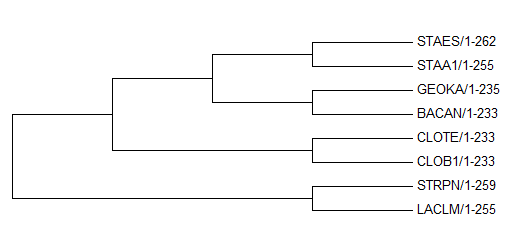
Рисунок 5. Филогенетическое дерево "Bootstrap consensus tree" построенное с помощью Neighbour joining с использованием бутстрэп анализа по 100 репликам.
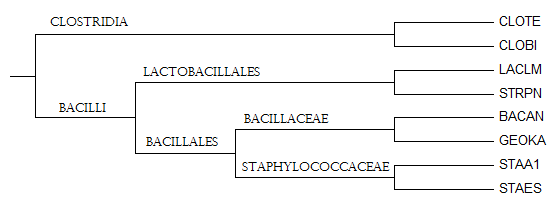
Рисунок 6. Правильное филогенетическое дерево.
Дерево полученное обычным образом совпадает с деревом, полученным с помощью бутстрэп анализа. Эти деревья неукорененные. При укоренении в ветвь Clostridia мы получим правильное дерево. Примечательно, что алгоритм Neighbour joining, который в практикуме 2 использовался для построения дерева через Jalview и давал неправильный результат, в данном случае строит правильное дерево.