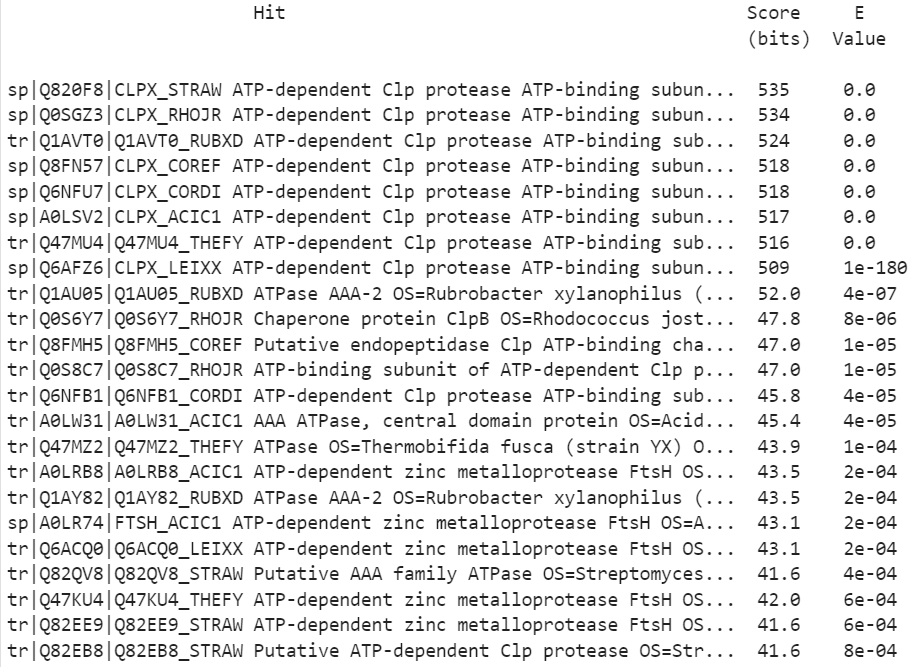
Выполним поиск гомологов АТФ-зависимой Clp протеазы среди бактерий, с которыми работали на предыдущих практикумах. Для этого перенесём себе в рабочую директорию полные протеомы отобранных бактерий, проиндексируем их и проведём поиск командой blastp, установив порог на E-value, равный 0,001. Получили следующие результаты (Табл.1):

Обсудим немного полученные находки. Среди них ожидаемо большое количество Clp протеаз. Также существенная доля находок приходится на цинковую металлопротеазу Ftsh, причём как и Clp протеаза она АТФ-зависимая. Интересно, что в находках оказались АТФазы типа ААА. Думаю, это можно связать с тем, что использованная для поиска гомологов последовательность Clp протеазы является АТФ-связывающей.
Последовательности находок поместили в один файл и подали его на ресурс NGPhylogeny.fr, где осуществили реконструкцию дерева. Конвейер включал выравнивание командой MAFFT и реконструкцию командой FastME c параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement". Был также проведён бутстреп-анализ с количеством реплик 100. Получили дерево, чья формула в Newick-формате представлена в файле. Визуализировала дерево в iTOL (Рис. 1).

Расшифровка идентификаторов. АТФ-зависимая Clp протеаза — CLPX, Q1AVTO, Q47MU4, Q0S8C7, Q6NFB1; АТФаза ААА — Q1AU05, A0LW31, Q47MZ2, Q1AY82; шаперон ClpB — Q0S6Y7; предполагаемая эндопептидаза Clp — Q8FMH5; АТФ-зависима цинковая металлопротеаза Ftsh — A0LRB8, FTSH, Q6ACQ0, Q47KU4, Q82EE9; предполагаемая АТФаза — Q82QV8; предполагаемая АТФ-зависимая Clp протеаза — Q82EB8. Расшифровка названий бактерий дана в предыдущем практикуме.
Выделим в полученном дереве ортологи и паралоги. Ортологи: CLPX_STRAW и Q47MU4, CLPX_COREF и CLPX_CORDI, A0LRB8_ACIC1 и Q47KU4_THEFY. Паралоги: Q0S8C7_RHOJR и CLPX_RHOJR, Q1AU05_RUBXD и Q1AVT0_RUBXD, Q6NFB1_CORDI и CLPX_CORDI.
В полученном дереве хорошо обособлены 2 группы: Clp протеаза и цинковая металлопротеаза Ftsh (Рис. 2):
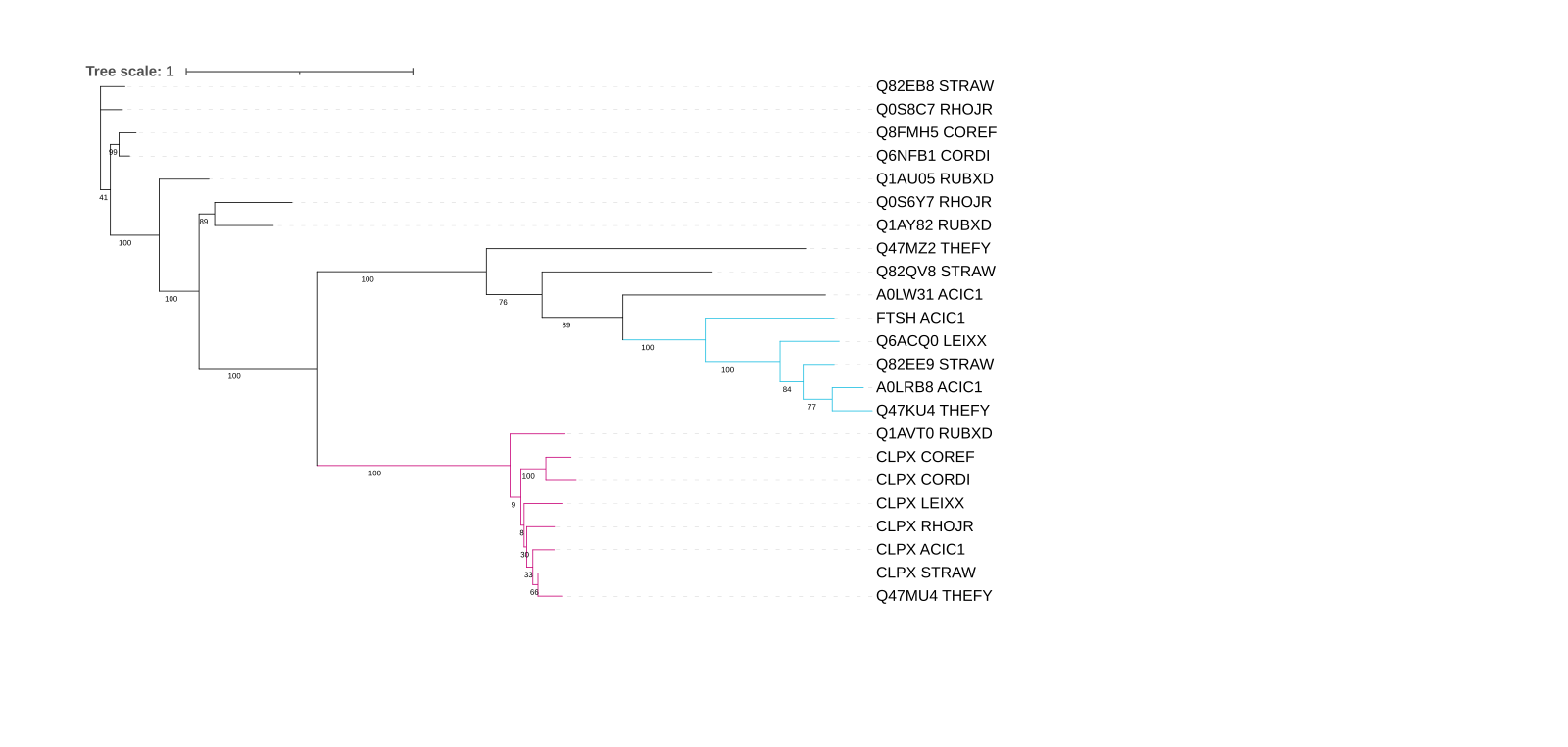
Схлопнем эти ветви (Рис. 3). В синюю ветвь (цинковая металлопротеаза — видимо, у ACIC1 произошла дупликация этого гена) войдут 5 листьев (представляюще 4 бактерии), в розовую — 8 листьев (все бактерии).

Обе схлопнутые ветви хорошо выделяют пару COREF и CORDI. Топология LEIXX, ACIC1, THEFY, STRAW в группе Clp протеазы соотвествует эталонной, но встроен лист RHOJR. Интересно, что в группе цинковой металлопротеазы топология этой группы листьев отличается от их топологии в группе Clp протеазы и эталонном дереве, но соответствует их топологии в дереве, построенному по 16S РНК.