Задание 1.
В шестом практикуме была получена консенсусная последовательность. Для выполнения задания необходимо выяснить функцию данной последовательности и организм, которому она принадлежит. Для этого нужен алгоритм blastn, потому что неизвестно, что это за последовательность и кодирует ли она белок. В качестве базы выбран Nucleotide collection (nr/nt), чтобы выяснить, есть ли запрос в аннотированных последовательностях.
Выдача BLAST (параметры: по умолчанию). Как можно заметить, процент идентичности во всех 100 находках от 90.31% до 99.03%, E-value достаточно маленький. Однако в выдачу попали и хромосомные сборки.
Все находки представляют собой последовательности, частично кодирующие белок гистон Н3. Посмотрев результат выдачи по таксономии, больше всего находок среди типа Annelida, класса Polychaeta. Для выявления более точной таксономии построила выравнивание отдельно для представителей Errantia (три находки) и отдельно для Sedentaria (четыре находки). Выравнивания в Jalview: Errantia, Sedentaria. Вышло, что процент идентичности гораздо выше для представителей первого подкласса.
Вероятно, искомая последовательность была получена из Glycera capitata, принадлежащему семейству Glyceridae, подклассу Errantia, классу Polychaeta, типу Annelida.
Задание 2.
С качестве нуклеотидной последовательности был выбран контиг из Acanthamoeba castellanii (contig48). Далее загоняю запрос в blastx (необходимо найти ген и предсказать функцию белка, поэтому сначала транслируем последовательность, а затем ищем среди гомологичных находок). База - Uniprot/Swiss-Prot, чтобы удостовериться среди аннотированных находок. Word size равен 3, чтобы увеличить чувствительность. Причём, исключаются находки, принадлежащие организму, из которого получили последовательность.
Выдача BLASTX. Процент покрытия разнится от 1% до 2%, E-value низкий, процент идентичности не превосходит 75%. Предположения такие:
- Cубъединица гамма T-комплекса белка (TCP-1-gamma), участвующего в сворачивании в третичную структуру белка во время гидролиза АТФ;
- Cубъединица альфа термосомы - молекулярный шаперон, связывает in vitro несвёрнутые полипептиды, обладает слабой АТФазной активностью;
- Cубъединица бета термосомы - молекулярный шаперон, связывает in vitro несвёрнутые полипептиды, стимулирует фолдинг белка, обладает АТФазной активностью;
- Cубъединица гамма термосомы - молекулярный шаперон, связывает in vitro несвёрнутые полипептиды, стимулирует фолдинг белка, обладает слабой АТФазной активностью;

Задание 3.
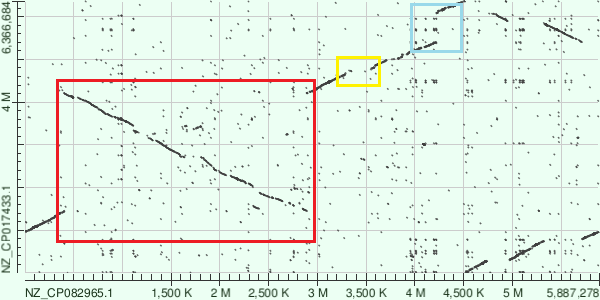
Были выбраны прокариоты с полной сборкой генома на хромосомном уровне из бета-протеобактерий: Achromobacter mucicolens, Achromobacter ruhlandii. Далее запускаем blastn (megablast - для сравнения двух близких последовательностей): query sequence - A. mucicolens, subject sequence - A. ruhlandii. Параметры по умолчанию.
В итоге, получили карту локального сходства двух последовательностей (рис.2). По dotplot`у видно, что есть возможные гомологичные участки, но их не так много, нежели неконсервативных участков.
Красными прямоугольниками помечены участки, в которых произошла инверсия (поворот на 180 градусов); жёлтыми - вставка/выпадение участка; синим - транслокация.