В данном практикуме ищем гомологи белка CLPX_ECOLI (АТФ-связанной субъединицы ClpX АТФ-зависимой протеазы Clp) среди бактерий из первого практикума.
- Для этого из директории /P/y20/term4/Proteomes копируем протеомы выбранных бактерий к себе в папку, потом в один файл:
- Делаем из них базу при помощи команды:
- Затем запускаем blastp на kodomo (выдача без названия и выравнивания приведена ниже):
cat selected_bacteria_proteomes > proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out proteomes
blastp -query CLPX_ECOLI.fasta -db proteomes -evalue 0.001 -out clpx.blastp
Score E
Sequences producing significant alignments: (Bits) Value
sp|Q820F8|CLPX_STRAW ATP-dependent Clp protease ATP-binding sub... 535 0.0
tr|Q1AVT0|Q1AVT0_RUBXD ATP-dependent Clp protease ATP-binding s... 524 0.0
sp|Q8FN57|CLPX_COREF ATP-dependent Clp protease ATP-binding sub... 518 0.0
sp|Q6NFU7|CLPX_CORDI ATP-dependent Clp protease ATP-binding sub... 518 0.0
sp|A0LSV2|CLPX_ACIC1 ATP-dependent Clp protease ATP-binding sub... 517 0.0
tr|Q47MU4|Q47MU4_THEFY ATP-dependent Clp protease ATP-binding s... 516 0.0
sp|B0RAS4|CLPX_CLAMS ATP-dependent Clp protease ATP-binding sub... 491 1e-173
tr|Q1AU05|Q1AU05_RUBXD ATPase AAA-2 OS=Rubrobacter xylanophilus... 52.0 3e-07
tr|Q8FMH5|Q8FMH5_COREF Putative endopeptidase Clp ATP-binding c... 47.0 1e-05
tr|Q6NFB1|Q6NFB1_CORDI ATP-dependent Clp protease ATP-binding s... 45.8 3e-05
tr|A0LW31|A0LW31_ACIC1 AAA ATPase, central domain protein OS=Ac... 45.4 3e-05
tr|Q47MZ2|Q47MZ2_THEFY ATPase OS=Thermobifida fusca (strain YX)... 43.9 1e-04
tr|A0LRB8|A0LRB8_ACIC1 ATP-dependent zinc metalloprotease FtsH ... 43.5 1e-04
tr|Q1AY82|Q1AY82_RUBXD ATPase AAA-2 OS=Rubrobacter xylanophilus... 43.5 1e-04
sp|A0LR74|FTSH_ACIC1 ATP-dependent zinc metalloprotease FtsH OS... 43.1 2e-04
tr|Q82QV8|Q82QV8_STRAW Putative AAA family ATPase OS=Streptomyc... 41.6 3e-04
tr|Q47KU4|Q47KU4_THEFY ATP-dependent zinc metalloprotease FtsH ... 42.0 4e-04
tr|Q82EE9|Q82EE9_STRAW ATP-dependent zinc metalloprotease FtsH ... 41.6 5e-04
tr|Q82EB8|Q82EB8_STRAW Putative ATP-dependent Clp protease OS=S... 41.6 6e-04
tr|B0RHW4|B0RHW4_CLAMS ATP-dependent zinc metalloprotease FtsH ... 41.2 7e-04
sp|Q1AV13|FTSH_RUBXD ATP-dependent zinc metalloprotease FtsH OS... 40.8 9e-04
tr|Q6NF92|Q6NF92_CORDI ATP-dependent zinc metalloprotease FtsH ... 40.8 0.001
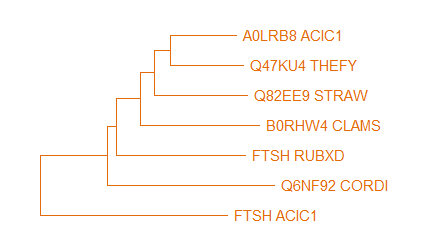
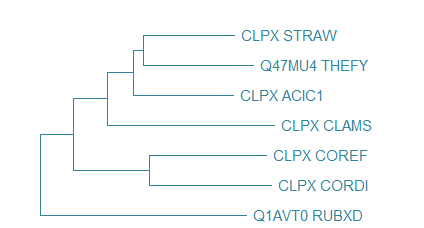
С помощью программы Jalview делаем выравнивание полученных последовательностей (алгоритм muscle с параметрами по умолчанию) и реконструируем филогению с помощью MEGAX (метод Neighbour Joining). На рис.1. приведено реконструированное дерево белков.
Скобочная формула:
(((((((((A0LRB8_ACIC1,Q47KU4_THEFY),Q82EE9_STRAW),B0RHW4_CLAMS),FTSH_RUBXD),Q6NF92_CORDI),FTSH_ACIC1),A0LW31_ACIC1),Q82QV8_STRAW),(Q1AVT0_RUBXD,((CLPX_CORDI,CLPX_COREF),(CLPX_CLAMS,(CLPX_ACIC1,(CLPX_STRAW,Q47MU4_THEFY))))),(Q47MZ2_THEFY,(Q1AY82_RUBXD,(Q1AU05_RUBXD,(Q82EB8_STRAW,(Q8FMH5_COREF,Q6NFB1_CORDI))))));
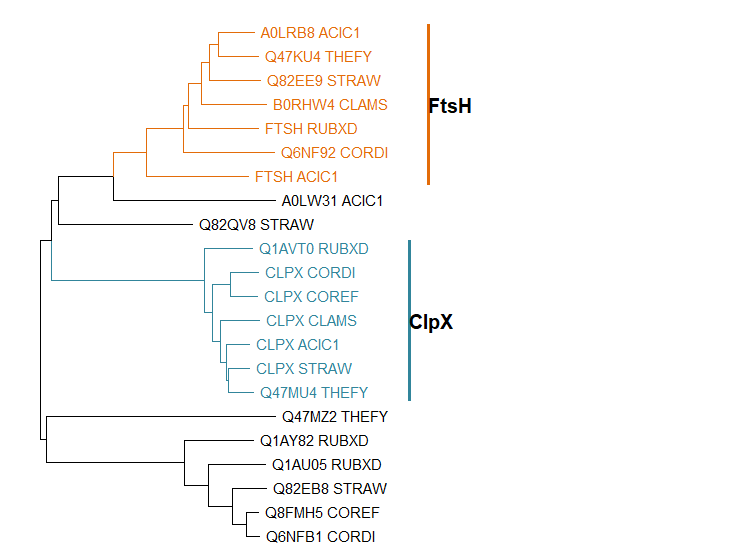
Выделим группы ортологов, покрасим их в разные цвета (см. рис. 2) и выделим по три пары ортологов и паралогов, смотря на исходное филогенетическое дерево (рис. 1).
Ортологи - гомологичные белки из разных организмов, причём, разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Паралоги - гомологичные белки из одного организма.
Ортологи:
- A0LRB8_ACIC1 и Q47KU4_THEFY
- CLPX_CORDI и CLPX_COREF
- CLPX_STRAW и Q47MU4_THEFY
Паралоги:
- FTSH_ACIC1 и CLPX_ACIC1
- Q82EE9_STRAW и CLPX_STRAW
- FTSH_RUBXD и Q1AVT0_RUBXD
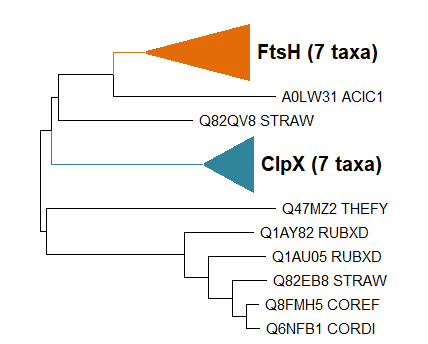
Рассмотрим ортологичные группы белков:
- FtsH (АТФ-зависимая металлопротеаза FtsH): всего 6 бактерий содержат этот белок, филогения белков не соответствует филогении бактерий, приведённой в первом практикуме. Белки FTSH_ACIC1 и A0LRB8_ACIC1 выполняют одну и ту же функцию и из одного организма Acidothermus cellulolyticus, однако первый взят из раздела swiss-prot, а второй - из TrEMBL Uniprot (рис.3).
- ClpX (АТФ-связанная субъединицы ClpX АТФ-зависимой протеазы Clp): все 7 бактерий содержат этот белок, филогения белков соответствует филогении бактерий из первого практикума (рис.4).