Пракикум 6: Секвенирование по Сэнгеру
Вводные по выполнению работы
- Файл хроматограммы: 15_F.ab1
- Программа для анализа: UGENE
- Обозначения: для гетерозиготных позиций использовались символы IUPAC
- Метод анализа: «на глаз» (визуальная оценка качества сигнала)
Результаты
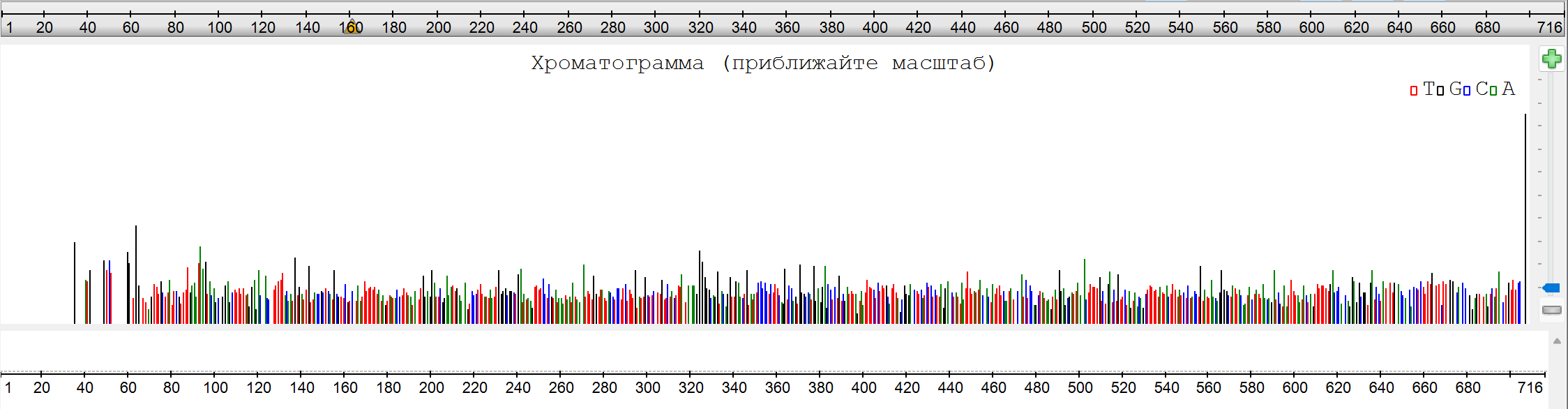
1. Общие параметры хроматограммы
- Общая длина последовательности: 716 нуклеотидов
- Качество секвенирования: хорошее
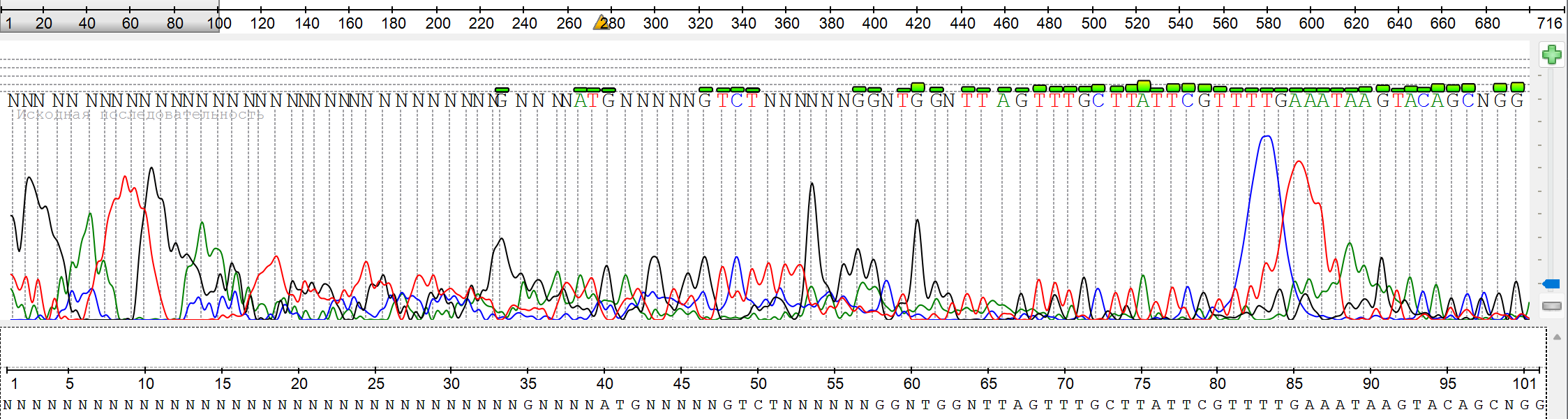
2. Трудночитаемые участки на концах
Как и ожидалось для метода Сэнгера, начальный и конечный участки имеют низкое качество:
- Позиции 1-100: высокий фон (Рис. 2)
- Позиции 611-716: сжатие пиков, наложения (Рис. 3)

3. Оценка уровня шума
- Общая характеристика: Уровень шума низкий на протяжении всей хроматограммы.
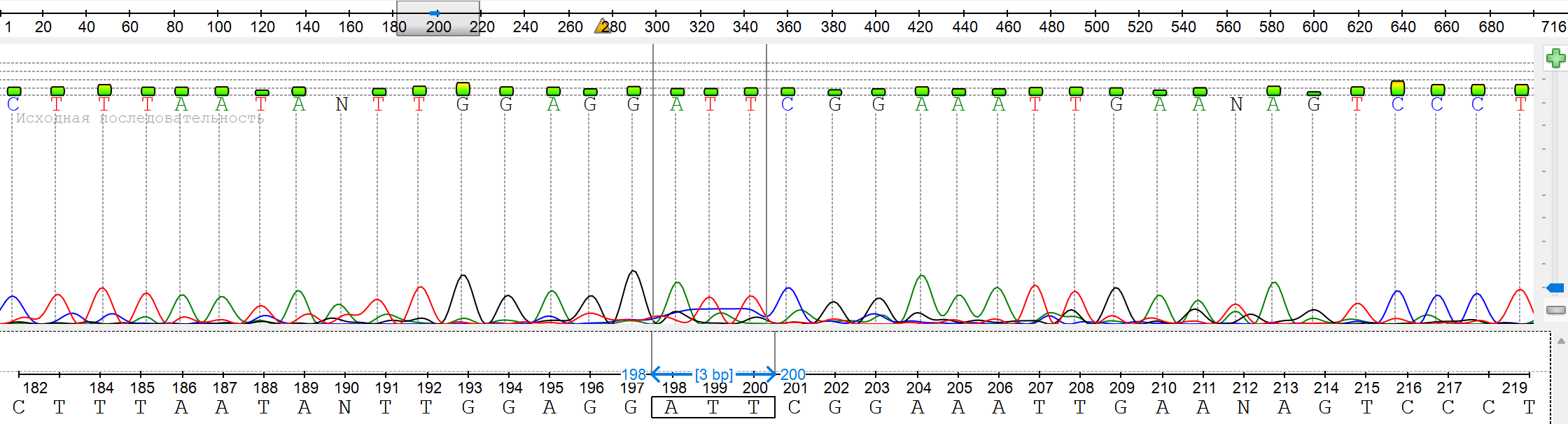
- Локальные проблемы: есть отдельные участки со сбоями (например, позиции 198-200, рис. 4)
Примеры разных ситуаций:
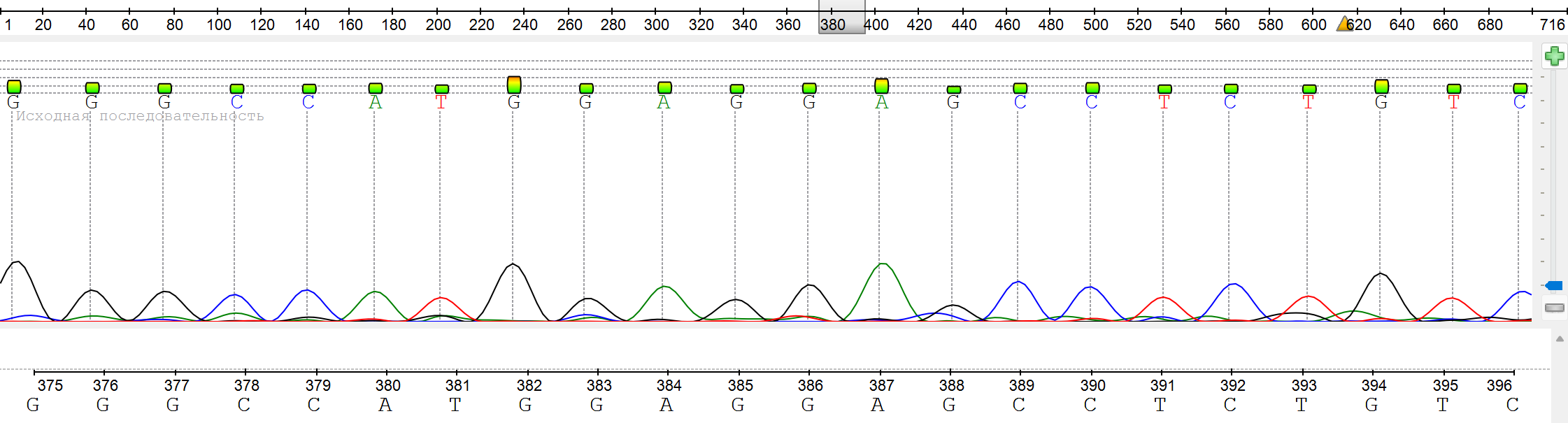
- Минимальный шум (376-395): Четкие разделенные пики (Рис. 5)
- Шум не мешает интерпретации (244-251): Фон присутствует, но не затрудняет чтение (Рис. 6)
- Шум мешает интерпретации (316-322, 369-374): Скачки фона искажают сигнал (Рис. 7 и рис. 8)
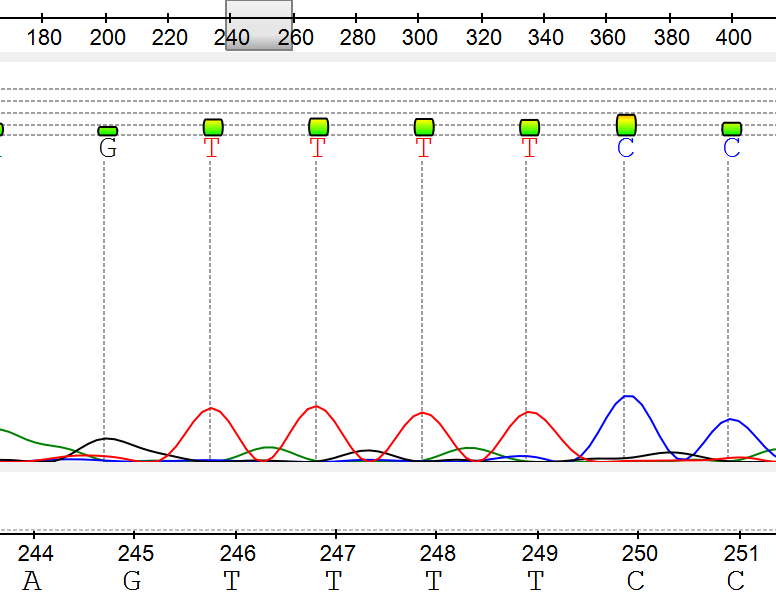
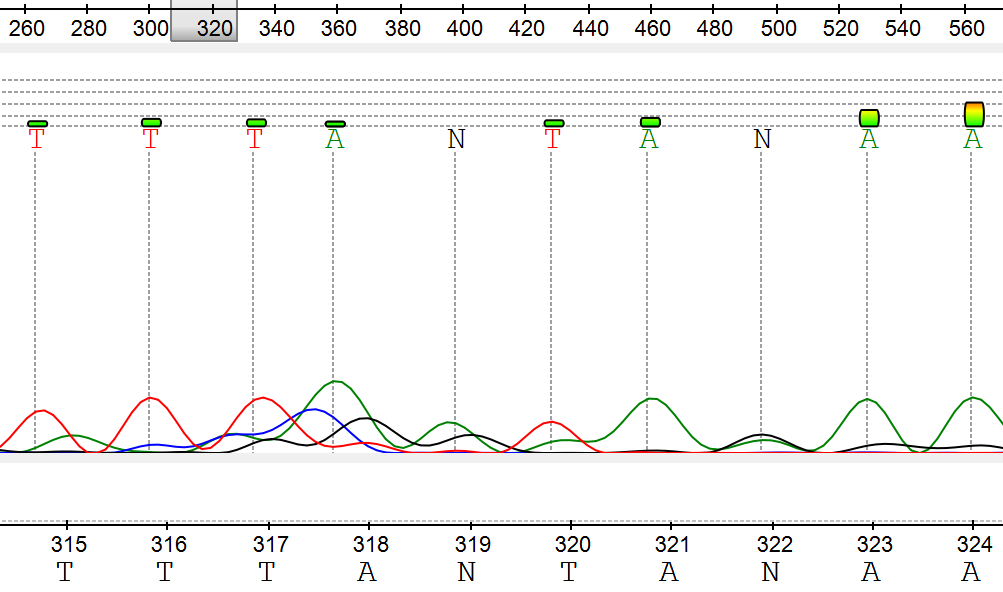
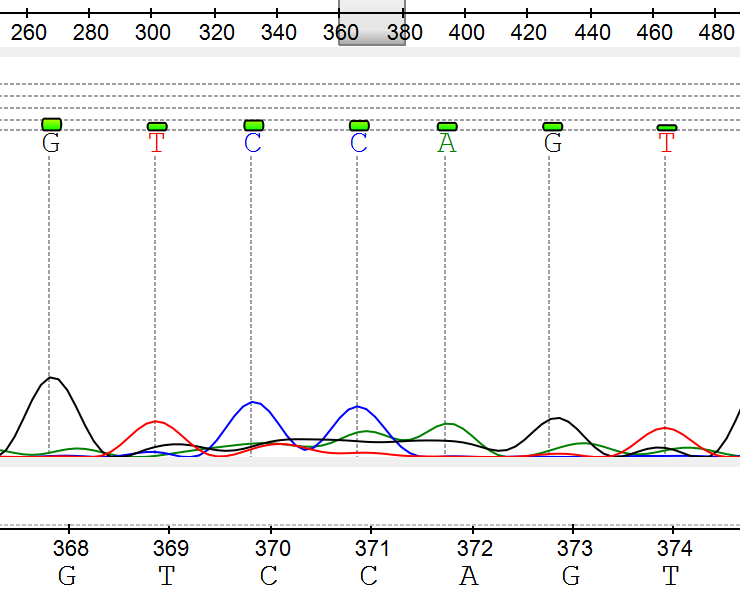
4. Выявленные полиморфизмы
В ходе анализа было идентифицировано 6 позиций, где автоматическое определение нуклеотида является ошибочным или неоднозначным. Все они представляют собой случаи гетерозиготных полиморфизмов, когда на одной и той же позиции присутствуют два разных нуклеотида.
Обозначения нуклеотидов согласно стандарту IUPAC – см. таблица 1, источник: www.hgmd.cf.ac.uk/docs/nuc_lett.html
- Позиция 160: Два ярко выраженных пика
- A и T → W (Рис. 9)
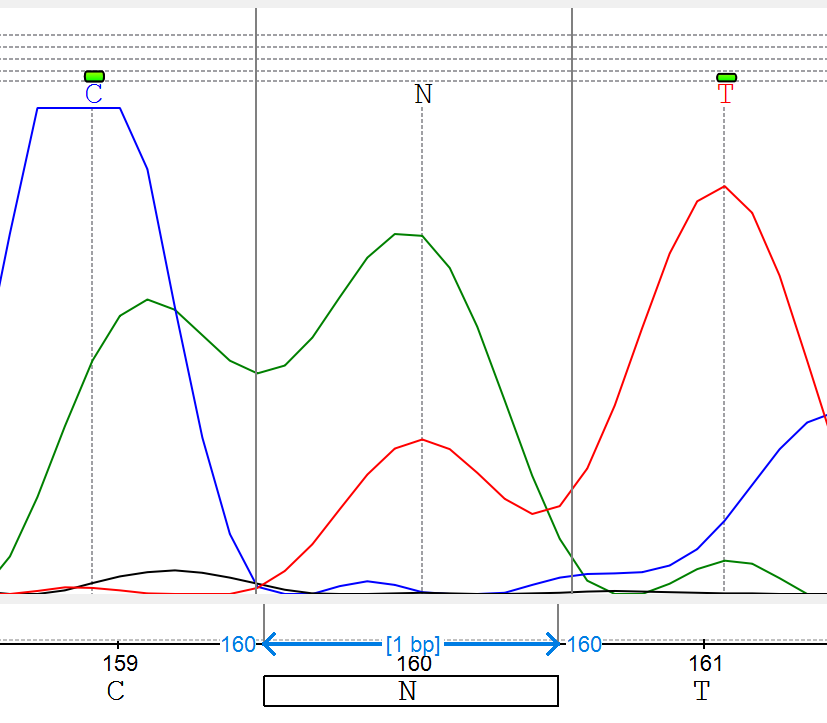
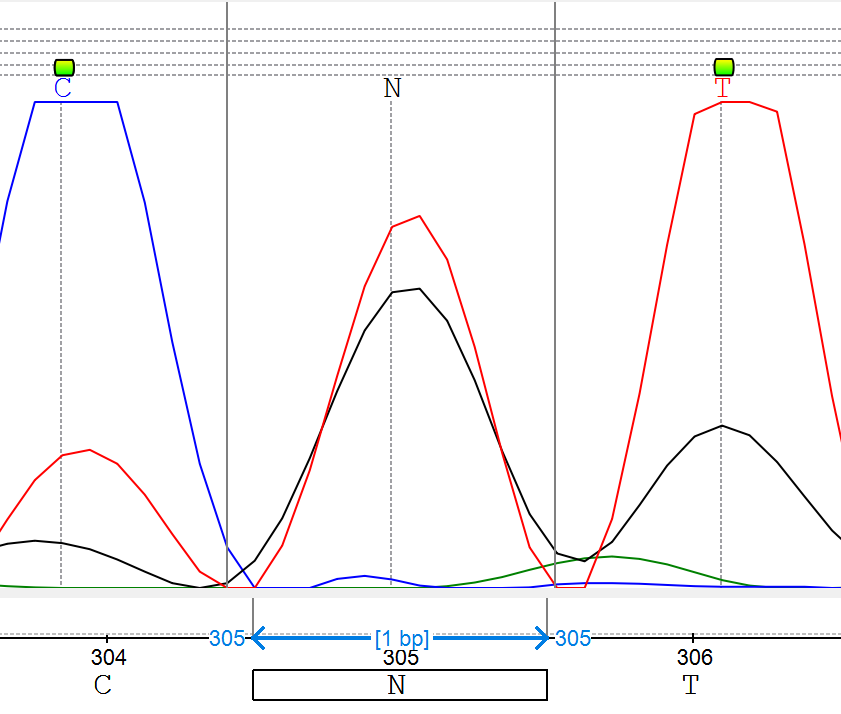
- Позиция 305: Два пика равной высоты
- T и G → K (Рис. 10)
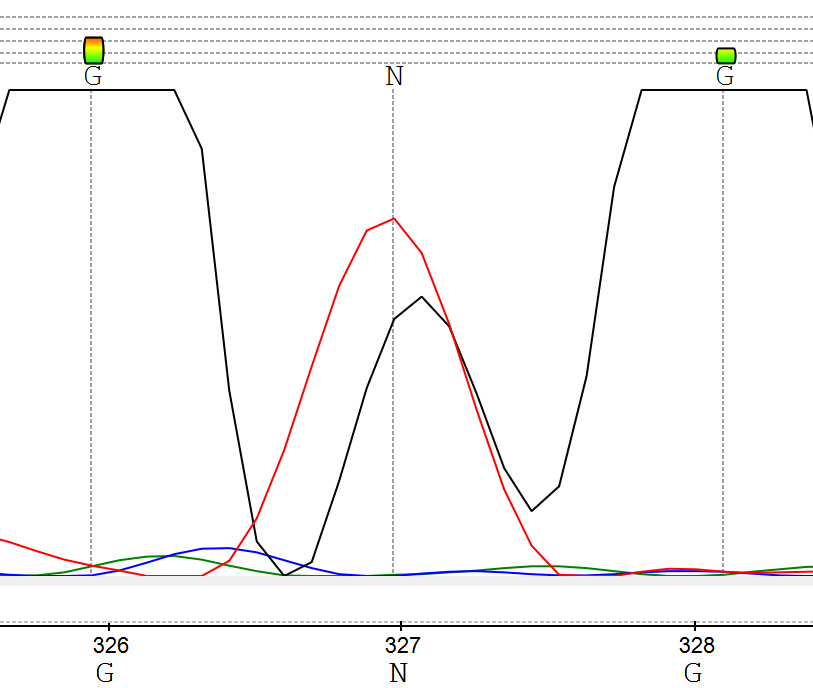
- Позиция 327: Два пика равной высоты
- T и G → K (Рис. 11)
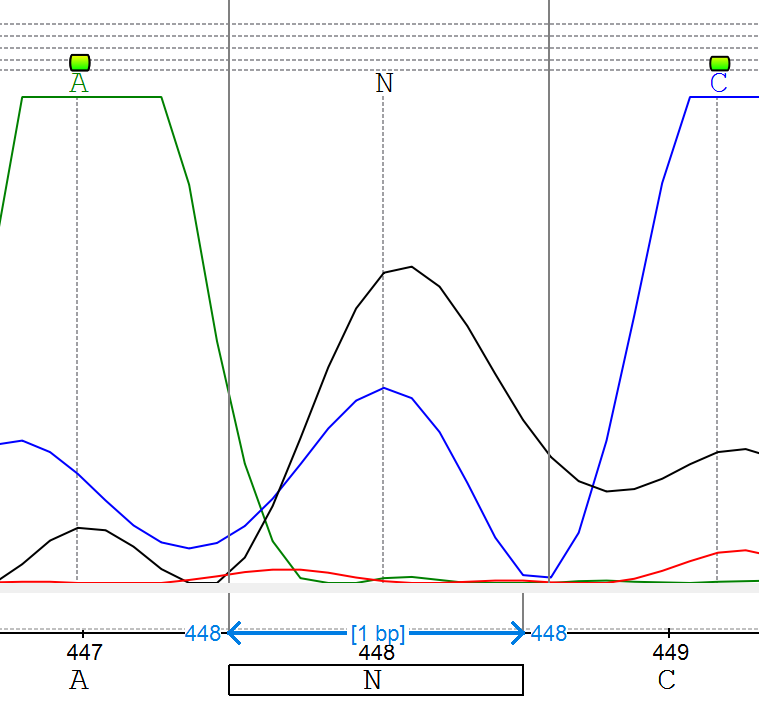
- Позиция 448: Два ярко выраженных пика
- C и G → S (Рис. 12)
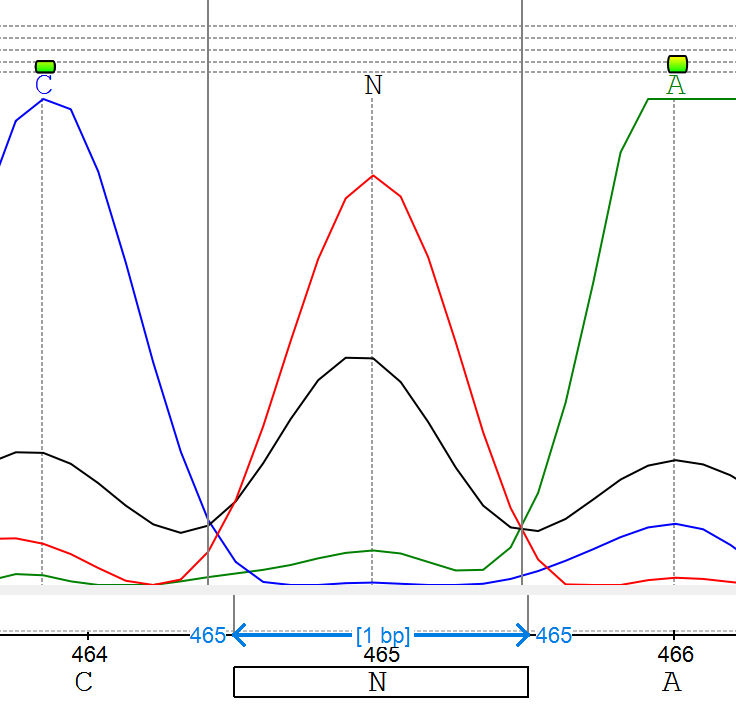
- Позиция 465: Два ярко выраженных пика
- T и G → K (Рис. 13)
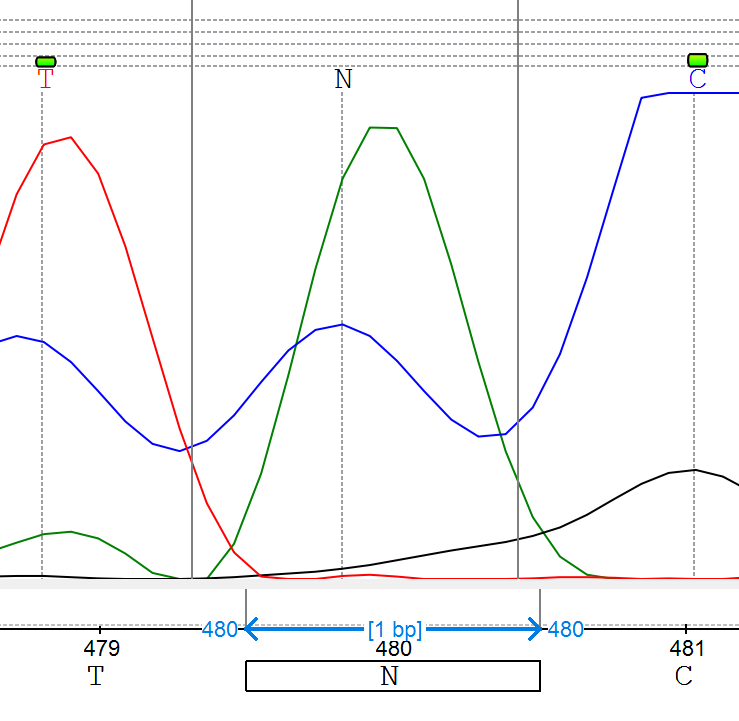
- Позиция 480: Два ярко выраженных пика
- C и A → M (Рис. 14)
| Nucleotide symbol | Full Name |
|---|---|
| A | Adenine |
| C | Cytosine |
| G | Guanine |
| T | Thymine |
| U | Uracil |
| R | Guanine / Adenine (purine) |
| Y | Cytosine / Thymine (pyrimidine) |
| K | Guanine / Thymine |
| M | Adenine / Cytosine |
| S | Guanine / Cytosine |
| W | Adenine / Thymine |
| B | Guanine / Thymine / Cytosine |
| D | Guanine / Adenine / Thymine |
| H | Adenine / Cytosine / Thymine |
| V | Guanine / Cytosine / Adenine |
| N | Adenine / Guanine / Cytosine / Thymine |
Итоги
Хроматограмма 15_F.ab1 демонстрирует высокое качество секвенирования. Выявлены характерные для метода Сэнгера проблемы на концах последовательности и несколько гетерозиготных позиций в середине. Все неоднозначности успешно разрешены с помощью символов расширенного алфавита IUPAC, что обеспечивает корректное представление генетической последовательности.