Множественное выравнивание последовательностей белков
Сравните выравнивания последовательностей разными программами.
Для выравнивания я выбрала три белка с кератолитической активностью, объединенных доменом фервидолизина (сериновая пептидаза). Кератолитические ферменты могут найти применение в дерматологии, трихологии и косметологии, очистке сточных вод, переработке отходов животноводства.
699 аминокислот, Peptidase S8
Fervidobacterium islandicum- A0A1B0VJM9_FERIS
677 аминокислот, S8 family serine peptidase
Bacteroides ovatus- A0A6A1XHX6_BACOV
619 аминокислот, Peptidase
Phocaeicola vulgatus- DW783_07685
Структуры этих белков были взяты из AlphaFold, за неимением экспериментальных или других данных о структуре. Как видно, эти белки по-разному удалены от “эталона” - обанаруженных в Fervidobacterium S8 пептидаз. Тем запутаннее должны быть результаты выравнивания. Выравнивание проводилось алгоритмами MUSCLE и MAFFT с выводом результатов в fasta. Затем результаты выравнивания были сопоставлены в VerAlign и с использованием програмы, созданной Елизаветой Плешко
VerAlign:
Видно, что MUSCLE(рис.1) предпочел выравниванию по метионину выравнивание по лизину, по какой-то причине предпочтя у третьего белка один из двух лизинов. Второй белок не участвовал в выравнивании (по метионину). Бонус в пользу MAFFT(рис2).
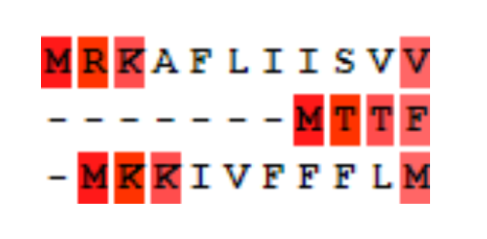
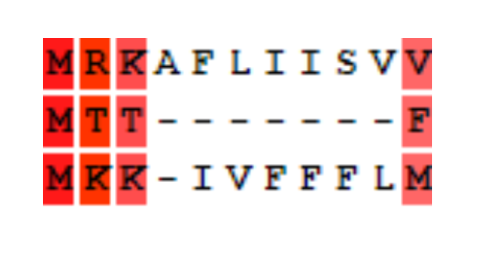
В этом случае MUSCLE(рис.3) упустил глутаминовую кислоту, совпадающую у второго и третьего белка, при этом в варианте MAFFT(рис.4) тоже сохраняются равные по длине и положению делеции. +1 в пользу MAFFT.
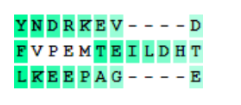
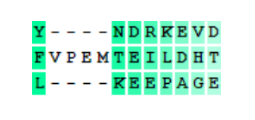
MUSCLE(рис.5) опять в проигрыше, упущено выравнивение по лейцину во всех трех белках. 3:0
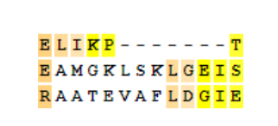
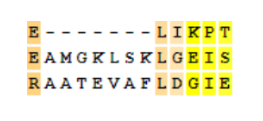
Неоднозначная ситуация.
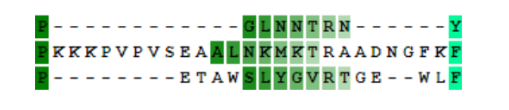
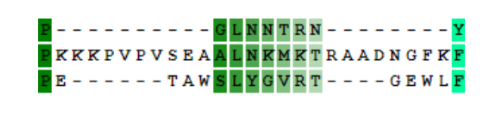
Далее по выравниванию тенденция сохраняется, MAFFT создает впечатление более “внимательного” алгоритма, находящего оптимальные решения почти в каждом неоднозначном случае. Кажется, что он дает гораздо больший приоритет выравниванию сразу по трем одинаковым аминокислотам и сохранению длины делеций/вставок, когда это возможно. Из за этих свойств MAFFT я выбрала его для разбора принципов работы. В целом видно, что белки уже очень далеко ушли друг от друга, встречается лишь несколько коротких высококонсервативных участков.
Программа Елизаветы Плешко.
Описание программы в составе 12 практикума.
Вывод в консоли: 0.51% совпадающих позиций для MAFFT и 0.52% для MUSCLE
Выравнивание по структуре
Для выравнивание по структуре применялся Pairwise Structure Alignment,куда были загружены структуры с AlphaFold. Выравнивание проводилось по методу FATCAT flexible (обоснование выбора flexible варианта). При просмотре 3D отображения выравнивания видно, что белки имеют очень схожую структуру.
Результат выравнивания, при сравнении его с результатом MAFFT, показывает практически полное отсутствие полезного результата. Выравнивание по структуре однозначно не подходит для больших и давно разошедшихся белков.
Работа MAFFT
Сначала производится попарное выравнивание и вычисляются схожие кластеры. Затем строится матрица расстояний для этих парных выравниваний и оценивается несходство между выравниваниями по балльной системе. За счет этого пары последовательностей можно упорядочить по сходству. По полученным результатам строится дерево, объединяющее все выравнивания, где узлы являются кластерами, а ветви - расстояниями между кластерами. Это дерево и используется для выравнивания, разбор этого завершающего алгоритма выходит за рамки краткости.