Практикум 7
Автор старался, но не может гарантировать отсутствие биологических ошибок.
Данный практикум посвящен рассмотрению записи о белке археи, которой в прошлом семестре посвящался
мини-обзор.
Из протеома
Saccharolobus shibatae B12
был выбран белок с ID
Q7LYV2_SACSH.
1. Выбор белка
При поиске белка для S. shibatae B12 было получено
3146 результатов
по таксономической принадлежности организма. Существование только
одной из структур (Q7LYV2)
доказано на уровне белка.
Не смотря на низкий балл по аннотации у данного белка в конечном итоге выбор был сделан в пользу него.
Поскольку опубликованных данных хватало для выполнения задания и для Q7LYV2 предсказана
3D структура.
О существовании белков
с 4/5 за аннотированность
было известно только из гомологии, не предлагалась их 3D модель. Рассмотрение структур с меньшей оценкой
аннотации нашла излишним.
Таким образом в итоге для S. shibatae рассматривался белок
Maltooligosyl trehalose synthase.
Ссылка на .txt файл:
Q7LYV2.
2. Информация о белке
Белок носит название мальтоолигозилтрегалозо-синтазы. Ген кодирующий белок - treY. Структура участвует в процессах метаболизма углеводов, конкретно в биосинтезе трегалозы (дисахарида, предположительно помогающего справиться с засушливыми условиями). Из примечательных структурных особенностей - есть два участка, участвующих в формировании дисульфидных связей. В файле ~/term2/pr7/protein_info.txt представлен отчет по некоторым характеристикам белка.
3. Кластеры похожих белков
| Уровень идентичности | Размер кластера |
|---|---|
| UniRef100 | 2 |
| UniRef90 | 22 |
| UniRef50 | 53 |
В файле ~/term2/pr7/protein_clusters.txt представлен отчет по кластерам UniRef50, UniRef90 и UniRef100 белка.
На мой взгляд размеры кластеров UniRef позволяют сделать вывод о малой распространенности (UniRef100),
но достаточно высокой степени консервативности (UniRef90 и UniRef50) белка.
При этом белок считается репрезентативным только в кластере UniRef100,
что подтверждает низкую оценку его аннотации.
4. Поисковые запросы
Далее приведены результаты поиска по белку и кодирующему его гену.
Поиск по UniParc
В первую очередь решила провести поиск своего белка по Uniparc.
Ссылка на запись.
Архив уникальных последовательностей предоставил информацию о специальном идентификаторе UPI00000627AD.
Запись включает не только выбранный белок, но и белок Q9UWN8 (последний присутствует в кластере UniRef100).
Помимо этого перечислены ссылки на базы EMBL. Все 4 представленные базы данных активны.
В записи также можно найти информацию по уникальной хеш-сумме (4E2502E37A5F534C), массе последовательности (86,192 Da).
Представлены и
InterPro
группы (классификация по функциям) с идентификаторами для источников данных (например,
Glycosyl hydrolase, family 13, catalytic domain).
Поиск протеома
В первую очередь был рассмотрен протеом всей археи: ссылка. Выявлно, что всего белков 3143, из которых 3120 кодируются хромосомными участками и 34 гена из плазмиды. ID протеома: UP000694018. Про мой белок (Q7LYV2) из базы SMART известно, что он относится к О-гликозил гидролазам (EC 3.2.1.). Был проведен поиск белков со схожей функцией в S. shibatae B12, результаты представлены в таблице 2. Был найден среди О-гликозил гидролаз белок с AC Q7LYV3 и аналогичным ID с рассматриваемым мною, поэтому было решено его изучить поглубже.
| Поисковый запрос | Классификация фермента | Количество белков |
|---|---|---|
| (ec:3.) AND (taxonomy_id:523848) | EC 3... | 136 |
| (ec:3.2.) AND (taxonomy_id:523848) | EC 3.2.. | 24 |
| (ec:3.2.1) AND (taxonomy_id:523848) | EC 3.2.1. | 20 |
| Характеристика | Q7LYV2 | Q7LYV3 |
|---|---|---|
| Название | Maltooligosyl trehalose synthase | Malto-oligosyltrehalose trehalohydrolase |
| Кодирующий ген | treY | treZ |
| Длина | 728 | 559 |
| Доказательство существования | На уровне белка | Выведен из гомологии |
| Наличие домена с SM00642 | Да | Да |
Дальнейшее изучение Q7LYV3
Ссылка на запись. Основная информация представлена в таблице 3.
Структура в целом хорошо аннотирована, даже где-то подробнее основной рассматриваемой.
Про белок известно, что он функционирует в цитоплазме и его структура предсказана AlphaFold.
Был выполнен поиск Q7LYV3 по кластерам нашего белка и выяснено, что даже в UniRef50 он отсутствует.
Из чего можно сделать вывод, что несмотря на схожесть в названиях и функциях эти белки
имеют и существенные отличия, отраженные на уровне их аминокислотной последовательности.
Вероятно участвуя в схожих биологических процессах, они активируются на разных этапах для
выполнения непохожих ролей.
Поиск по кодирующему гену
Выполнялся поиск по treY. Результаты представлены в таблице 4. Ген более распространен у бактерий, для архей он малоизучен, вероятно, потому что и сам таксон изучен не так глубоко. Ген не сильно распространен, что видно по его малым вхождениям в метагеномные конструкции. Больше белков treY в S. shibatae B12 не кодирует ((gene:treY) AND (taxonomy_id:523848)).
| Группа организмов | Общее число записей | Записи в Swiss-Prot | Поисковый запрос |
|---|---|---|---|
| Всего | 5585 | 3 | (gene:treY) |
| Бактерии | 5561 | 3 | (gene:treY) AND (taxonomy_id:2) |
| Археи | 21 | 0 | (gene:treY) AND (taxonomy_id:2157) |
| Метагеном | 3 | 0 | (gene:treY) NOT (taxonomy_id:2) NOT (taxonomy_id:2157) |
5. Поиск источника аннотации
При описании доказательства какой-то информации используются специальные тэги и код из ECO (Evidence and Conclusion Ontology) указывает на тип источника, также может прилагаться информация о самом источнике. В Q7LYV2 представлено три типа ECO кодов.
ECO:0000313|EMBL:AAM81591.1
Ссылка на источник
Данный код используется в автоматически составленных записях
для информации взятой из другой базы данных (название и идентификатор источника указываются).
Отсылка здесь на EMBL, где представлена статья по treY в изучаемой архее. Можно получить доступ к
EMBL
и
FASTA форматам.
Указано, что стартовым кодоном является первый.
В записи Q7LYV2 отсылка на этот источник идет довольно часто: полное название - DE, название гена - GN
(ссылка идет на запись, посвященную этому гену),
идентификатор S. shibatae B12 - OX, а также в референсных полях номера - RN и комметариев - RC.
Записи доверяю в достаточной степени. Составлена в 2002 году, но была перепроверена в 2016, например,
удалялись векторные контаминации.
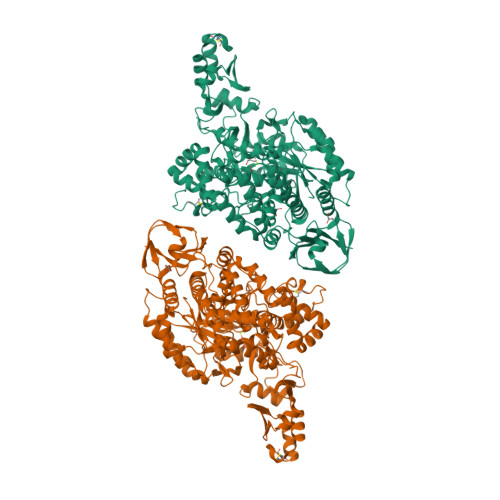
ECO:0007829|PDB:5ZCR
Ссылка на источник
Код используется в автоматически составленных записях для информации, полученной
комбинацией лабораторных и компьютерных методов. Используется для больших данных о протеоме и, как
в данном случае, для 3D структур, в работе с которыми использовались вычислительные машины.
PDB код предоставляется.
Белок классифицируют в гидролазы, считается синтазой гликозилтрегалозы.
Он состоит из двух отличных биологических единиц, есть мутации.
Из малых молекул есть магний и глицерин.
В записи Q7LYV2 отсылка на этот источник идет пореже: во втором референсном номере - RN,
в ключевых словах по поводу 3D-структуры - KW (отсылка идет на базу данных третичных структур),
при обозначении дисульфидых связей - FT (снова связка с пространственной структурой).
Запись составлена в 2018 году, хорошо аннотирована, доверяю.
ECO:0000259|SMART:SM00642
Ссылка на источник
Код используется в автоматически составленных записях для доказательств из InterPro.
InterPro занимается функциональным анализом белка, классификацией по семействам, доменам.
В записи дана краткая справка по группе, к которой относится данный белок - О-гликозил-гидролазы
(EC 3.2.1.13). Они гидролизуют гликозидную связь между двумя и более углеводородами или между углеводородом
и неуглеводородной группой. Таким образом для мальтоолигозилтрегалоз-синтазы характерна
ферментативная активность, проявляемая в процессах метаболизма углеводов.
Описана типичная структурная домена (alpha-amylase domain) с его активным сайтом.
В записи Q7LYV2 отсылка на этот источник идет всего один раз,
при упоминании домена третичной структуры - FT (соответсвует сфере деятельности InterPro).
На мой взгляд запись составлена неплохо, но функциональной классификации не сильно бы доверяла.
Сходство доменов тем не менее зачастую удобно, особенно при предсказании белков.