Практикум 8
UniProt Proteomes, EMBOSS, bash
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Выбор и скачивание протеомов
В качестве основного был выбран протеом штамма 98/2 SULC Saccharolobus solfataricus (Sulfolobus solfataricus),
поскольку этот организм относится к тому же роду, что и архея, которой в прошлом семестре посвящался
мини-обзор.
При этом при поиске искались представилители из Saccharolobus с BUSCO от 97%, с целью получить более
качественный протеом:
(taxonomy_id:2100760) AND (busco:[97 TO *]).
Подбирался протеом с неотличительным размером: CPD Standard. Из всех выбиравшихся микроорганизмов
только для протеома S. solfataricus приводились ссылки на статьи, поэтому решено было искать среди его
штамов:
(taxonomy_id:2287)и
(taxonomy_id:2287) AND (cpd:1). В итоге решено было взять
штамм 98/2 SULC.
Контрольный протеом был взят для Nitrosopumilus maritimus (strain SCM1). Поскольку Saccharolobus
относится к археям, то и контроль искался из данной группы организмов:
(taxonomy_id:2157) AND (cpd:1) AND (busco:[95 TO *]). Многие археи относятся к Thermoproteata, но в данном
случае искался протеом организма не приспособленного к жизни при экстремальных температурах и pH.
Nitrosopumilus maritimus подошел по этим параметрам, являясь часто встречаемой в морских водах археей.
Аннотация протеома также показалсь достаточно качественной, особенно учитывая, что он был отнесен к референсным.
Результаты первичного сравнительного анализа протеомов приведены в таблице 1.
| Сведения | Протеом близкородственной археи | Контрольный протеом |
|---|---|---|
| Идентификатор | UP000033057 | UP000000792 |
| Таксон | Saccharolobus solfataricus (Sulfolobus solfataricus) | Nitrosopumilus maritimus (strain SCM1) |
| Общее количество белков | 2604 | 1795 |
| Степень изученности (status) | Other proteome | Reference proteome |
| Количество белков протеома в базе swiss-prot | 2 (0,08%) | 93 (5,18%) |
| BUSCO | C:99.3% (S:99.1% D:0.2%) F:0.1% M:0.6% | C:99.3% (S:98.8% D:0.6%) F:0.3% M:0.4% |
| CPD | Standard | Standard |
2. Сравнение протеомов по представленности определенных групп белов
Для вычисления долей, которые составляют белки некоторой "функциональной группы" решено было воспользоваться скриптами на python. Kоды всех использованных программ. При этом для того чтобы понять, где указывается информация о конкретной функциональной группе использовались разнообразные команды bash. Примеры использованных команд. Основные результаты представлены в таблице 2.
| "Функциональная группа" | UP000033057 | UP000000792 (контроль) |
|---|---|---|
| Трансмембранные белки | 19.97% (520) | 17.66% (317) |
| Ферменты | 16.01% (417) | 19.00% (341) |
| ДНК-связывающие белки | 0.81% (21) | 0.11% (2) |
| Галактозидазы | 0.08% (2) | 0.00% (0) |
| Белки теплового шока | 0.12% (3) | 0.28% (5) |
| Домены холодового шока | 0.00% (0) | 0.06% (1) |
Анализ результатов
В протеоме Saccharolobus solfataricus содержится больший процент трансмембранных белков чем в Nitrosopumilus maritimus, с числом ферментативных белков ситуация противоположная. Тем не менее представленность обеих "функциональных групп" практически идентичная. В случае поиска числа ферментов по базам UniProtKB получались те же результаты:
вероятно, потому что существует конкретная система определения
принадлежности белка к ферментам и единый способ кодировки EC:*.
В качестве третьей, четвертой и пятой группы белков были выбраны белки характерные для археи S. solfataricus.
Для клады Термопротеот характерны особые белки, связывающие ДНК, поэтому мне стало интересно изучить насколько
представленность ДНК-связывающих белков в моем основном протеоме отличается от представленности в контрольном
- получилось, что в Saccharolobus их в 7 раз больше. Примечательно, что при поиске по UniprotKB:
получается, что в обоих протеомах только по одному ДНК-связывающему белку. Предположительно, это говорит
о не сильно качественной аннотированности протеомов, в особенности для S. solfataricus.
При попытке поиска галактозидаз в базе UniProtKB получены нулевые результаты в обоих случаях:
- (proteome:UP000033057) AND (cc_function:galactosidase);
- (proteome:UP000000792) AND (cc_function:galactosidase).
Результаты скрипта на Python представлены в таблице 2 и оба белка имели ECO:0000313:
- DE SubName: Full=Alpha-galactosidase {ECO:0000313|EMBL:AKA75954.1};
- DE SubName: Full=Beta-galactosidase {ECO:0000313|EMBL:SAI86668.1}.
Таким образом записи об обоих белках были автоматически составлены и информация взята из соответствующих баз в EMBL. Возможно, именно этим вызваны неточности при попытке использования поисковых запросов к UniProtKB.
Последняя группа белков - белки теплового шока по определению должны быть у термофильных, а тем более
гипертермофильных организмов, какими и являются саххаралобусы. Результаты поиска представлены в таблице 2, а также
в разделе результатов программ. Видно, что у S.solfataricus действительно представлены белки теплового шока,
но макромолекулы с такой же функцией и даже в большем отношении есть и у N. maritimus. Это удивительно,
потому что изначально не предполагается термоустойчивости у Nitrosopumilus.
Также было сделано и вполне логичное открытие: помимо белков теплового шока у N. maritimus также есть домен
холодового шока, который обнаруживается в ДНК-связывающих белках и позволяет переживать спады температур. Возможно
он также влияет на малость числа ДНК-связывающих белков в протеоме этой археи, ведь повышается их качество.
При поиске по базе UniProtKB все белки находятся успешно:
- (proteome:UP000033057) AND Heat-shock;
- (proteome:UP000000792) AND "heat shock protein";
- (proteome:UP000000792) AND "Cold shock".
в случае поиска белков теплового шока даже выводится
шаперон DNAK_NITMS, который возможно выполняет функцию восстановления структур белка,
пострадавших от повышения температуры.
Практически все из этих 9 белков имеют код характерный для автоматически составленных
записей на информации взятой из другой базы данных. Но у N. maritimus одна запись (ECO:0000255)
была составлена частично автоматически, частично с ручным вмешательством человека на том или ином этапе.
Результаты выдачи программ
Трансмембранные белки
aliserana@kodomo:~/term2/pr8$ ./searchtm.py
This program calculates the fraction of transmembrane proteins in a proteome.
Where to get information from?
UP000033057.swiss.gz
Occurence of transmembranes: 520 in 2604 proteins,
fraction: 19.97aliserana@kodomo:~/term2/pr8$ ./searchtm.py
This program calculates the fraction of transmembrane proteins in a proteome.
Where to get information from?
UP000000792.swiss.gz
Occurence of transmembranes: 317 in 1795 proteins,
fraction: 17.66Ферменты
aliserana@kodomo:~/term2/pr8$ ./searchen.py
This program calculates the fraction of enzyme proteins in a proteome.
Where to get information from?
UP000033057.swiss.gz
Occurence of enzymes: 417 in 2604 proteins,
fraction: 16.01aliserana@kodomo:~/term2/pr8$ ./searchen.py
This program calculates the fraction of enzyme proteins in a proteome.
Where to get information from?
UP000000792.swiss.gz
Occurence of enzymes: 341 in 1795 proteins,
fraction: 19.00ДНК-связывающие белки
aliserana@kodomo:~/term2/pr8$ ./searchdnab.py
This program calculates the fraction of DNA-binding proteins in a proteome.
Where to get information from?
UP000033057.swiss.gz
Occurence of DNA-binding proteins: 21 in 2604 proteins,
fraction: 0.81aliserana@kodomo:~/term2/pr8$ ./searchdnab.py
This program calculates the fraction of DNA-binding proteins in a proteome.
Where to get information from?
UP000000792.swiss.gz
Occurence of DNA-binding proteins: 2 in 1795 proteins,
fraction: 0.11Галактозидазы
aliserana@kodomo:~/term2/pr8$ ./searchgal.py
This program calculates the fraction of galactosidases in a proteome.
Where to get information from?
UP000033057.swiss.gz
Occurence of galactosidase proteins: 2 in 2604 proteins,
fraction: 0.08aliserana@kodomo:~/term2/pr8$ ./searchgal.py
This program calculates the fraction of galactosidases in a proteome.
Where to get information from?
UP000000792.swiss.gz
Occurence of galactosidase proteins: 0 in 1795 proteins,
fraction: 0.00Белки теплового шока
aliserana@kodomo:~/term2/pr8$ ./searchhsp.py
This program calculates the fraction of heat-shock proteins in a proteome.
Where to get information from?
UP000033057.swiss.gz
DE SubName: Full=Heat-shock protein Hsp20 {ECO:0000313|EMBL:SAI86285.1};\n'
DE SubName: Full=Heat-shock protein Hsp20 {ECO:0000313|EMBL:SAI86105.1};\n'
DE SubName: Full=Heat-shock protein HspX {ECO:0000313|EMBL:AKA78293.1};\n'
Occurence of heat-shock proteins: 3 in 2604 proteins,
fraction: 0.12aliserana@kodomo:~/term2/pr8$ ./searchhsp.py
This program calculates the fraction of heat-shock proteins in a proteome.
Where to get information from?
UP000000792.swiss.gz
DE AltName: Full=Heat shock 70 kDa protein {ECO:0000255|HAMAP-Rule:MF_00332};\n'
DE SubName: Full=Cold-shock DNA-binding domain protein {ECO:0000313|EMBL:ABX12423.1};\n'
DE SubName: Full=Heat shock protein Hsp20 {ECO:0000313|EMBL:ABX13604.1};\n'
DE SubName: Full=Heat shock protein DnaJ domain protein {ECO:0000313|EMBL:ABX13663.1};\n'
DE SubName: Full=Heat shock protein DnaJ domain protein {ECO:0000313|EMBL:ABX12633.1};\n'
DE SubName: Full=Heat shock protein HSP20 {ECO:0000313|EMBL:ABX11910.1};\n'
Occurence of heat-shock proteins: 5 in 1795 proteins,
fraction: 0.283. Сравнение протеомов
Для всех вычислений решено было воспользоваться скриптами на python. Kоды всех использованных программ.
Анализ результатов
В первую очередь была произведена попытка найти общие для обоих изучаемых протеомов белки, используя базу UniProtKB: (proteome:UP000033057) AND (proteome:UP000000792). Получилось, что общих белков согласно базе данных у архей нет. Действительно, в записях каждый белок закреплен за конкретным организмом.
Затем исследования выполнялись с использованием Python.
Определено было, является ли метионин первой аминокислотой в каждом белке, и выявлено, что
все белки в обоих протеомах начинаются со старт-кодона (таблица 3).
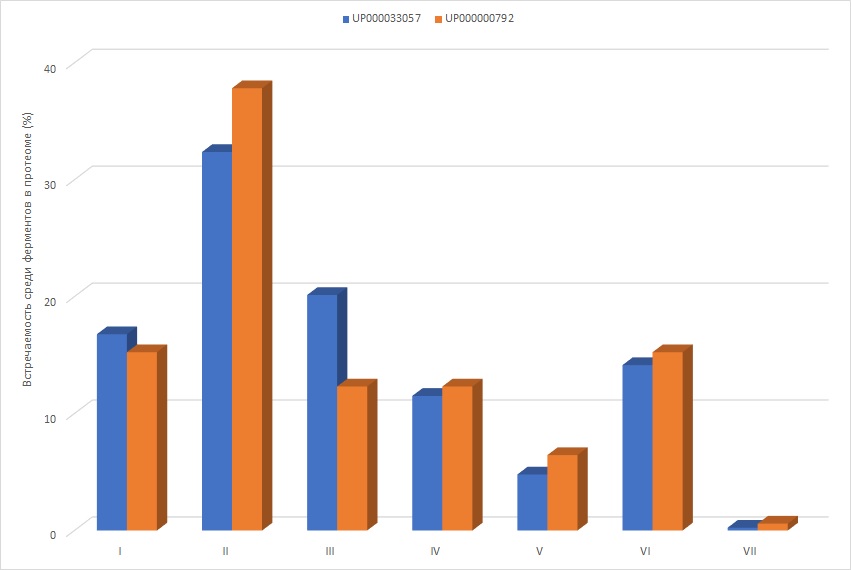
Выявление распространенности каждого типа фермента в соответствующих протеомах показало, что
в протеоме у обоих архей присутствуют все группы ферментов (рисунок 1).
Предположительно содержание изомераз для контрольного протеома выше, потому что Nitrosopumilus
обитает в более умеренных условиях и вероятность, что изменившаяся конфигурация уменьшит его
шансы на выживание ниже.
Для S. solfataricus характернее гидролазы,
а для N. maritimus -, наоборот, трансферазы.
Возможно, для контрольного организма это связано с его участием в нитрификации и метаболизмом, основанным на
окислении аммиака - в ходе биохимических циклов они вынуждены часто переносить одни молекулы на другие.
Для Saccharolobus связать преобладание гидролаз можно с их экологией. Ацидофильность обуславливает
необходимость присутствия большой концентрации свободных протонов и вероятно реакции гидролиза (в ходе,
которых может высвобождаться H+ помогают археям переживать неблагоприятные для них условия.
S. solfataricus термофилен и высокие температуры при наличии термостойких ферментов способствуют еще
большему ускорению реакций, предположительно поэтому
эти организмы не нуждаются в трансферазах настолько же, насколько мезофилы.
| "Признак сравнения" | UP000033057 | UP000000792 (контроль) |
|---|---|---|
| Белки, начинающиеся с Met | 100% (2604) | 100% (1795) |
| Наиболее и наименее распространенные аминокислоты во всем протеоме |
Leu: 10.31% (74437)
Cys: 0.62% (4476) |
Ile: 8.45% (41712)
Trp: 0.62% (4404) |
| Наиболее популярные среди наиболее и наименее популярных аминокислот в каждом отдельном белке |
Leu: 39.36% (1025)
Cys: 5.28% (1700) |
Lys: 29.53% (530)
Trp: 56.49% (1014) |
| Отношение полярно заряженных к полярно незаряженным аминокислотам | 0.89 | 0.95 |
Затем были проведены исследования на встречаемость аминокислот. В первую очередь это общее содержание каждой аминокислоты в протеоме (рисунок 2). Согласно Ghosch et al высокое содержание глутаминовой кислоты в термофильных организмах повышает энтропию конформаций белка, но в данном случае, наоборот, содержание аминокислоты в основном изучаемом протеоме даже слегка ниже чем в контрольном. Глутамина и аспарагина у S. solfataricus должно быть, наоборот, меньше и видно, что это выполняется только для глутамина. У гипертермофиллов Ser, Gly, Lys, Asp должны стремиться заменяться на Thr, Ala, Arg и Glu соответственно. Но в данном случае, можно предположить только замену некоторой доли лизинов на аргинины у S. solfataricus. Высокое содержание изолейцина и валина можно связать с их вкладом гидрофобность и жесткость белков.
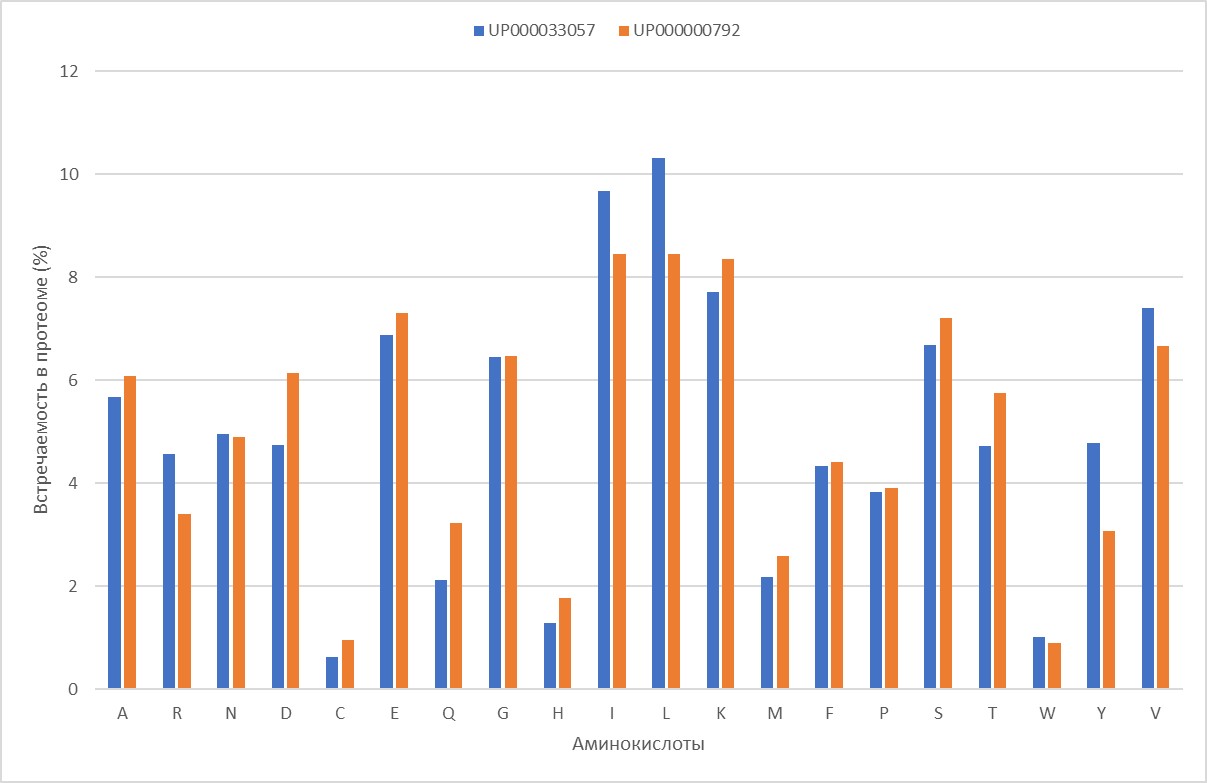
Наиболее и наименее распространенные аминокислоты при охвате всего протеома и самые популярные среди
наиболее и наименее популярных аминокислот в каждом отдельном белке почти совпадают. Стоит отметить, что
высокое содержание лейцина в S. solfataricus вероятно связано с разветвленностью и неполярностью остатка
этой аминокислоты. Цистеин и триптофан, как правило, нечасто встречаются среди архей. Характерные для
N. maritimus изолейцин и особенно лизин
обычно не являются одними из самых распространенных аминокислот среди архей.
Среди термофилов, по сравнению с другими группами организмов, характерно более высокое отношение полярных
заряженных аминокислот к полярным незаряженным, но в данном случае наблюдается обратная ситуация.
Результаты выдачи программ
Определение первой аминокислоты
aliserana@kodomo:~/term2/pr8$ ./searchmet.py
This program calculates if there are any proteins that have not methionine as their first aminoacid.
Where to get information from?
UP000033057.swiss.gz
Occurence of methionine: 2604 in 2604 proteins,
fraction: 100.00aliserana@kodomo:~/term2/pr8$ ./searchmet.py
This program calculates if there are any proteins that have not methionine as their first aminoacid.
Where to get information from?
UP000000792.swiss.gz
Occurence of methionine: 1795 in 1795 proteins,
fraction: 100.00Определение класса ферментов
aliserana@kodomo:~/term2/pr8$ ./searchenclass.py
This program calculates the fraction of each type of enzyme proteins in a proteome.
Where to get information from?
UP000033057.swiss.gz
Occurence of all enzymes: 417 in 2604 proteins,
fraction: 16.01
Occurence of oxidoreductases: 70 (16.79%) in 417 enzymes
Occurence of transferases: 135 (32.37%) in 417 enzymes
Occurence of hydrolases: 84 (20.14%) in 417 enzymes
Occurence of lyases: 48 (11.51%) in 417 enzymes
Occurence of isomerases: 20 (4.80%) in 417 enzymes
Occurence of ligases: 59 (14.15%) in 417 enzymes
Occurence of translocases: 1 (0.24%) in 417 enzymesaliserana@kodomo:~/term2/pr8$ ./searchenclass.py
This program calculates the fraction of each type of enzyme proteins in a proteome.
Where to get information from?
UP000000792.swiss.gz
Occurence of all enzymes: 341 in 1795 proteins,
fraction: 19.00
Occurence of oxidoreductases: 52 (15.25%) in 341 enzymes
Occurence of transferases: 129 (37.83%) in 341 enzymes
Occurence of hydrolases: 42 (12.32%) in 341 enzymes
Occurence of lyases: 42 (12.32%) in 341 enzymes
Occurence of isomerases: 22 (6.45%) in 341 enzymes
Occurence of ligases: 52 (15.25%) in 341 enzymes
Occurence of translocases: 2 (0.59%) in 341 enzymesОпределение встречаемости каждой аминокислоты в протеоме
aliserana@kodomo:~/term2/pr8$ ./searchamprot.py
This program calculates the most popular aminoacid in the entire protein.
Where to get information from?
UP000033057.swiss.gz
A: 5.68% (40990) R: 4.57% (32977) N: 4.96% (35816) D: 4.75% (34276) C: 0.62% (4476) E: 6.89% (49705) Q: 2.13% (15348) G: 6.46% (46588) H: 1.28% (9247) I: 9.68% (69863) L: 10.31% (74437) K: 7.71% (55675) M: 2.18% (15759) F: 4.34% (31321) P: 3.82% (27586) S: 6.68% (48176) T: 4.72% (34076) W: 1.02% (7396) Y: 4.78% (34488) V: 7.41% (53478)
Occurence of the most popular aminoacid(s) L: 74437 (10.31%) in 721678 aminoacids (2604 proteins).
Occurence of the least popular aminoacid(s) C: 4476 (0.62%) in 721678 aminoacids (2604 proteins).This program calculates the most popular aminoacid in the entire protein.
Where to get information from?
UP000000792.swiss.gz
A: 6.08% (30006) R: 3.41% (16826) N: 4.91% (24217) D: 6.15% (30341) C: 0.95% (4713) E: 7.31% (36092) Q: 3.22% (15895) G: 6.47% (31944) H: 1.77% (8747) I: 8.45% (41712) L: 8.45% (41693) K: 8.35% (41226) M: 2.59% (12776) F: 4.40% (21741) P: 3.90% (19261) S: 7.20% (35560) T: 5.75% (28383) W: 0.89% (4404) Y: 3.08% (15216) V: 6.66% (32867)
Occurence of the most popular aminoacid(s) I: 41712 (8.45%) in 493620 aminoacids (1795 proteins).
Occurence of the least popular aminoacid(s) W: 4404 (0.89%) in 493620 aminoacids (1795 proteins).Определение отношения полярных заряженных к полярным незаряженным аминокислотам
aliserana@kodomo:~/term2/pr8$ ./searchamprotdev.py
This program calculates the most popular aminoacid in the entire protein.
Where to get information from?
UP000033057.swiss.gz
Ratio of polar charged versus polar uncharged amino acids: 0.89.aliserana@kodomo:~/term2/pr8$ ./searchamprotdev.py
This program calculates the most popular aminoacid in the entire protein.
Where to get information from?
UP000000792.swiss.gz
Ratio of polar charged versus polar uncharged amino acids: 0.95.Определение встречаемости аминокислот по наиболее и наименее популярным в каждом отдлельном белке
aliserana@kodomo:~/term2/pr8$ ./searchampr.py
This program calculates the most popular aminoacid among the most popular for each protein.
Where to get information from?
UP000033057.swiss.gz
Most popular: GLIKVAERSNDTYPFCQ
Least popular: CWNHSQMPFYDRTAELVKGI
Occurence of the most popular aminoacid(s) L: 1025 (39.36%) proteins in (2604 proteins).
Occurence of the least popular aminoacid(s) C: 1700 (65.28%) proteins in (2604 proteins).aliserana@kodomo:~/term2/pr8$ ./searchampr.py
This program calculates the most popular aminoacid among the most popular for each protein.
Where to get information from?
UP000000792.swiss.gz
Most popular: SKIEALGVFTPDRNQCM
Least popular: CWHQYMGVFNDPRALKTSIE
Occurence of the most popular aminoacid(s) K: 530 (29.53%) proteins in (1795 proteins).
Occurence of the least popular aminoacid(s) W: 1014 (56.49%) proteins in (1795 proteins).Вывод
Протеомы Saccharolobus solfataricus и Nitrosopumilus maritimus безусловно похожи между собой. Но есть и отличия, которые не всегда совпадают с ожиданиями.
Список литературы
- Информация о S. solfataricus
- Информация о N. maritimus
- Информация о cold-shock domain
- Информация об ацидофилах
- Информация о встречаемости аминокислот
-
Ghosh, Shilpi & Lepcha, Khusboo & Basak, Arijita & Mahanty, Ayan. (2020). Thermophiles and thermophilic hydrolases. 10.1016/B978-0-12-818322-9.00016-2.