Практикум 4
Автор старался, но не может гарантировать отсутствие биологических ошибок.
1. Составление списка гомологичных белков, включающих паралоги
Был проведен поиск достоверных гомологов белка CLPX_ECOLI среди раннее выбранных мною бактерий. Для этого воспользовались файлами полных протеомов бактерий, лежащими на kodomo в директории /P/y22/term4/Proteomes. Протеомы моих бактерий были перенесены в общий файл ~/term4/pr4/myproteomes.fasta для дальнейшего создания библиотеки на их основе. Затем был проведен поиск программой blastp (Protein-Protein BLAST 2.11.0+) гомологов по протеомам моих бактерий. Далее представлены использованные команды:
aliserana@kodomo:/P/y22/term4/Proteomes$ cat AROAE.fasta BURMA.fasta HAEIN.fasta NEIMA.fasta POLAQ.fasta ROSDO.fasta SERP5.fasta THIDA.fasta > ~/term4/myproteomes.fasta
aliserana@kodomo:~/term4$ makeblastdb -dbtype prot -in pr4/myproteomes.fasta -out myprot_db
aliserana@kodomo:~/term4/pr4$ blastp -db myprot_db -query P0A6H1.fasta -out homologs -evalue 0.0005Список находок из выдачи BLAST: файл, распределение по организмам - таблица 1.
| Название | Мнемоника | Количество находок | ID находок |
|---|---|---|---|
| Aromatoleum aromaticum | AROAE | 2 | CLPX_AROAE, HSLU_AROAE |
| Burkholderia mallei | BURMA | 3 | CLPX_BURMA, HSLU_BURMA, A0A0H2WJ72_BURMA |
| Haemophilus influenzae | HAEIN | 3 | CLPX_HAEIN, HSLU_HAEIN, FTSH_HAEIN |
| Neisseria meningitidis | NEIMA | 2 | CLPX_NEIMA, RUVB_NEIMA |
| Polynucleobacter asymbioticus | POLAQ | 2 | CLPX_POLAQ, A4SXL5_POLAQ |
| Roseobacter denitrificans | ROSDO | 3 | CLPX_ROSDO, HSLU_ROSDO, RUVB_ROSDO |
| Serratia proteamaculans | SERP5 | 4 | CLPX_SERP5, HSLU_SERP5, A8GCD8_SERP5, A8G901_SERP5 |
| Thiobacillus denitrificans | THIDA | 4 | CLPX_THIDA, Q3SFW1_THIDA, Q3SJR4_THIDA, Q3SJH1_THIDA |
2. Реконструкция и визуализация
Подоготовка последовательностей
Поместим последовательности находок в fasta-файл, отредактировав названия - оставим только ID.
Реконструкция дерева
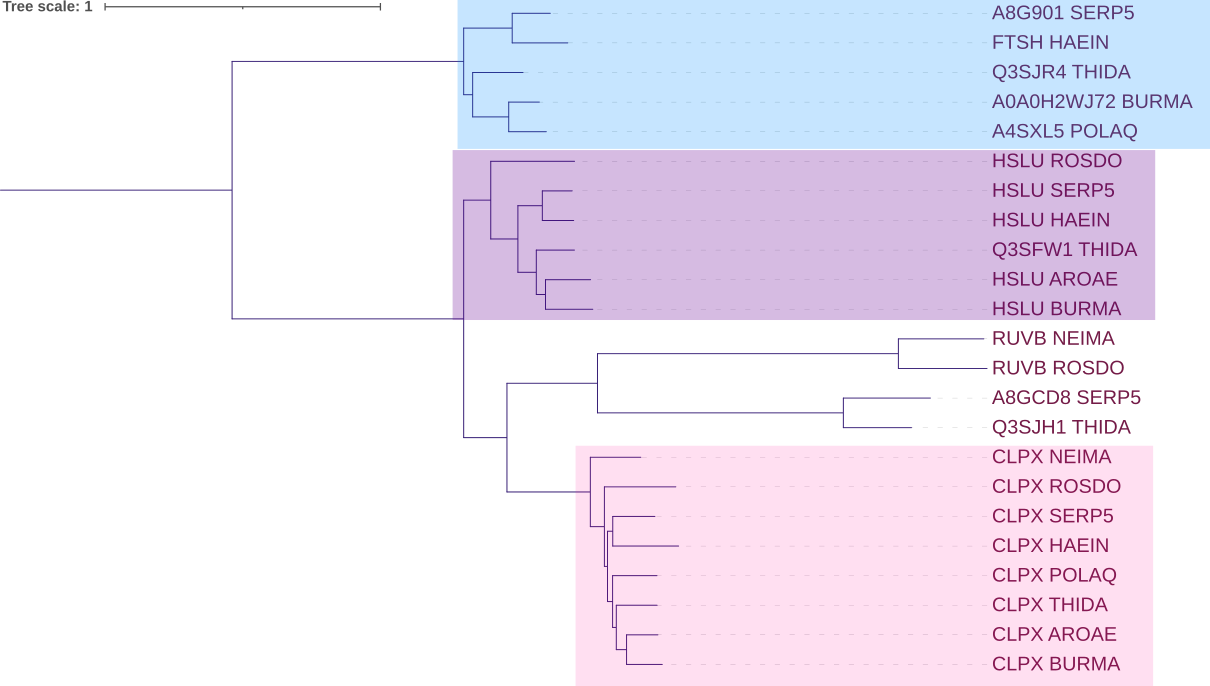
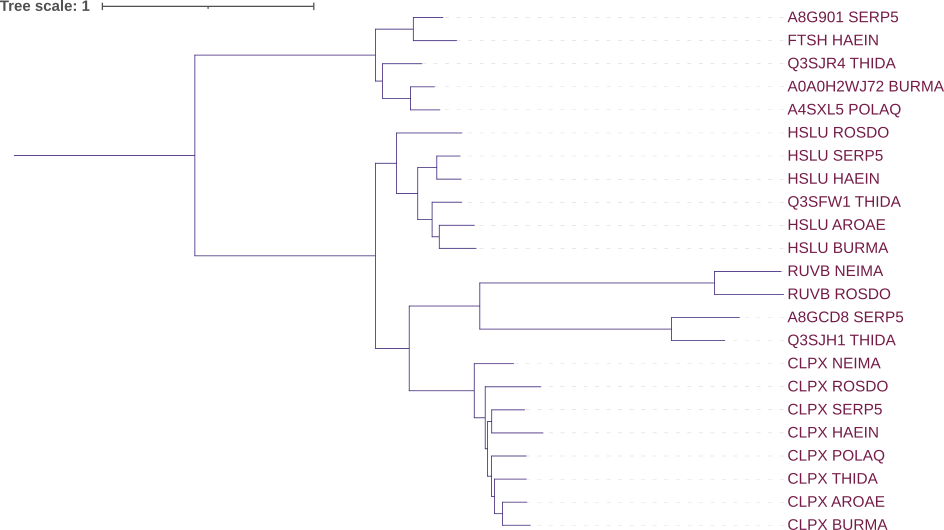
Реконструируем дерево найденных гомологов через сервис NGPhylogeny конвейером из MAFFT и программы FastME со следующими параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик. Ссылка на результат. Newick формула дерева без укоренения, с укоренением в среднюю точку. На рисунке 1 представлено полученное дерево после укоренения в среднюю точку.
Паралоги и ортологи
Будем считать, что наше дерево реконструировано правильно. Найдем по нему пары паралогов и ортологов. Считаем, что гомологичные белки ортологи, если они из разных организмов, разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования, два белка - паралоги, если они из одного организма.
Паралоги
- RUVB_ROSDO и CLPX_ROSDO
- A8G902_SERP5 и HSLU_SERP5
- Q3SFW1_THIDA и Q3SJR4_THIDA
Ортологи
- CLPX_THIDA и CLPX_HAEIN
- RUVB_NEIMA и RUVB_ROSDO
- HSLU_SERP5 и HSLU_HAEIN
Изображения дерева
Изобразим разные ортологические группы (то есть наборы попарно ортологичных белков) разными цветами (две группы, содержащие две последовательности, остались чёрными) - рисунок 2.
- FISH - группа АТФ-зависимых цинк металлопротеаз, контролирует качество интегральных мембранных белков.
- HSLU - группа АТФазных субъединиц HslU АТФ-зависимых протеаз. Связывание АТФ и его последующий гидролиз необходимы для подготовки субстрата к HslV гидролизу.
- CLPX - группа АТФ-связыващих субъединиц ClpX АТФ-зависимых протеаз Clp. ClpX направляет протеазу к специфичному субстрату.
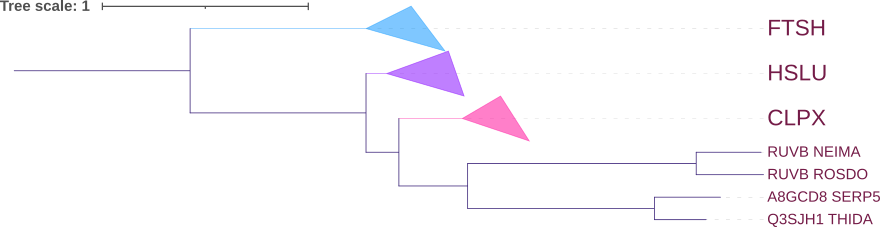
"Схлопнем" все ортологические группы, содержащие более трёх последовательностей - рисунок 3. Таблица 2 показывает белки каких бактерий отстуствуют в данных ортологических группах. По рисункам 4-6 определим соответствие филогений белков и бактерий (дерево).
- FISH - для представленных в ней белков филогения по ним полностью соответствует филогении бактерий.
- HSLU - для представленных белков наблюдается несоответствие внутри клады {HSLU_AROAE, HSLU_BURMA, Q3SFW1_THIDA} с соответствующей для бактерий, в остальном деревья совпадают.
- CLPX - филогения белков не соотвествует филогении бактерий. Правильно выделены нетривиальные ветви для {SERP5, HAEIN}, {AROAE, THIDA, BURMA, POLAQ}. При этом филогения внутри последней клады для белков не совпадает с той у бактерий.



| Группа | FTSH | HSLU | CLPX |
|---|---|---|---|
| Число белков | 5 | 6 | 8 |
| Состав | A0A0H2WJ72_BURMA, FTSH_HAEIN, A4SXL5_POLAQ, A8G901_SERP5, Q3SJR4_THIDA | HSLU_AROAE, HSLU_BURMA, HSLU_HAEIN, HSLU_ROSDO, HSLU_SERP5, Q3SFW1_THIDA | CLPX_AROAE, CLPX_BURMA, CLPX_HAEIN, CLPX_NEIMA, CLPX_POLAQ, CLPX_ROSDO, CLPX_SERP5, CLPX_THIDA |
| Непредставленные организмы | AROAE, NEIMA, ROSDO | NEIMA, POLAQ | - |