Поиск мотивов (блоков достоверного выравнивания) среди гомологов белка ybbD_BACSU.
MEME - наиболее популярная программа множественного локального выравнивания. Она находит блоки - участки локальных выравниваний без гэпов. В пакете EMBOSS эта программа носит название ememe. Именно ей я воспользовалась для нахождения мотивов среди гомологов белка ybbD_BACSU. Для получения результатов был введён следующий запрос:
ememe seq.fasta memeout -nmotifs 3,
где memeout - имя директории для результата, а -nmotifs 3 - это ограничение по количеству мотивов, выдаваемых в результатах. Полную выдачу программы можно посмотреть здесь.
Таблица 1. Краткая характеристика мотивов.
| Мотив | Сколько последовательностей его содержит (всего 36) | Длина | E-value | LOGO |
| MOTIF 1 | 27 | 29 | 4.8e-399 |  |
| MOTIF 2 | 27 | 29 | 5.6e-379 |  |
| MOTIF 3 | 35 | 29 | 1.5e-397 | 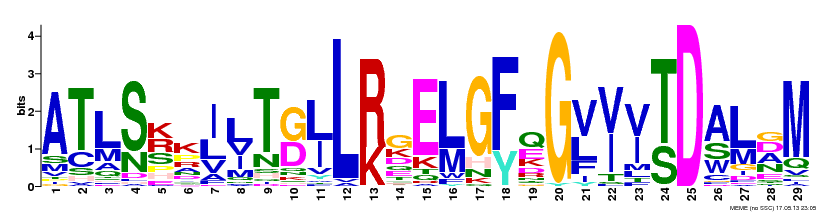 |
Сравнение блоков с полным выравниванием, составленным muscle.
С помощью средств Jalview были сопоставлены полученные выравнивания с множественным выравниванием гомологов белка ybbD_BACSU. Результаты можно посмотреть на рисунке 1. Поскольку MEME выбрал мотивы не во всех последовательностях, чаще искулючая эукариот, то в выравнивании эти мотивы часто разделены гэпами, т.к. в 1-2 последовательностях видна вставка внутри мотива.
Рис.1. Сравнение мотивов MEME с множественным выравниванием белка ybbD_BACSU. Номера мотивов слева направо - 3,1,2.
Можно заметить, что повсеместно всю картину портят эукариоты, всё-таки их белки сильнее отличаются от прокариотических, чем прокариотические между собой. Если исключить эукаротов из выравнивания, то можно будет не разделять мотивы гэпами, тогда они полностью совпадут с выравниванием. Проект в формате .jar можно скачать здесь
Поиск найденных мотивов в других последовательностях
С помощью программу MAST можно искать мотивы MEME в других последовательностях. Для этого нужно создать файл в формате fasta с последовательностями, где имеется домен Glyco_hydro_3.
degapseq seed.txt seed.fasta
Далее запустить программу MAST в пакете EMBOSS.
emast -dfile seed.fasta meme.txt mastout.html
Выходной файл программы можно посмотреть здесь.
Всего в файле seed.fasta оказалось 39 последовательностей. Первый мотив встречается в 15 из них, второй - в 25, третий - в 38. Все мотивы присутствуют в 12 последовательностях, причем в 2х из них второй мотив встречается трижды в последовательности. Выравнивание Pfam соответсвует расположению мотивов в белке: сначала 2, потом 1, затем 3 мотивы.
 Вконтакте
Вконтакте allakarpova@kodomo.fbb.msu.ru
allakarpova@kodomo.fbb.msu.ru vseokeyboss@gmail.com
vseokeyboss@gmail.com