Описание выбранного домена в Pfam
Домен выбиралася следующим образом: в Pfam были найдены домены белка с 1 круса ybbD_BACSU, но оба домена представлены более чем в 3000 видах, поэтому Pfam отказывался строить деревья. Из архитектур этих доменов был выбран другой, с которым они сочетались.АС - PF03422.
ID - CBM_6.
Ссылка на страницу домена Pfam
Функция домена - связывание углеводородов (carbohydrate-binding module), обычно располагается в ферментах, осуществляющих превращения углеводородов.
Ссылка на доменную архитектуру с доменом CBM_6.
С Pfam было скачено выравнивание, в JalView оно было окрашено, одна последовательность проассоциирована с pdb структурой.
Из 402 возможных архитектур были выбраны две для работы:
- Glyco_hydro_43, CBM_6 (~360 последовательностей)
- Glyco_hydro_16, CBM_6 (~70 последовательностей)
Создание выборки
Для создания выборки из файла, содержащего информацию о всех доменах последовательностей на Uniprot, с помощью скрипта был составлен список всех последовательностей, содержащих домен CBM_6.
Для получения таксономии данных псоледовательностей, на Uniprot->Retrieve скачивались все записи с этими последовательностями, однако часть записей вообще не обнаружена на Uniprot, как например эта.
На основе частоты встречаемости доменов, архитектур, которые они составляют и представленности в таксонах потом и кровью были выбраны выше указанные доменные архитектуры. Для каждой архитектуры был выбран таксон Bacteria и подтаксоны Actinobacteria и Proteobacteria.
Таблицу excel можно скачать здесь.
| Архитектура | Число последовательностей | Bacteria | |
| Actinobacteria | Proteobacteria | ||
| CBM+GH43 | 20 | 13 | 7 |
| CBM+GH16 | 22 | 15 | 7 |
Анализ данных
Было построено выравнивание выбранных последовательностей, которое было скорректированно, были удалены выбивающиеся последовательности, концы выравнивания и окрашено ClustalX с порогом консервативности 30%. Одна последовательность, не входящая в выборку, была добавлена и проассоциирована с PDB структурой, но она слишком сильно отличается от выбранных последовательностей, чем мало помогает для анализа данных. Финальное выравнивание можно найти здесь.
Закодированы последовательности были следующим образом: NN_X_YYYYYY, где NN - число 43 или 16, соответствующее доменам Glyco_hydro_43 и Glyco_hydro_16, соответственно, Х - это А(Actinobacteria) или Р(Proteobacteria),YYYYYY - ID последовательности.
Последовательности также были разбиты на две группы согласно архитектурам. Можно сделать вывод, что в разных архитектурах разные а.о. домена CBM_6 более консервативны, разное количество консервативных остатков в целом, что можно объяснить разными функциями белков этих архитектур.
Построение филогенетического дерева
В программе MEGA методом Maximum Likelihood было построено дерево на основе полученного выравнивания из предыдущего задания. скобочная формула дерева.
С помощью сервера ITOL полученное дерево было раскрашенно.
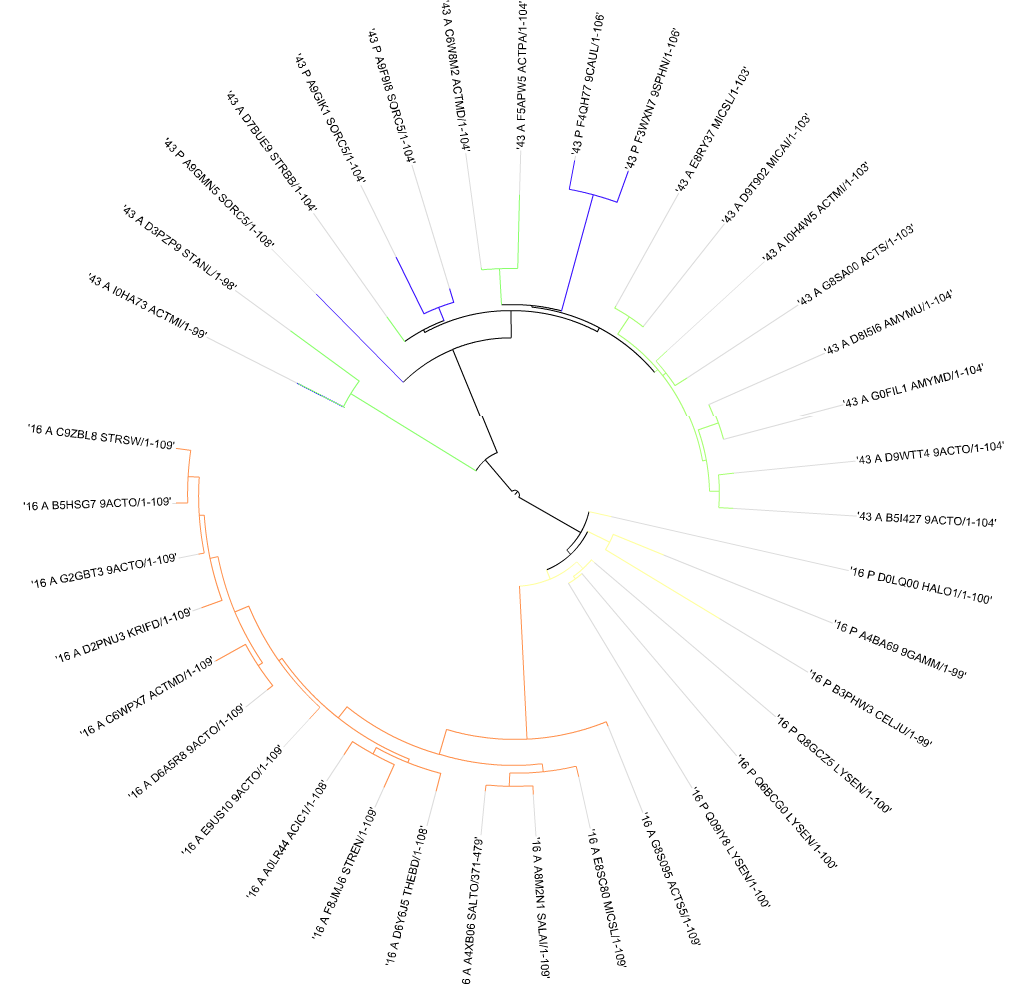
Рис.1. Дерево филогении бактерий на основе домена CBM_6.
Выводы
Так как на дереве четко разделены две архитектуры, то можно точно сказать, что архитектуры разделились раньше чем выбранные бактерии. Т.е. они раздились у какого-то общего предка.
Также внутри архитектуры с доменом Glyco_hydro_16 четко разнесены два бактериальных филума, что говорит о том, что сначала бактерии разделились на эти филумы, а потом уже активно эволюционировала архитектура с доменом Glyco_hydro_16.
Архитектура с Glyco_hydro_43 эволюционировала несколько сложнее, запутаннее и закономерность выявить сложно.
 Вконтакте
Вконтакте allakarpova@kodomo.fbb.msu.ru
allakarpova@kodomo.fbb.msu.ru vseokeyboss@gmail.com
vseokeyboss@gmail.com