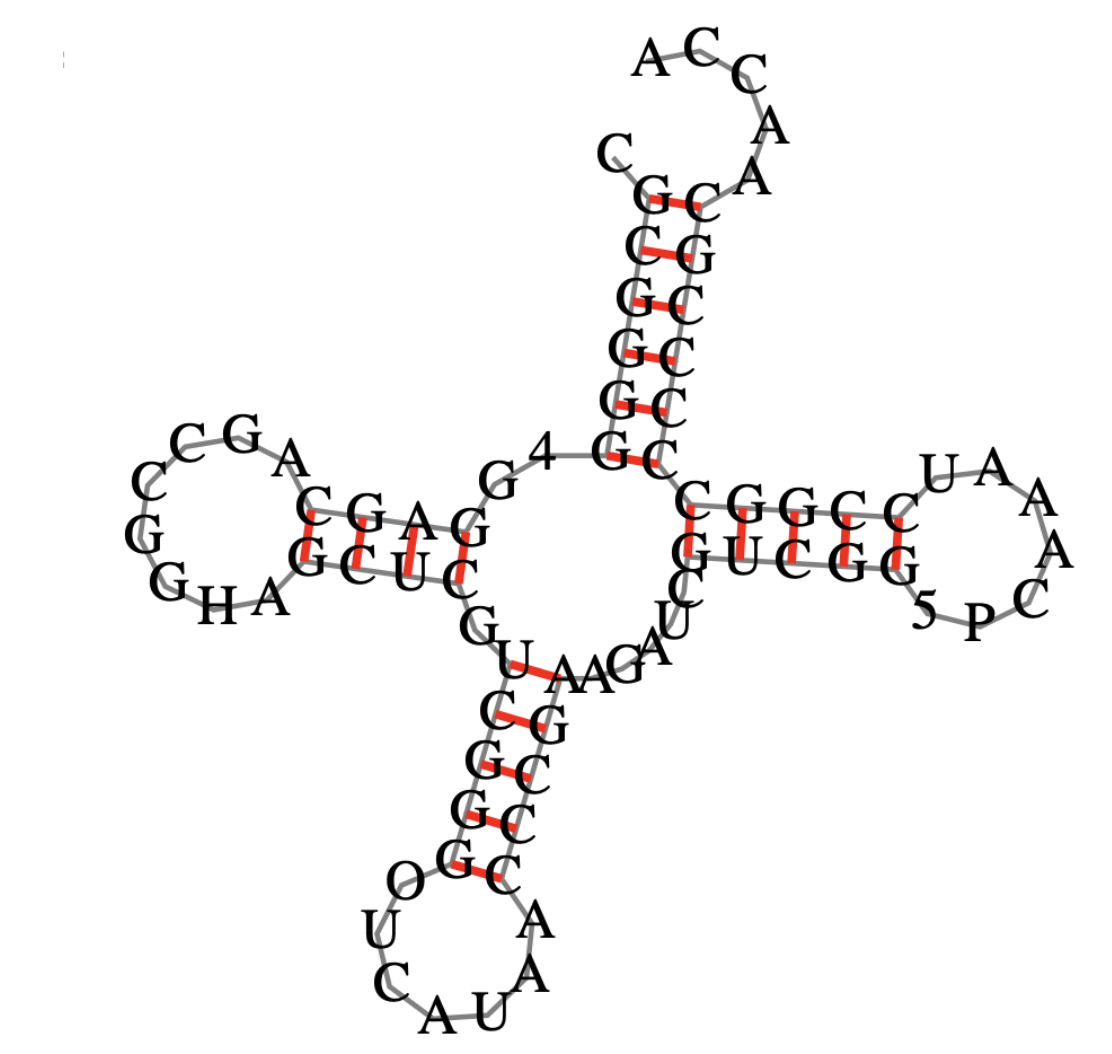
В этом задании нужно было предсказать вторичную структуру тРНК с помощью разных программ: find_pair, einverted, RNAfold (по алгоритму Зукера).
Мне досталась тРНК из файла 2fmt.pdb. Результаты представлены в таблице 1
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-2-7-3' 5'-67-72-3' 6 пар |
5'-1-6-3' 5'-66-71-3' смещение на 1 нуклеотид |
5'-2-7-3' 5'-67-72-3' 6/6 пар |
| D-стебель | 5'-10-13-3' 5'-23-26-3' 4 пары |
- | 5'-10-13-3' 5'-23-26-3' 4/4 |
| T-стебель | 5'-50-55-3' 5'-59-66-3' 6 пар |
- | 5'-50-55-3' 5'-62-66-3' 5/6 |
| Антикодоновый стебель | 5'-27-34-3' 5'-36-45-3' 8 пар |
5'-28-35-3' 5'-40-44-3' 6/8 |
5'-28-35-3' 5'-40-44-3' 6/8 |
| Общее число канонических пар нуклеотидов | 21 пара | 6 пар верно | 21 пара |

Команды для выбора множеств атомов в JMol:
select set1, chain R+Y and name O3'+O4'+O5'
select set2, chain R+Y and name OP1+OP2
select set3, chain R+Y and symbol N
Последовательные скрипты для демонстрации разных множеств:
1) hide all
show cartoon, chain M+R+Y
2) hide all
show sticks, chain R+Y
3) set sphere_scale, 0.5
show spheres, set1
4) hide spheres, set2
show spheres, set3
Используя скрипт, были найдены контакты атомов:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 19 | 70 | 89 |
| остатками фосфорной кислоты | 34 | 37 | 71 |
| остатками азотистых оснований со стороны большой бороздки | 8 | 15 | 23 |
| остатками азотистых оснований со стороны малой бороздки | 7 | 11 | 18 |
В основном белок взаимодействует с большой бороздкой и остатками дезоксирибозы.
Результат работы nucplot:
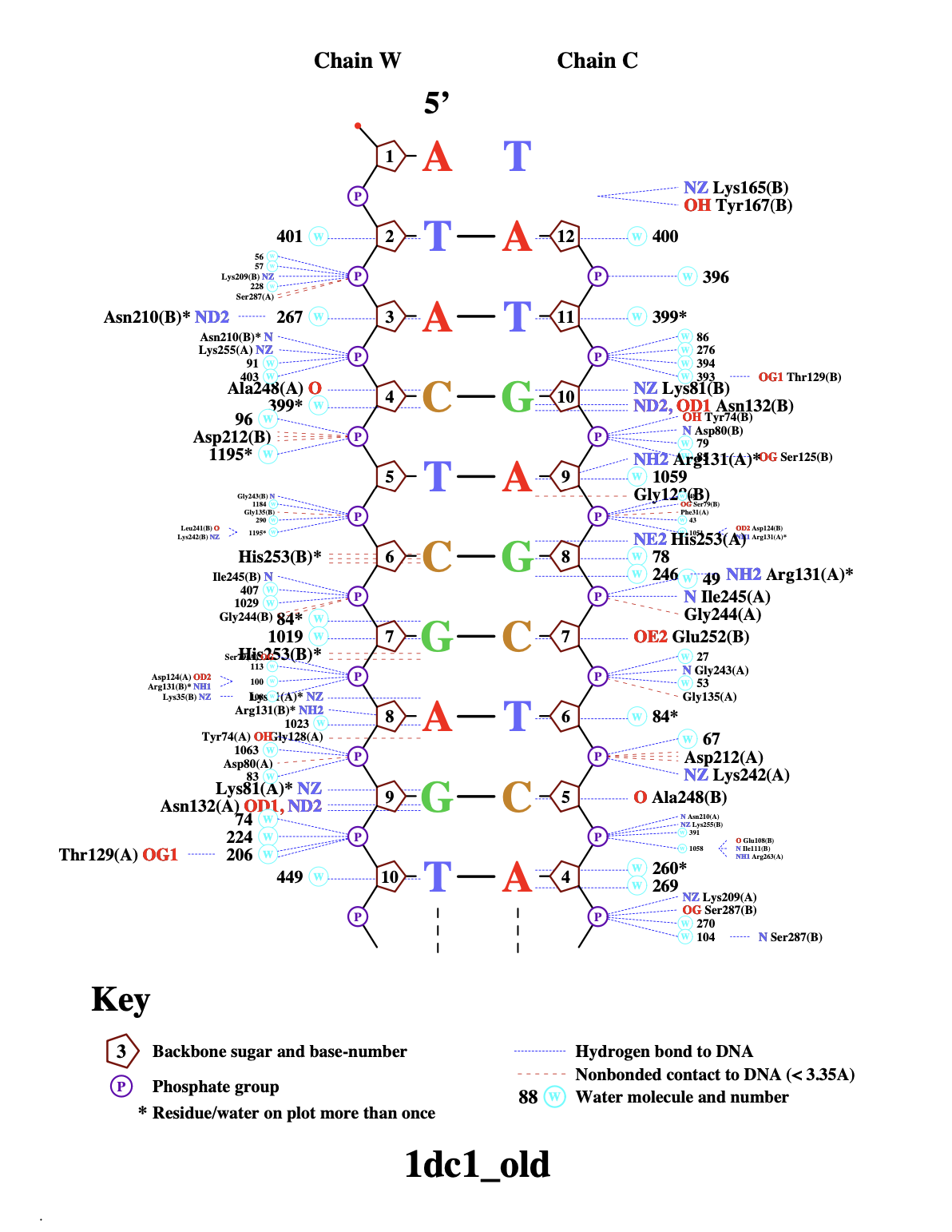
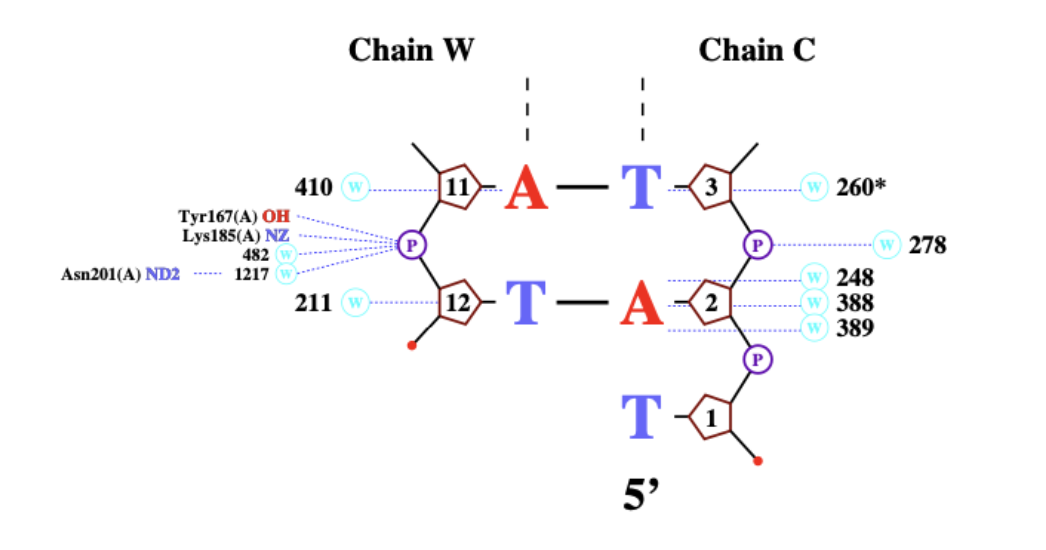
На этих рисунках видно, в каком месте какая аминокислота взаимодействует с сахарофосфатным остовом.
Судя по выводу nucplot, наиболее важна аминокислотa Arg131 - она образует шесть водородных связей
Логично, что это именно аргинин, потому что он положительно заряжен и очень хороший донор водородных связей. Его положительный заряд может взаимодействовать с отрицательно заряженным сахарофосфатным остовом.
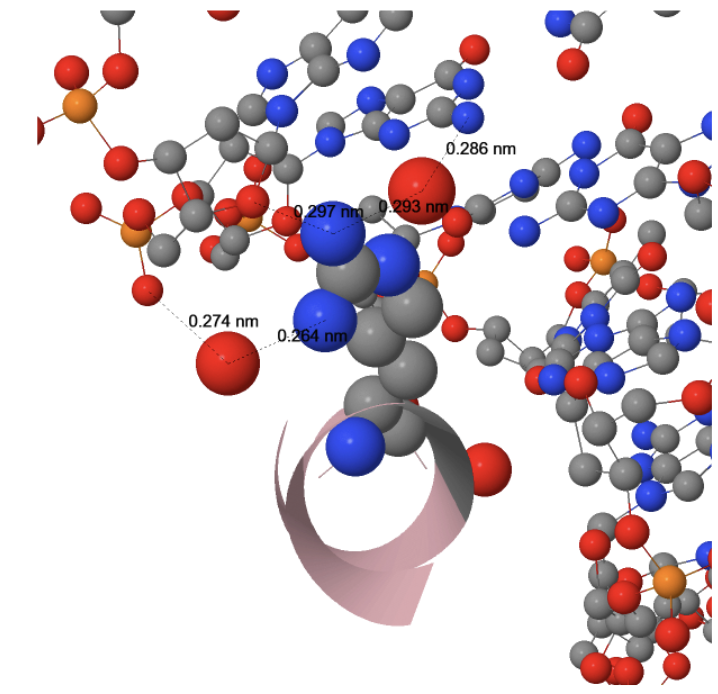
Фрагмент файла old_1dc1.bond:
**** Hydrogen Bonds ***************************
Donor Acceptor Distance
LYS B 209 NZ A W 3 O1P 2.48
ASN B 210 N C W 4 O1P 2.76
LYS A 255 NZ C W 4 O2P 2.73
C W 4 N4 ALA A 248 O 2.61
GLY B 243 N C W 6 O1P 2.67
ILE B 245 N G W 7 O1P 2.88
SER A 79 OG A W 8 O2P 2.69
ARG B 131 NH2 A W 8 O4* 2.86
LYS A 81 NZ A W 8 N7 2.99
TYR A 74 OH G W 9 O1P 2.64
LYS A 81 NZ G W 9 O6 2.84
G W 9 N2 ASN A 132 OD1 2.88
ASN A 132 ND2 G W 9 N3 2.89
LYS A 185 NZ T W 12 O1P 2.74
TYR A 167 OH T W 12 O2P 2.92
LYS A 209 NZ A C 4 O1P 2.76
SER B 287 OG A C 4 O1P 2.65
ASN A 210 N C C 5 O1P 2.81
LYS B 255 NZ C C 5 O2P 2.93
C C 5 N4 ALA B 248 O 2.73
LYS A 242 NZ T C 6 O1P 2.62
GLY A 243 N C C 7 O1P 2.70
C C 7 N4 GLU B 252 OE2 2.97
ILE A 245 N G C 8 O1P 2.90