Протеомы; частоты остатков в протеомах
Мы исследовали частоту аминокислотных остатков в протеомах бактерий Thermotoga maritima MSB8 и Escherichia coli К12
Информация о протеомах
Escherichia coli:
- Идентификатор протеома UP000000625
- Число последовательностей 4306
- Число остатков 1356086
Thermotoga maritima :
- Идентификатор протеома UP000008183
- Число последовательностей 1852
- Число остатков 582800
| Остаток | % в протеоме T.maritima | % в протеоме E.coli | Разность процентов |
| L | 10,04 | 10,67 | 0,63 |
| E | 8,92 | 5,76 | 3,16 |
| V | 8,61 | 7,07 | 1,54 |
| K | 7,62 | 4,41 | 3,21 |
| I | 7,18 | 6,01 | 1,17 |
| G | 6,91 | 7,37 | 0,46 |
| A | 5,85 | 9,52 | 3,67 |
| S | 5,65 | 5,8 | 0,15 |
| R | 5,54 | 5,51 | 0,03 |
| F | 5,19 | 3,89 | 1,3 |
| D | 4,97 | 5,15 | 0,18 |
| T | 4,53 | 5,4 | 0,87 |
| P | 3,99 | 4,46 | 0,47 |
| N | 3,61 | 3,95 | 0,34 |
| Y | 3,58 | 2,85 | 0,73 |
| M | 2,4 | 2,82 | 0,42 |
| Q | 2,01 | 4,44 | 2,43 |
| H | 1,59 | 2,27 | 0,68 |
| W | 1,1 | 1,53 | 0,43 |
| C | 0,71 | 1,16 | 0,45 |
| U | 0 | 0,0002 | 0,0002 |
*Для удобства данные округлены.
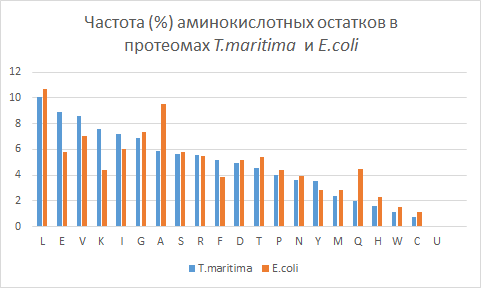
Данная диаграмма демонстрирует частоту аминокислотных остатков в протеомах T.maritima и E.coli. В протеоме T.maritima наиболее часто встречаются лейцин, глутаминовая кислота и валин. В протеоме E.coli преобладают лейцин, аланин, глицин и валин (почти равная доля). Как видно, лейцин и валин входят в список самых частовстречающихся аминокислот в протеомах обоих организмов. Список встречающихся редко аминокислот сходен в обоих протеомах: гистидин, триптофан, цистеин. Причем частота этих остатков в протеоме E.coli выше, чем в протеоме T.maritima. Кроме того, в протеоме E.coli встречается неканоническая аминокислота селеноцистеин (3 остатка). Наблюдается наибольшая относительная и, в то же время, абсолютная разность частоты встречаемости аланина (3,7%, 94950 остатков). Также довольно большая абсолютная разность встречаемости лейцина и глицина. Полученные данные представлены в таблице .
Выполняя данное задание, мы пользовались программой wordcount из пакета EMBOSS.
Она подсчитывает количество уникальных слов в последовательности. Программа запрашивает
у пользователя длину слова и название файла для записи выходных данных. Мы получили таблицу из двух колонок.
В первой колонке названия аминокислотных остатков, во второй – количество соответствующих остатков в протеоме.
Таблица отсортирована по убыванию значений во второй колонке.
Также для выполнения этого задания можно было пользоваться программой compseq.
Программа получает на вход, анализируемую последовательность (последовательности), в формате USA.
Также она запрашивает длину слова и имя выходного файла. Программа выдает таблицу из пяти колонок.
В первой колонке расположены в алфавитном порядке встретившиеся слова (аминокислотные остатки). Во второй колонке – общее количество данных аминокислотных остатков в протеоме.
Кроме того программа подсчитывает суммарное количество слов и встречаемость каждого слова, программа wordcount не делает этого. Данные о частоте аминокислотных остатков
в протеоме можно найти в третьей колонке.
В четвертой колонке так называемая предположительная частота (встречаемость). Она может быть рассчитана исходя из (неверного) предположения,
что все слова данной длины встречаются во входной последовательности с равной частотой. Так можно проанализировать протеом T.maritima.
Также можно использовать флаг "-infile" и ввести имя полученного ранее compseq-файла. Тогда программа будет считать предположительной частотой встречаемости
встречаемость слов (аминокислотных остатков) в данном файле. В пятой колонке данные об относительной частоте: фактическая частота слов делится на предположительную.
Для того, чтобы стало понятнее:
C помощью программы compseq мы получили данные о протеоме T.maritima. Сохранили их в файле Thema.compseq.
Далее хотим хотим узнать частоту аминокислотных остатков в протеоме E.coli и сравнить ее с оной в протеоме T.maritima. Для этого в командной строке пишем:
"compseq -infile Thema.compseq".
Остальные данные: входная последовательность, длина слова, имя выходного файла программа попросит ввести.
Далее программа обработает данные, и мы получим таблицу из пяти колонок: в первой название аминокислотных остатков, во второй – их количество, в третьей – встречаемость в протеоме E.coli.
В четвертой – "предположительная встречаемость" – частота этих остатков в протеоме T.maritima,
в пятой колонке относительная частота – данные из третьей колонки, поделенные на данные из четвертой колонки. Мы можем узнать, частота каких остатков в протеомах примерно одинаковая,
а каких аминокислотных остатков больше в протеоме одной из бактерий.
Таким образом, программа compseq оказывается более удобной для выполнения поставленной задачи.
