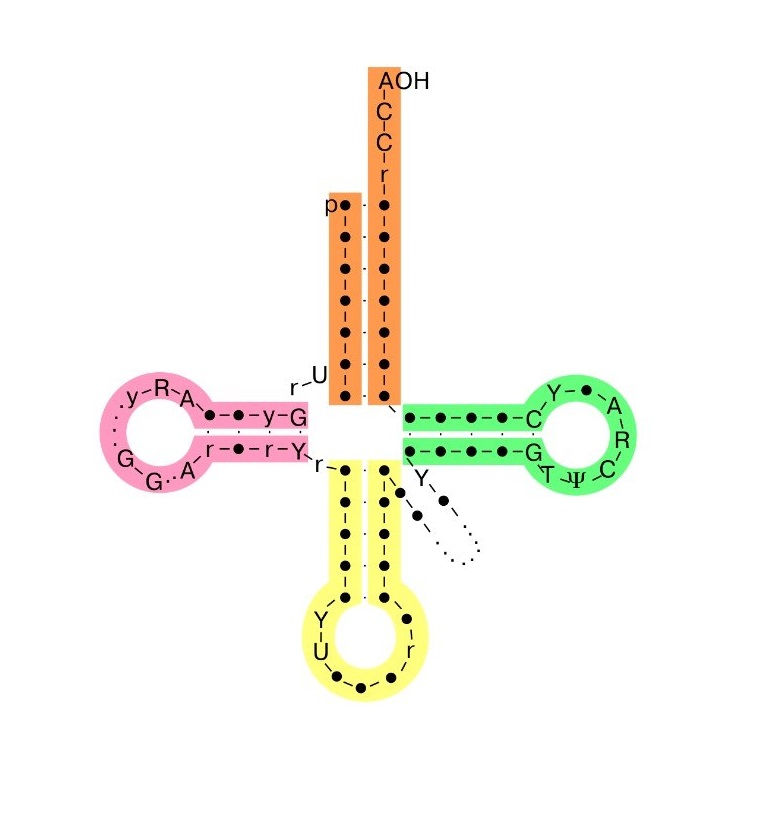
Предсказание вторичной структуры заданной тРНК
В предыдущем практикуме (pr2) с помощью программы find_pair мы нашли пары нуклеотидов, которые образуют стебли тРНК. Они представлены на рисунке 1.

Рисунок 1. Спаренные нуклеотиды тРНК
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Работаем с программой einverted, которая позволяет найти инвертированные участки в нуклеотидных последовательностях. Долго пытались подобрать параметры, так, кажется, нормально и не подобрали. Будем смотреть, что получилось.
Инвертированные повторы. Вывод программы.
Fasta-файл. Вывод программы einverted.
Структура тРНК предсказывается очень плохо, так и не получилось подобрать параметры так, чтобы предсказание было качественным. Лучшее, что получилось сделать -- предсказать 4 пары в акцепторном стебле.
Предсказание по алгоритму Зукера
cat trna.fasta | RNAfold --MEA > out.txt
Была использована программа RNAfold из пакета Viena Rna Package, которая реализует алгоритм Зукера. RNAfold принимает на вход последовательность РНК в формате fasta и рассчитывает вторичную структуру РНК с минимальной свободной энергией (mfe).
Выдача программы:
Файл
Точки обозначают неспаренные нуклеотиды, круглые скобки - спаренные.
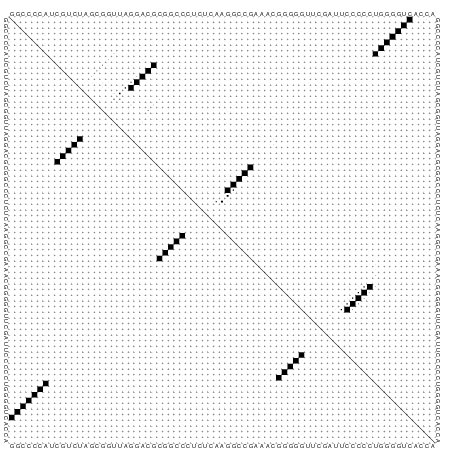
Рис.3. Точечный график, показывающий вероятность образования пар оснований, RNAfold
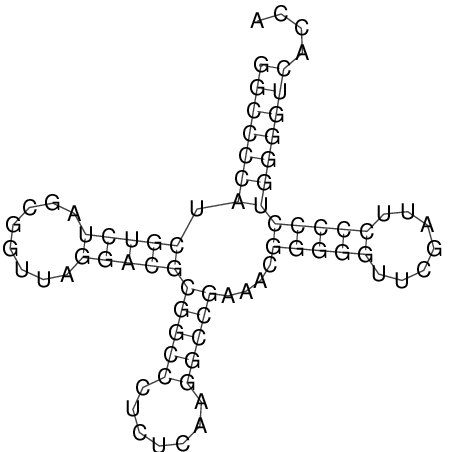
Рис.4. Предсказанная вторичная структура тРНК с минимальной свободной энергией, RNAfold
Программа с большой точностью предсказывает структуру тРНК. Предсказала по одной лишней паре в D- и Т- стеблях. Сравнение предсказания с реальной структурой представлено в таблице 1.
Таблица 1. Сранение реальной и предсказанной вторичной структуры тРНК из файла 1n78.pdb
| Участок структуры (расшифровку названий см. на рис. 2 в статье О.О.Фаворовой) | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-501-507-3'5'-566-572-3' Всего 7 пар |
предсказано 4 пары из 7 реальных | 7 из 7 |
| D-стебель | 5'-510-513-3'5'-522-525-3'Всего 4 пары | 0 | 5 (одна лишняя), т.е. 4 верно предсказанных |
| T-стебель | 5'-549-553-3'5'-561-565-3'Всего 5 пар | 0 | 5 из 5 |
| Антикодоновый стебель | 5'-538-544-3'5'-526-532-3' Всего 7 пар |
0 | 5 из 7 |
| Общее число канонических пар нуклеотидов | 23 | 4 | 21 |
ДНК-белковые комплексы
|
|
Описание ДНК-белковых контактов
Будем считать полярными атомы кислорода и азота, а неполярными -- атомы углерода, фосфора и серы.
Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å.
Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å.
Определим число контактов в ДНК-белковом комплексе.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 12 | 31 | 43 |
| остатками фосфорной кислоты | 21 | 7 | 28 |
| остатками азотистых оснований со стороны большой бороздки | 0 | 12 | 12 |
| остатками азотистых оснований со стороны малой бороздки | 11 | 2 | 13 |
Текст скрипта -- точнее набор использованных команд.
Больше всего полярных контактов с участием остатков фосфорной кислоты; на втором месте количество неполярных контактов с остатками 2'-дезоксирибозы. Контактов с участием остатков азотистых оснований со стороны малой бороздки было найдено 13, преобладают полярные контакты. С атомами большой бороздки 12 неполярных контактов. Можно заключить, что остов ДНК больше склонен к образованию связей, чем остатки азотистых оснований.
Схема ДНК-белковых контактов
Получили популярную схему ДНК-белковых контактов с помощью программы nucplot
remediator --old 1i3j.pdb >1i3j_old.pdb
nucplot 1i3j_old.pdb
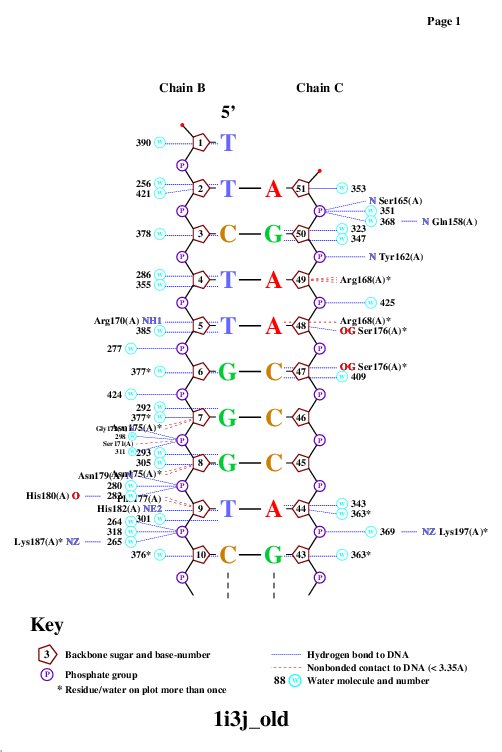
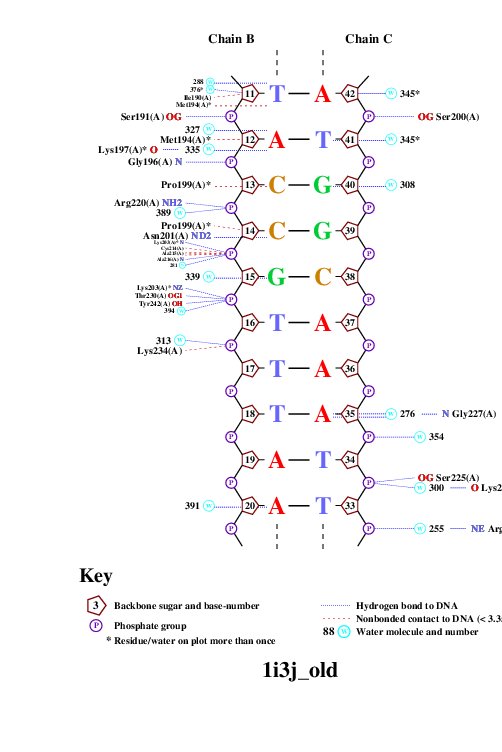
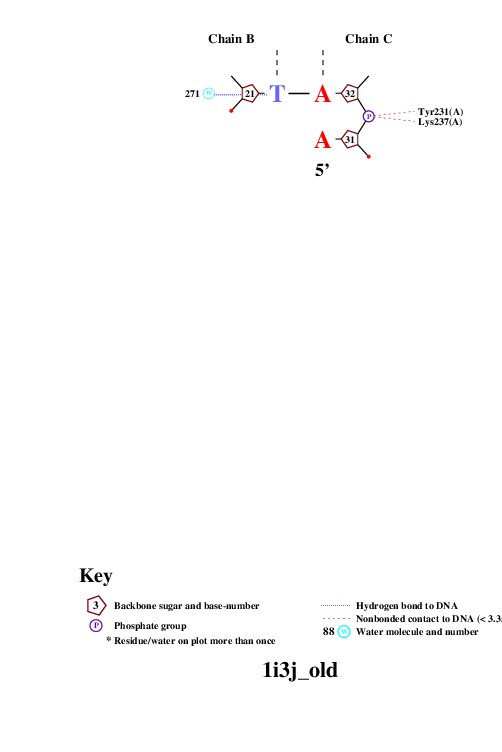
Наибольшее число контактов с ДНК имеют Arg168 и Asn175 (по 4 контакта). При этом Arg168 имеет контакт с азотистым основанием.
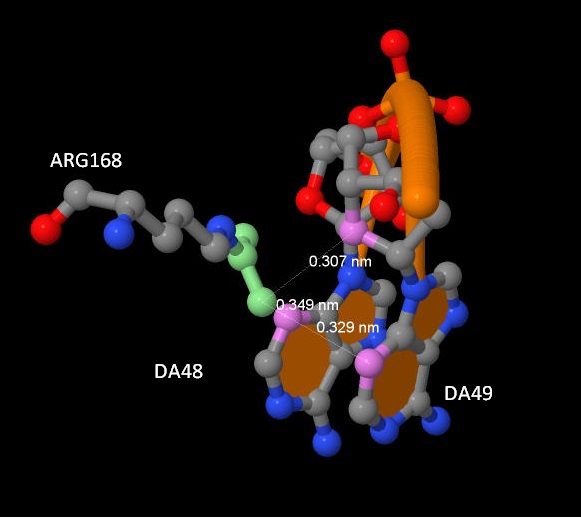
Arg168 и нуклеотиды, с которыми он контактирует. Раскраска по атомам. Зеленым атомы белка, фиолетовым атомы ДНК, находящиеся на расстоянии 3.5А друг от друга.
Аминокислотный остаток, наиболее важный для распознавания последовательности ДНК -- SER176, т.к. образует водородные связи с двумя нуклеотидами: DC47 и DA48
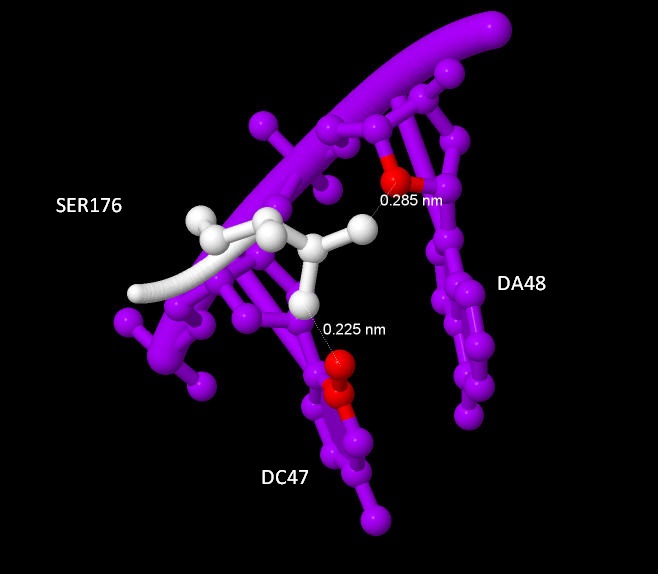
Ser176 и нуклеотиды, с которыми он контактирует. Показаны водородные связи.