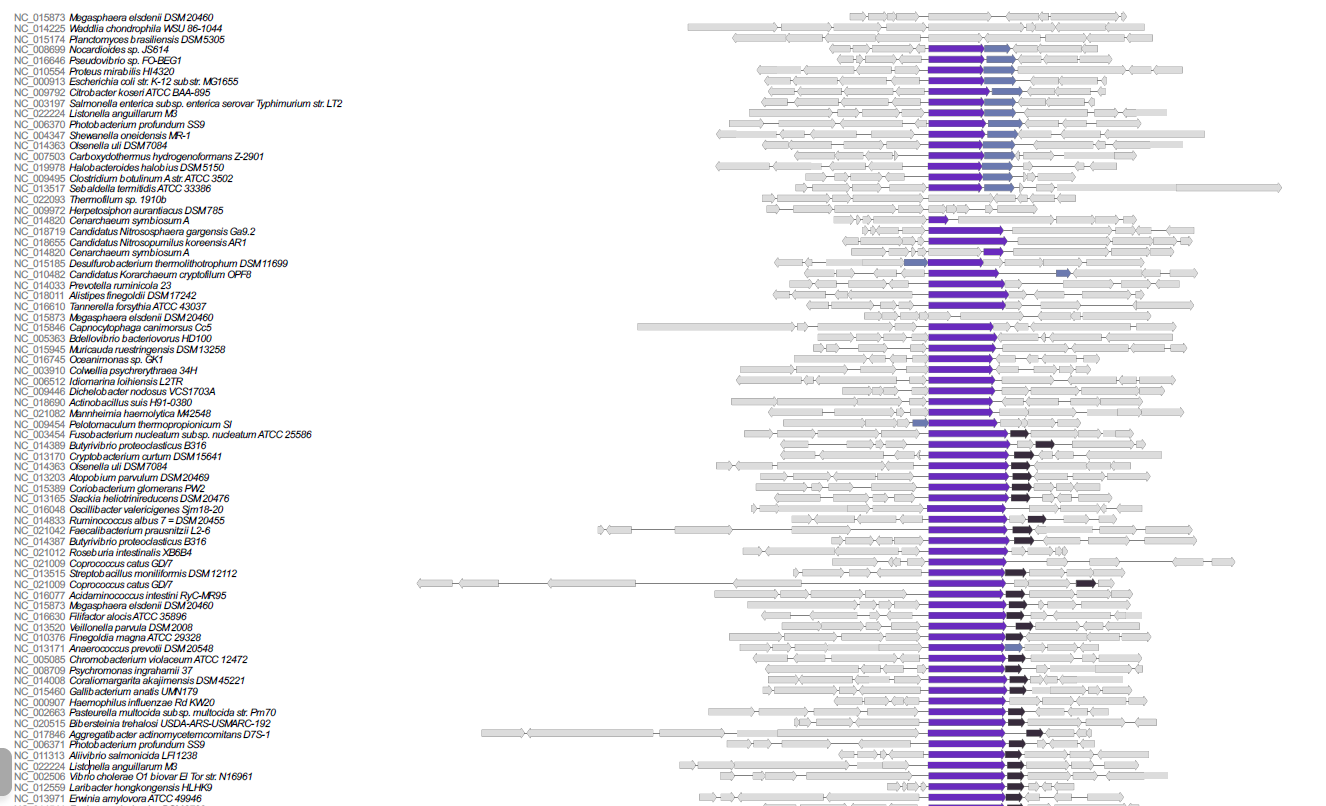
Геномное окружение. База данных GO
В первом семестре мне был выдан белок NP_228195.1 из генома Thermotoga maritima MSB8.
>NP_228195.1 Thermotoga maritima MSB8 anaerobic ribonucleoside triphosphate reductase
MKVQYSFEREFEELMSDLLSKYGYEMFQMDGLGDQLDVVKFTEDFVRRGIIESTIDANAN
VRVTNISTYFIEISKPHTYLYSLYRIWQKMKEMFGKGVADEFVEAQINGAVYLHDRHHAA
LMPYCFAYTLKPIVEKGLPFIKTIKSEPAKHLSTFIQHVIQFVMFASNQSSGAVGLPDFF
VWMWYFVKKDLKEGIIPRDKLDWYIEQHFQILTYSLNQPIRTTQSPYTNFTYLDRNYIKA
IFEGERYPDGSLITDHVEDIIALQKHYWEWVSRERERQMFTFPVLTASLLYKDGKFLDED
SARFINKINMKWQDTNWYISDSIDAVASCCRLTSSTQTLKKFSLSSEEEEKLKGRMNSIG
GSDLNIGSFKVITVNLPRIALESGGDREKYLQILRHRVQLIKKALAAVREIIKERISEGL
LPLYENGLMLLNRQYGTIGVTGVWESASIMGLTTEDIDGLKYTEEGEVFVDNVLDTIREE
AEKGYHEYGFTFNIEQVPAEKAAVTLAQKDRFLFGEKQPFEIYSNQWVPLMANTDVLNRI
RYSGKWDKKVSGGAILHINLGESFKTEEESFNMVKMIADMGVMYFAFNTKISVCEDGHAF
YGERCPVCGKAKVDEYMRIVGYLVPVSAFNKERREIEYPRRQFYDSLTIRR
Получение информации о КОГе, к которому относится белок
С помощью сервиса
CDD (Conserved Domain Database) для данного белка был получен список хитов (Рис. 1),
из которого нужно было выбрать те, которые относят белок к тому или иному КОГу.

Рис. 1. Список хитов для белка NP_228195.1
В списке оказался единственный хит относящий белок к какому-либо КОГу, информация о нем приведена в таблице 1.
Таблица 1. Информация о КОГе
| ID |
COG1328 |
| e-value для отнесения белка к данному КОГу |
0e+00 |
| Координаты |
1-647 |
| Название КОГа и функциональная категория |
Anaerobic ribonucleoside-triphosphate reductase [Nucleotide transport and metabolism] |
| Анаэробная рибонуклеозид-трифосфат редуктаза (транспорт и метаболиз нуклеотидов |
Визуализация геномного окружения
Получили изображение геномного окружения для обнаруженного
КОГа с помощью STRING
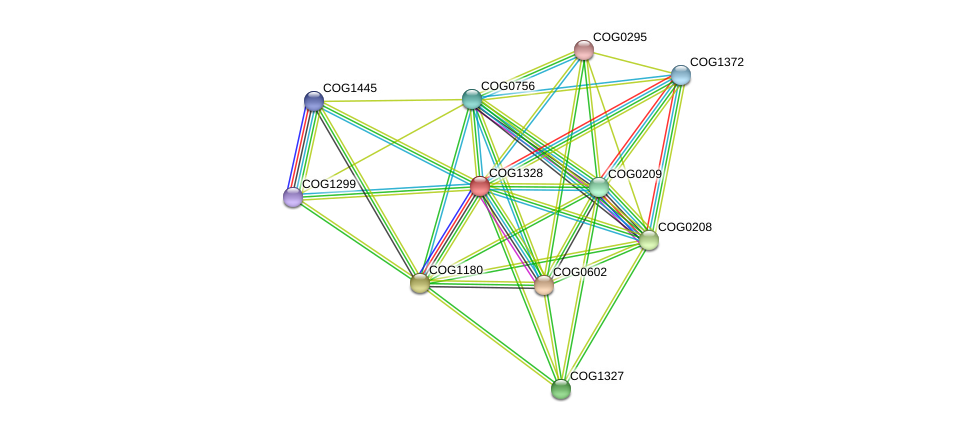
Рис. 2. Геномное окружение COG1328, программа STRING
Узлы графа символизируют белки. Если для даннго белка известна пространственная структура,
то узел отображается более крупно, и внутри него расположена соответствующая 3D структура.
Ребра отражают белок-белковые связи. Их цвета несут определенный смысл:
фиолетовый означает экспериментально доказанные взаимосвязи, голубой — взаимосвязи, информация о которых получена из курируемых баз данных,
зеленый обозначает соседство в геноме, синий — совместную встречаемость,
красный — слияние генов. Салатовый означает совместное упоминание данных белков в Pub-Med Abstract, черный — коэкспрессию, а светло-синий — гомологию.
Рис. 3. Значения цветов ребер
Выдача сервиса включает таблицу со всеми взаимосвязями КОГа с остальными КОГами на графе, а также score данных взаимосвязей.

Рис. 4. Взаимосвязи COG1328 c другими КОГами
Также мы воспользовались сервисом COGNAT.
Выдача COGNAT в формате pdf.
COG1328 имеет консервативное геномное окружение, в которое входят COG1180 (Pyruvate-formate lyase-activating enzyme) и
COG0602 (Organic radical activating enzyme).
Согласно выдаче STRING, эти КОГи расположены рядом и вместе экспрессируются.
Следует обратить внимание, что, согласно выдаче COGNAT, в геномах бактерий с COG1328 соседствует либо COG1180, либо COG0602, все три КОГа
одновременно не встречаются.
Отнесение белка NP_228195.1 из генома Thermotoga maritima MSB8 к терминам GO
С помощью инструмента AmiGO поиком BLAST в базе данных GO нужно было найти белок, наиболее похожий на NP_228195.1
Наилучшей находкой был неохарактеризованный белок CPS_0092 из
Colwellia psychrerythraea 34H , E-value находки 3.4e-58. Выравнивание представлено ниже.
28% идентичных позиций и 51% сходных позиций — приемлимое сходство.
>UNIPROTKB|Q48AQ3 [details] [associations]
symbol:CPS_0092 "Putative uncharacterized protein"
species:167879 "Colwellia psychrerythraea 34H" [GO:0003674
"molecular_function" evidence=ND] [GO:0005575 "cellular_component"
evidence=ND] [GO:0008150 "biological_process" evidence=ND]
EMBL:CP000083 GenomeReviews:CP000083_GR eggNOG:COG1328 KO:K00527
RefSeq:YP_266860.1 STRING:Q48AQ3 GeneID:3519225 KEGG:cps:CPS_0092
PATRIC:21463603 HOGENOM:HOG000249497 OMA:IAYNYRI
ProtClustDB:PRK08271 BioCyc:CPSY167879:GI48-195-MONOMER
InterPro:IPR014192 TIGRFAMs:TIGR02827 Uniprot:Q48AQ3
Length = 593
Score = 369 (135.0 bits), Expect = 3.4e-58, Sum P(2) = 3.4e-58
Identities = 100/349 (28%), Positives = 178/349 (51%)
Query: 53 STIDANANVRVTNISTYFIEISKPHTYLYSLYRIWQKMKEMFGKGVADEFVEAQINGAVY 112
S +DANANV NI+T E+ K + + K+++ F +A E++ + +Y
Sbjct: 29 SKMDANANVSHKNIATLEAELLKDCFVQINRALVHDKIRDTFDADLAKEYLRQIEDHEIY 88
Query: 113 LHDRHHAALMPYCFAYTLKPIVEKGLPFIKTIKSEPAKHLSTFIQHVIQFVMFASNQSSG 172
+HD +L PYC + ++ P + GL + +S+ KHL +F + + S Q +G
Sbjct: 89 VHDE--TSLKPYCTSISMYPFLLDGLTKLGG-ESKAPKHLESFCGSFVNLIFAISAQFAG 145
Query: 173 AVGLPDFFVWMWYFVKKDLKEGIIPRDKLDWYIEQHFQILTYSLNQPI--RTTQSPYTNF 230
AV +F + YF + + + K + IE H Q + Y++NQP R QS + N
Sbjct: 146 AVATVEFLTYFDYFARLEYGNDYLSTHKSE--IENHLQHVVYAINQPAAARGYQSVFWNI 203
Query: 231 TYLDRNYIKAIFEGERYPDGSLITDHVEDIIALQKHYWEWVSRERERQMFTFPVLTASLL 290
+ D++Y ++F +PD S + + + LQ+ + W+++ERE+ + TFPV+TA++L
Sbjct: 204 SLFDQHYFASMFGEFVFPDFS--KPNWQTVDQLQQFFLGWINKEREQAVLTFPVVTAAML 261
Query: 291 YKDGKFLDEDSARFINKINMKWQDTNWYISDSIDAVASCCRLTSSTQTXXXXXXXXXXXX 350
++G+ D D AR + + + Y+SDS D++ASCCRL +
Sbjct: 262 TENGQCKDLDFARKLAQQKAEGNSFFIYLSDSADSLASCCRLRNEISDNTFSY------- 314
Query: 351 XXXGRMNSIGGSDLNIGSFKVITVNLPRIALESGGDREKYLQ-ILRHRV 398
++G + GS VITVN+ R+ ++ G D + ++ I +++V
Sbjct: 315 -------TLGAGGVATGSINVITVNMNRL-VQDGRDLKTEIEKIQKYQV 355
Оказалось [1], что данный белок ассоциирован со слишком общими терминами:
"биологический процесс", "компонент клетки", "молекулярная функция".
Поэтому я взяла другую находку, с меньшим сходством.
Я взяла белок BA_3663 — анаэробную рибонуклеозид-трифосфат редуктазу из организма Bacillus anthracis str. Ames.
E-value этой находки 8.2e-13, 25% идентичных позиций, 45% сходных. Выбранный белок выполняет ту же самую функцию, что и мой белок.
Выравнивание приведено ниже.
>TIGR_CMR|BA_3663 [details] [associations]
symbol:BA_3663 "anaerobic ribonucleoside-triphosphate
reductase, putative" species:198094 "Bacillus anthracis str. Ames"
[GO:0008998 "ribonucleoside-triphosphate reductase activity"
evidence=ISS] [GO:0009265 "2'-deoxyribonucleotide biosynthetic
process" evidence=ISS] InterPro:IPR012833 EMBL:AE016879
EMBL:AE017334 EMBL:AE017225 GenomeReviews:AE016879_GR
GenomeReviews:AE017225_GR GenomeReviews:AE017334_GR GO:GO:0006260
KO:K00527 TIGRFAMs:TIGR02487 GO:GO:0008998 HOGENOM:HOG000222475
RefSeq:NP_845927.1 RefSeq:YP_020298.1 RefSeq:YP_029654.1
HSSP:P07071 ProteinModelPortal:Q81Y93 DNASU:1086039
EnsemblBacteria:EBBACT00000010561 EnsemblBacteria:EBBACT00000017743
EnsemblBacteria:EBBACT00000020912 GeneID:1086039 GeneID:2816693
GeneID:2848849 KEGG:ban:BA_3663 KEGG:bar:GBAA_3663 KEGG:bat:BAS3398
OMA:NNTRGEE ProtClustDB:PRK14704
BioCyc:BANT260799:GJAJ-3459-MONOMER
BioCyc:BANT261594:GJ7F-3569-MONOMER Uniprot:Q81Y93
Length = 618
Score = 211 (79.3 bits), Expect = 8.2e-13, Sum P(2) = 8.2e-13
Identities = 78/308 (25%), Positives = 140/308 (45%)
Query: 356 MNSIGGSDLNIG--SFKVITVNLPRIALESGGDREKYLQILRHRVQL-IKKALAAVREII 412
M++I G + IG + ++NL ++AL SG +E + + L + + L IK+ L
Sbjct: 309 MSNIHGEETAIGRGNLSFTSINLVKLALISGS-KEAFYEALNYYLDLGIKQLLERFEYQC 367
Query: 413 KERISEGLLPLYENGLMLLNRQYGTI-GVTGVWESASI----MGLTT--EDIDGLKYTEE 465
+R + LY G+ + V + + ++ +GL + G + E+
Sbjct: 368 TKRARDFRF-LYSQGVWRGGEKLQPEDSVASILKQGTLSLGFIGLAECLVALTGKHHGED 426
Query: 466 GEVFV--DNVLDTIREEAEKGYHEYGFTFNIEQVPAEKAAVTLAQKDRFLFGEKQPF--- 520
E + ++ +R+ +K E+ F++ PAE + +KDR FG
Sbjct: 427 EESWKLGYEIISFMRDRMDKATEEHELNFSVIATPAEGLSGKFVKKDREEFGVISGITNH 486
Query: 521 EIYSNQW-VPLMANTDVLNRIRYSGKWDKKVSGGAILHINL-GESFKTEEESFNMVKMIA 578
Y+N + +P+ N +N+IR G + +GG I +I L G + ++ +V+ +A
Sbjct: 487 NYYTNSFHIPVYYNMQAINKIRLEGPFHALCNGGHITYIELDGAAMHNKKALKQIVQAMA 546
Query: 579 DMGVMYFAFNTKISVCE--DGHAFYGERCPVCG---KAKVDEYMRIVGYLV-PVSAFNKE 632
+ GV Y + N + C+ H G CP CG +A ++ RI GYLV +S +N
Sbjct: 547 EHGVGYGSINHPVDRCKCCSYHGVIGNECPSCGNEDEANIERIRRITGYLVGDMSKWNSA 606
Query: 633 RREIEYPR 640
+R E R
Sbjct: 607 KRSEEMDR 614
Score = 38 (18.4 bits), Expect = 8.2e-13, Sum P(2) = 8.2e-13
Identities = 10/19 (52%), Positives = 13/19 (68%)
Query: 187 VKKDLKEGII-PRDKLDWY 204
VKK + E I+ P D LD+Y
Sbjct: 63 VKKAINENILYPHD-LDFY 80
На странице найденного белка по ссылке N term associations была получена информация о терминах GO, отнесенных к данному белку.
Их оказалось 2. Найденные термины представлены в таблице 2.
Таблица 2. Термины GO, отнесенные к белку с идентификатором BA_3663
| Аспект |
Идентификатор GO |
Название термина |
Перевод названия термина |
Код типа достоверности |
| Биологический процесс (Biological process) |
GO:0009265 |
2'-deoxyribonucleotide biosynthetic |
Биосинтез 2'-дезоксирибонуклеотидов |
ISS |
| Молекулярная функция (Molecular function) |
GO:0008998 |
ribonucleoside-triphosphate reductase |
рибонуклеозид-трифосфат редуктаза |
ISS |
Для обоих терминов GO код типа доставерности ISS. Информация о данном коде представлена в таблице 3.
Таблица 3. Описание кодов достоверности, использованных в Таблице 2.
| Код типа достоверности |
Расшифровка кода типа достоверности |
Объяснение |
| ISS |
Inferred from Sequence or Structural Similarity
(Основан на сходстве последовательностей или структурном сходстве) |
Используется, когда аннотация проводилась на основании анализа последовательностей, причем данный анализ был проверен вручную. |