Реконструкция эволюции доменной архитектуры
Построение выравнивания представителей домена Pfam белков с разной доменной архитектурой
Я выбрала домен HWE_HK (ID: HWE_HK; AC: PF07536) HWE гистидиновая киназа (HWE histidine kinase).
HWE домен обнаруживается в составе двухкомпонентной системы, состоящей из гистидиновой киназы и родственного регуляторного белка. Отличительная черта -- присутствие высококонсервативного остатка Гистидина в киназном домене.В выбранном семействе 1627 последоватальностей, которые принадлежат 377 видам. Домен входит в 228 архитектур (ссылка). Для 4 последовательностей имеется 3D структура.
Для дальнейшей работы я выбрала 2 архитектуры. Информация о них представлена в таблице 1.
Таблица 1. Информация о выбранных доменных архитектурах
| Домены, входящие в архитектуру | Число последовательностей | Изображение доменной архитектуры |
|---|---|---|
| HWE_HK | 269 |  |
| PAS_4, HWE_HK | 113 |  |
A Per-Arnt-Sim (PAS) domain ( PAS_4 )-- это белковый домен, обнаруживаемый во всех царствах. PAS играет роль молекулярной "липучки". Этот мотив вовлечен в белок-белковые взаимодействия и взаимодействие белков с малыми молекулами. Также это типичный мотив сигнальных белков,в которых функционирует в качестве сенсора.
С помощью Jalview построили выравнивание доменных участков всех белков, содержащих домен HWE_HK (окраска ClustalX порог консервативности 30%).
Также была добавили 3D структура одного домена. Для этого последовательность LOVHK_BRUA2 была связана с PDB ID 5EPV.
Соответствие между Uniprot ID и PDB кодами можно было наити в разделе Structures.
Ссылка на Jalview-проект: pr11_pfam.jvp .
Для получения информации об архитектуре всех последовательностей, содеражщих выбранный домен, был использован скрипт
swisspfam-to-xls.py, который преобразует информацию о доменах в последовательности из файла swisspfam в таблицу для Excel.
python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -p PF07536 -o PF07536.xls
В результате получили таблицу PF07536.xls
Добавили в таблицу таксономию при помощи VLOOKUP (ВПР).
Сама таксономия была получена при помощи скрипта. Для этого была создана таблица с информацией о таксономии из Uniprot:
python uniprot-to-taxonomy.py -i uniprot.txt -o taxonomy.xls
Получили файл taxonomy.xls
Для дальнейшей работы выбрали таксон: Класс Alphaproteobacteria и подтаксоны: порядки Rhizobiales и Rhodobacterales.
python filter-alignment.py -i align_pr11.mfa -m ids -o my_align.fasta -a "/"
Итоговая таблица:
table.xlsx
Построение филогенетического дерева домена
Bспользуя полученное выше выравнивание постороили филогенетическое дерево методом Neighbor-Joining. Для удобства ввели обозначения -- расшифровка в таблице 2.
Таблица 2. Используемые обозначения:
Обозначение |
Расшифровка | |
|---|---|---|
| Подтаксон | Доменная архитектура | |
| H_rhiz | Rhizobiales | HWE_HK |
| P_rhiz | Rhizobiales | PAS_4 &HWE_HK |
| H_rho | Rhodobacterales | HWE_HK |
| P_rho | Rhodobacterales | PAS_4 & HWE_HK |
Скобочная формула дерева tree.txt

Рис. 1. Дерево, построенное методом Neighbor-Joining в программе MEGA
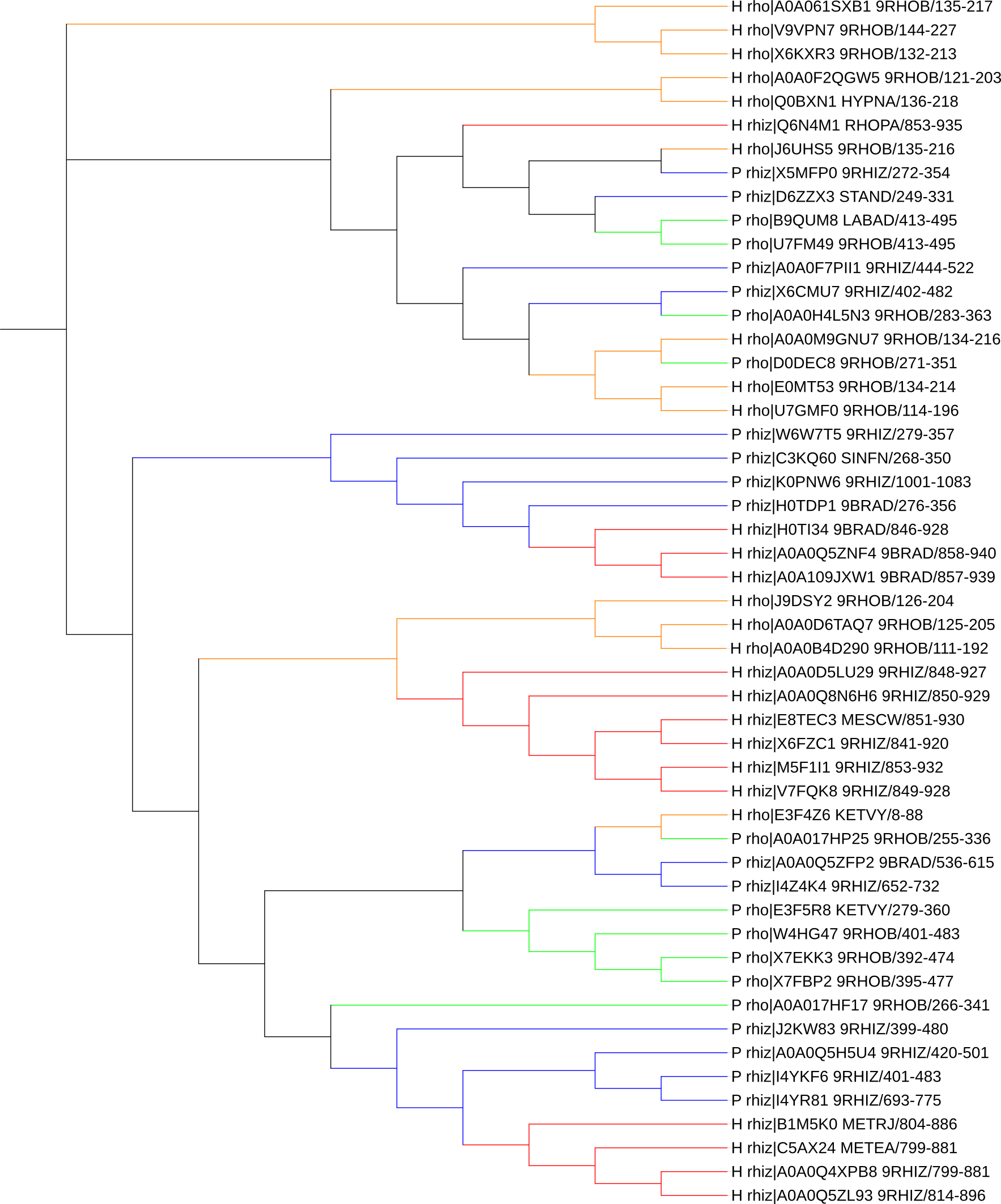
Рис. 2. Дерево, раскрашеное с помощью ITOL
Дерево получилось довольно странным, некоторые последовательности выбиваются из клад. В целом нет групировки клад по таксонам или по типам архитектуры, только на отдельных ветвях.
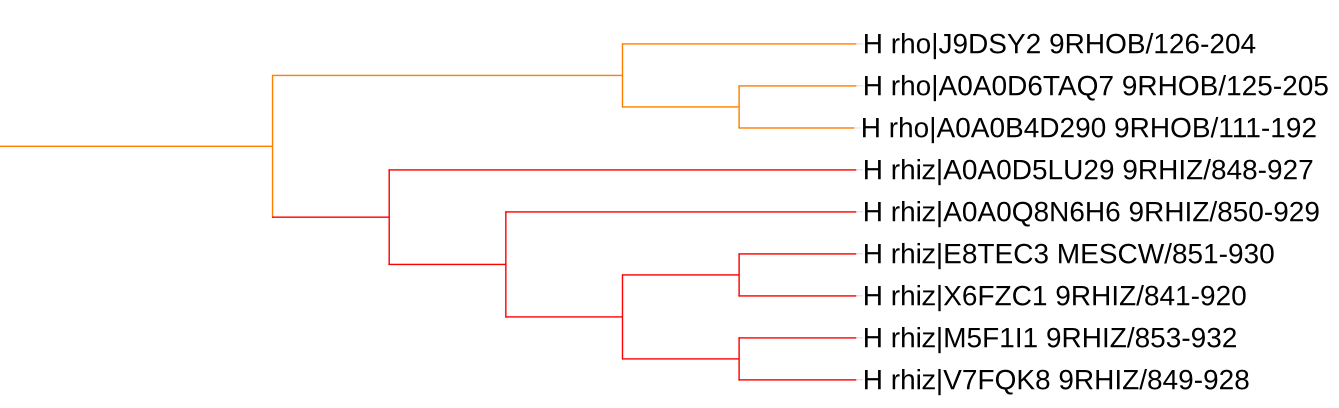
Рис. 3. Ветвь дерева, ветвление в соответствии с таксономией.
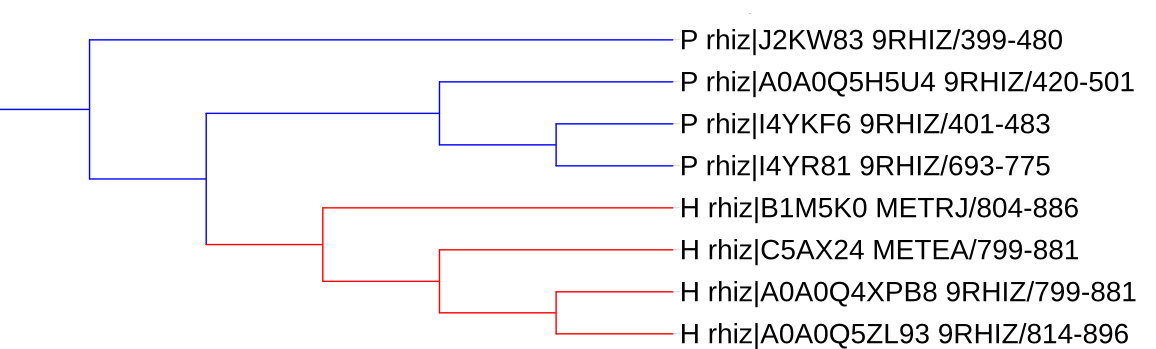
Рис. 4. Ветвь дерева, ветвление в соответствии типам архитектур
Возможно, выбор таксонов для работы оказался неудачным -- я выбрала системетические группы класс и порядки.
