Реконструкция филогении по нуклеотидным последовательностям. Паралоги.
Для выполнения заданий данного практикума были скачаны последователньости 16S рибосомной РНК для бактерий из предыдущего практикума. Данные последвоательнгости были выровнены в JalView с помощью алгоритма Muscler with Defaults и по полученному выравниванию, с помощью программы MTGA, методом максимального правдободобия было построено дерево, предсталенно на рисунке 1.
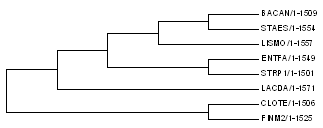 Рис. 1. Дерево, полученно по выравниванию последвоательностей 16S рибосомной РНК
Рис. 1. Дерево, полученно по выравниванию последвоательностей 16S рибосомной РНК
Это дерево не абсолютно идентично "правильному" дереву из предыдущего практикума, есть одно отличие: нет ветви {BACAN, LISMO}, но появилась ветвь {BACAN, STAES}. По сравнению с предсказанием пло белковым последовательностям из предыдущего практикума, предсказание по 16S рибосомальной РНК лучше.
Для выполнения следующего задания была создана база данных из протеомов данных бактерий типа "prot". С помощью команды, показанной ниже, был проведён поиск гомологов данной в задании последовательности белка из генома бактерии Bacillus subtilis
blastp -query P50866.fasta -db al.fasta -evalue 0.001 -outfmt 7 -word_size 3 -out homol.fasta
Полученные последовательности гомологов можно скачать по следующей ссылке. Эти последовательности были выравнены с помощью алгоритма Muscle with Defaults, по полученному выравниванию было построено дерево, показанное на рисунке 2.
 Рис. 2. Дерево для гомологов белка P50866 из геномов выбранных бактерий
Рис. 2. Дерево для гомологов белка P50866 из геномов выбранных бактерий
Ортологами называют гомологичные белки, разошедшиеся в результате видообразования. Примером ортологиченых белков, например, является ветвь, выделенная красным, на рисунке 2.
Паралоги - это гомологичные белки, принадлежащие одному организму, примерами таких белкв, например, являются: Q898D1, QQ891B9, Q899H3 (выделены зелёным цветом на рисунке 2). принадлежащие Clostridium tetani.
Файл проекта в JalView, содержащего выравнивания из данного рпатикума можно скачтаь пос ледующей ссылке.