 |
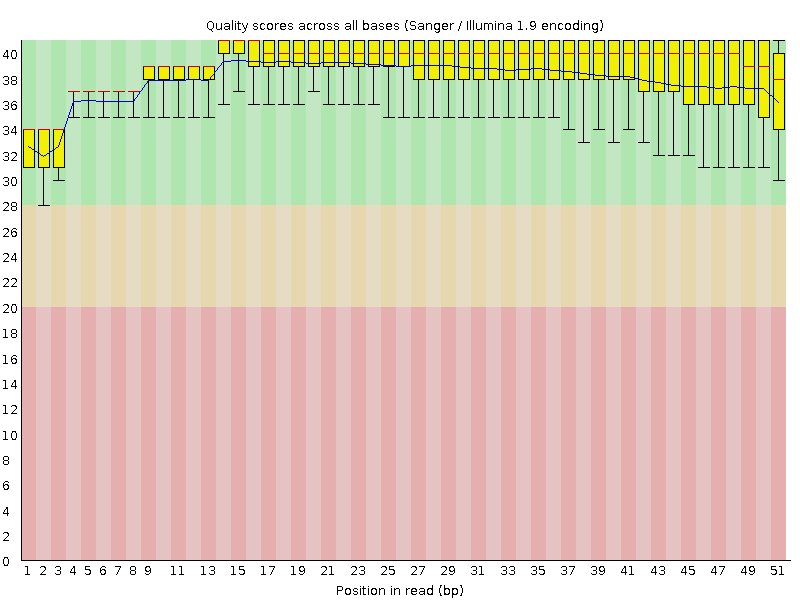
| Кол-во ридов - 3565, длина 41-51, %GC - 55% |
3565 reads; of these:
3565 (100.00%) were unpaired; of these:
47 (1.32%) aligned 0 times
3507 (98.37%) aligned exactly 1 time
11 (0.31%) aligned >1 times
98.68% overall alignment rate
| координаты | длина | направление цепи | описание | ген | число ридов, в которых встречается |
| 2447858 - 2447961 | 103 | - | protein_coding | RP4-734P14.4 | 8 |
| 2443608 - 2443683 | 75 | - | snoRNA | SNORD119 | 3 |
| 2442736 - 2443264 | 529 | - | protein_coding | SNRPB | 79 |