Реконструкция деревьев по нуклеотидным последовательностям
Построение дерева по нуклеотидным последовательностям
Для работы были получены последовательности в формате fasta 16S рибосомальной РНК бактерий. Особенности 16S РНК показаны ниже в таблице.
| Мнемоника | AC записи EMBL | Координаты РНК | Цепь |
| BACSU | AL009126 | 30279..31832 | Прямая |
| CLOB1 | CP000726 | 9282..10783 | Прямая |
| ENTFA | AE016830 | 248466..249987 | Прямая |
| FINM2 | AP008971 | 197837..199361 | Прямая |
| GEOKA | BA000043 | 10421..11973 | Прямая |
| LACDA | CR954253 | 45160..46720 | Прямая |
| LISMO | AL591980 | 96266..97811 | Обратная |
| STAA1 | AP009324 | 531922..533476 | Прямая |
Вырежем нужные участки командой seqret. Поместим последовательности в один файл all.fasta и отредактируем названия последовательностей, осавив только мнемонику видов. Создадим выравнивание и построим дерево в программе MEGA по алгоритму максимального правдоподобия.
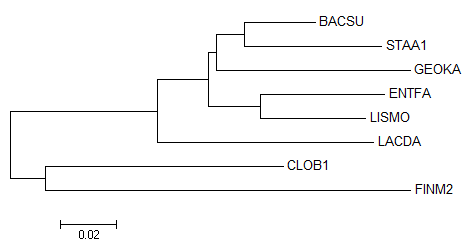
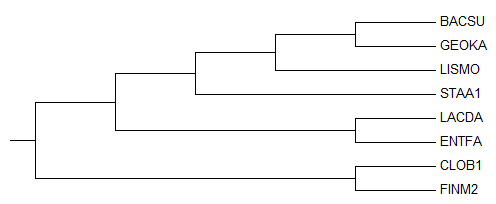
Ниже показано правильное дерево:
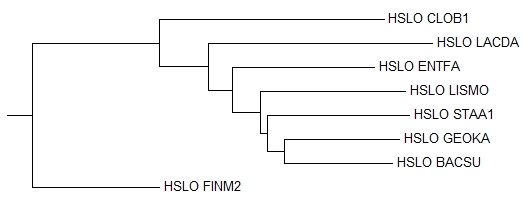
И одно из деревьев, реконструированное по белкам:
Как мы можем заметить, дерево, построенное по нуклеотидным последовательностям отличается от правильного. Появляются ветви {BACSU,STAA1} vs {GEOKA,ENTFA,LISMO,LACDA,CLOB1,FINM2} вместо {BACSU,GEOKA} vs {LISMO,STAA1,LACDA,ENTFA,CLOB1,FINM2}; {BACSU,STAA1,GEOKA} vs {ENTFA,LISMO,LACDA,CLOB1,FINM2} вместо {BACSU,GEOKA,LISMO} vs {STAA1,LACDA,ENTFA,CLOB1,FINM2}; {BACSU,STAA1,GEOKA,ENTFA,LISMO} vs {LACDA,CLOB1,FINM2} вместо {BACSU,GEOKA,LISMO,STAA1,LACDA} vs {ENTFA,CLOB1,FINM2}.
Дерево, построенное по нуклеотидным последовательностям должно быть хуже, чем построенное по белкам. Это объясняется тем, что в нуклеотидных последовательностях всего четыре различных элемента алфавита (против 20 в белковых). Дерево, построенное по белковым последовательностям (белок шаперонин - HSLO), оказалось более удачным (присутствует всего одна нетривиальная ветвь, которой нет в правильном дереве), нежели построенное по нуклеотидным (пять нетривиальных ветвей).
Построение и анализ дерева, содержащего паралоги
Найдем в наших бактериях достоверные гомологи CLPX_BACSU. Чтобы найти гомологов в заданных организмах воспользуемся файлом proteo.fasta на диске P там лежат записи банка UNIPROT, относящиеся к бактериям, перечисленным в таблице к заданию 1. Проведем поиск программой blastp гомологов (с порогом на E-value 0,001) и отберем по мнемонике видов только те находки, которые относятся к отобранным нами бактериям.
makeblastdb -in proteo.fasta -out prot -dbtype prot
blastp -query clpx_bacsu.fasta -db prot -out prot_clpx.txt -evalue 0.001
Получили набор белков prot_clpx.txt и создали файл prot.txt, содержащий только те находки,которые относятся к отобранным нами бактериям. Полученный файл импортировали в программу MEGA (с использованием метода максимального правдоподобия) и построили дерево.
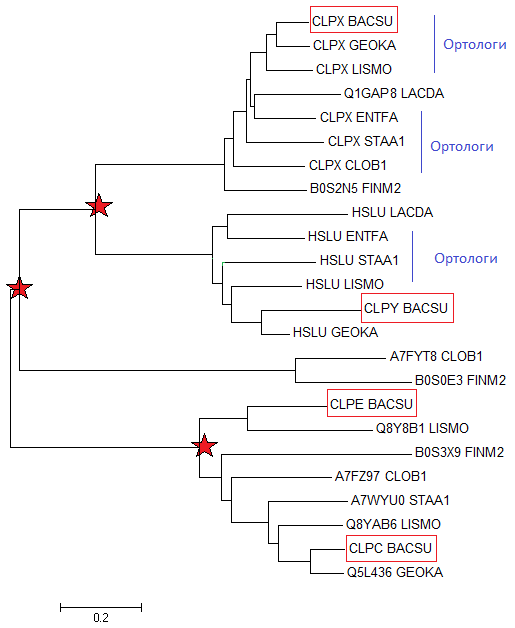
Считая дерево реконструированным верно, укажем несколько пар ортологов и несколько пар паралогов. Ортологи - это гомологи, возникшие в результате видообразования, то есть это белки в разных организмах, имеющие одну функцию. Паралоги - гомологи, возникшие в результате дупликации одного и того же гена в конкретном организме (то есть белки, гены которых появились из одного путем дупликации, но благодаря расхождению и мутациям эти белки имеют разные функции).
На рисунке ортологи помечены синей линией. Группа паралогов выделена рамочкой красного цвета, места дупликаций указаны звездочками.