|
| Для нахождения отличий между различными типами множественного выравнивания я использовала шесть последовательностей гомологичных белков
(семейства HSP70 (два из эукариот (PDB ID HSP7C_HUMAN_1;
HSP71_YEAST_1), два из бактерий (PDB ID: DNAK_THEFY_1; DNAK_THEVO_1), два из архей (PDB ID: DNAK_BEUC1_1: DNAK_THEAC_1),
множественное выравнивание которых строилось раннее.
При помощи данного ресурса было построено два различных множественых выравнивания данных последовательностей:
|
Рис.1 Сравнение множественных выравниваний  |
КОММЕНТАРИЙ К ВЫРАВНИВАНИЮ |
Сверху представлено выравнивание Clustal Omega, снизу Kalign. Как видно из Рис.1 между ними есть некоторые различия:
|
|
В данной работе рассматривался белок Рибонуклеазы J (RNJ1_BACSU), структура которого уже описывалась ранее. Данный белок обнаруживается в бактерии кишечной палочки, является эндонуклеазой и играет роль в стабилизации и деградации рРНК и мРНК. С помощью PFAM была получена доменная аркитектура Рибонуклеазы J, которая представлена на рисунке ниже. Рис.2 Доменная архитектура Рибонуклеазы J
|
Как видно из рисунка, в состав белка входят два домена:
|
РАЗЛИЧНЫЕ ВАРИАНТЫ ДОМЕННОЙ АРХИТЕКТУРЫ, СОДЕРЖАЩЕЙ ДОМЕН LACNAMASE_B1)24009 последовательностей содержат лишь домен Lactamase_B(координаты 21-210):  2)Достаточно распространенной является также архитектура, при которой последовательность содержит и домен Lactomase_B и домен RMMLB. В том числе 2809 последовательностей имеют следующую архитектуру: домен lactamase_B(координаты:17-294) и домен RMMLB (координаты:361-416) 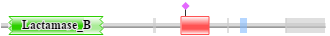 Таже белок содержащий оба данных домена может иметь следующую архитектуру (1272 последовательности):домен lactamase_B(координаты:9-269), домен Beta-Casp(координаты: 250-379) и домен RMMLB (координаты:361-416).  3) Пример достаточно редкой доменной архитектуры, характернйо для 23 последовательностей:домен lactamase_B(координаты: 36-233), неполный домен Flavodoxin_1(координаты: 264-396) и неполный домен Flavin_reduct (координаты: 428-574).  |
РАЗЛИЧНЫЕ ВАРИАНТЫ ДОМЕННОЙ АРХИТЕКТУРЫ, СОДЕРЖАЩЕЙ ДОМЕН RMMBLДомен RMMBL является более редким, и практически всегда встречается в арзитекутрах, так или иначе содержащих домены лактамаз В, и достаточно часто домены Beta-Casp. Наиболее распространенным типом архитектуры, содержащей данный домен являются архитектуры, описанные в пункте 2). Достаточно распросранена также доменная архитектура, содержащая домен СPSF73-100_C (418 последовательностей): домен lactamase_B(координаты:19-228), домен Beta-Casp(координаты: 255-381), домен RMMLB (координаты:397-466) и домен СPSF73-100_C( координаты: 542 - 774).  |
| Главнaя страница |
