ВЫБОР ДОМЕНА И ПОСТРОЕНИЕ ВЫРАВНИВАНИЯ | ||||||
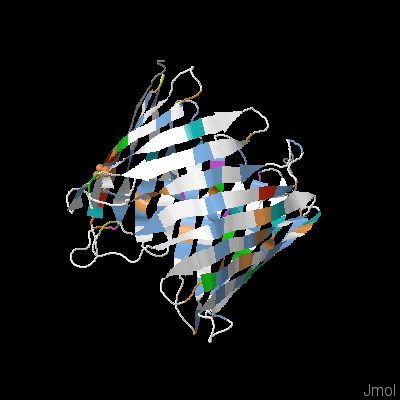
В качестве домена был выбран домен Porin_3 (AC: PF01459). Домен составляет основу потенциал-зависимых анионных каналов на наружней эукариотической митохондриальной мембране. Данные белки ведут себя как диффузные поры, проницаемые для небольших гидрофобных молекул. Иммет открытую конформацию при нулевом мембранном потенциале и закрытую при потенциале 30-40 мВ. Больше информации о домене содержится на странице PFAM. | ||||||
| ||||||
Из базы данных PFAM было скачано выравнивание всех 2735 последовательностей. К нему была добавлена 3D структура белка, представленная на Рис.2. По ссылке можно ознакомиться с проектом выравнивания и выравниванием в формате fasta. | ||||||
ВЫБОР ДОМЕННЫХ АРХИТЕКТУР | ||||||
| Для дальнейшей работы были выбраны две доменные архитектуры, представленные ниже, наиболее представленные. Только для двух данных архитектур количество последовательностей превысило 10. | ||||||
Porin_3 - 2460 последовательностей. (X0KGY6_FUSOX; Mitochondrial import receptor subunit tom-40 {ECO:0000313|EMBL:EXM07931.1})  | ||||||
Porin_3 x 2 - 110 последовательностей (V5FDX7_BYSSN; (Paecilomyces variotii)] Outer mitochondrial membrane protein porin {ECO:0000313|EMBL:GAD95544.1})  | ||||||
С помощью скрипта swisspfam_to_xls.py была получена таблица с информацией о всех последовательностях (2816), содержащих выбранный домен Porin_3, координатах этого домена в последовательностях и др. Строки полученной таблицы содержат последовательности, колонки - информации о домене. Для этого была использована следующая команда: | ||||||
python swisspfam_to_xls.py -z -i /srv/databases/pfam/swisspfam.gz -p PF01459 -o PF01459.xls | ||||||
Из данной таблицы была получена сводная, содержащая информацию о доменной архитекутре каждой из найденных последовательностей. Строчки представляют из себя информацию об одной последовательности, столбцы содержат информацию о присутствии конкретного домена в последовательности. | ||||||
Из базы данных uniprot был скачен текстовый файл с информацией о всех встретившихся в таблице последовательностях по их AC. Полученный файл содержал информацию о 2617 последовательностях. С помощью скрипта uniprot_to_taxonomy.py при помощи следующей команды была получена таблица с данными о таксономической принадлежности каждой последовательности. | ||||||
python uniprot-to-taxonomy.py -i uniprot_ID.txt -o taxonomy.xls | ||||||
С помощью функции Excel ВПР к сводной таблиц были добавлены таксономия для каждой последовательности. Для каждой выбранной архитектуры были выбраны три подтаксона из Eukariota - Fungi, Metazoa и Viridiplantae. Из каждого подтакссона были выбраны по 20 последовательней для обеих архитектур. С полным списоком отобранных последовательностей можно ознакомиться на листе таблицы Final. | ||||||
ИТОГОВАЯ ТАБЛИЦА | ||||||
ВЫРАВНИВАНИЕ ВЫБРАННЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | ||||||
При помощи последовательного применения скриптов , filter-alignment.py и к информации представленной на листе final итоговой таблицы было получено отфильтрованное выравнивание, содержащее лишь домены отобранных последовательностей. К названию каждой последовательности были добавлены буквы, указывающие на ее принадлежность к первой или второй доменной архитектуре, а также к подтаксону. Фильтрация последовательностей осуществлялась командой: | ||||||
python filter_alignment.py -i PFAM_porin_alignment.mfa -m IDs_porin3.txt -o selected_alignment.fasta -a "_" | ||||||
В полученном выравнивании последовательности были расположены в порядке принадлежности к подтаксонам и объеденины в группы по доменным архитектурам. Из выравнивания были удалены все пустые колонки, а также малоинформативные участи последовательностей и сомнительные последовательности. Была осуществлена покраска по группам. С проктом выравнивания можно ознакомиться по ссылке на проект выравнивания .jar, .jvp. | ||||||
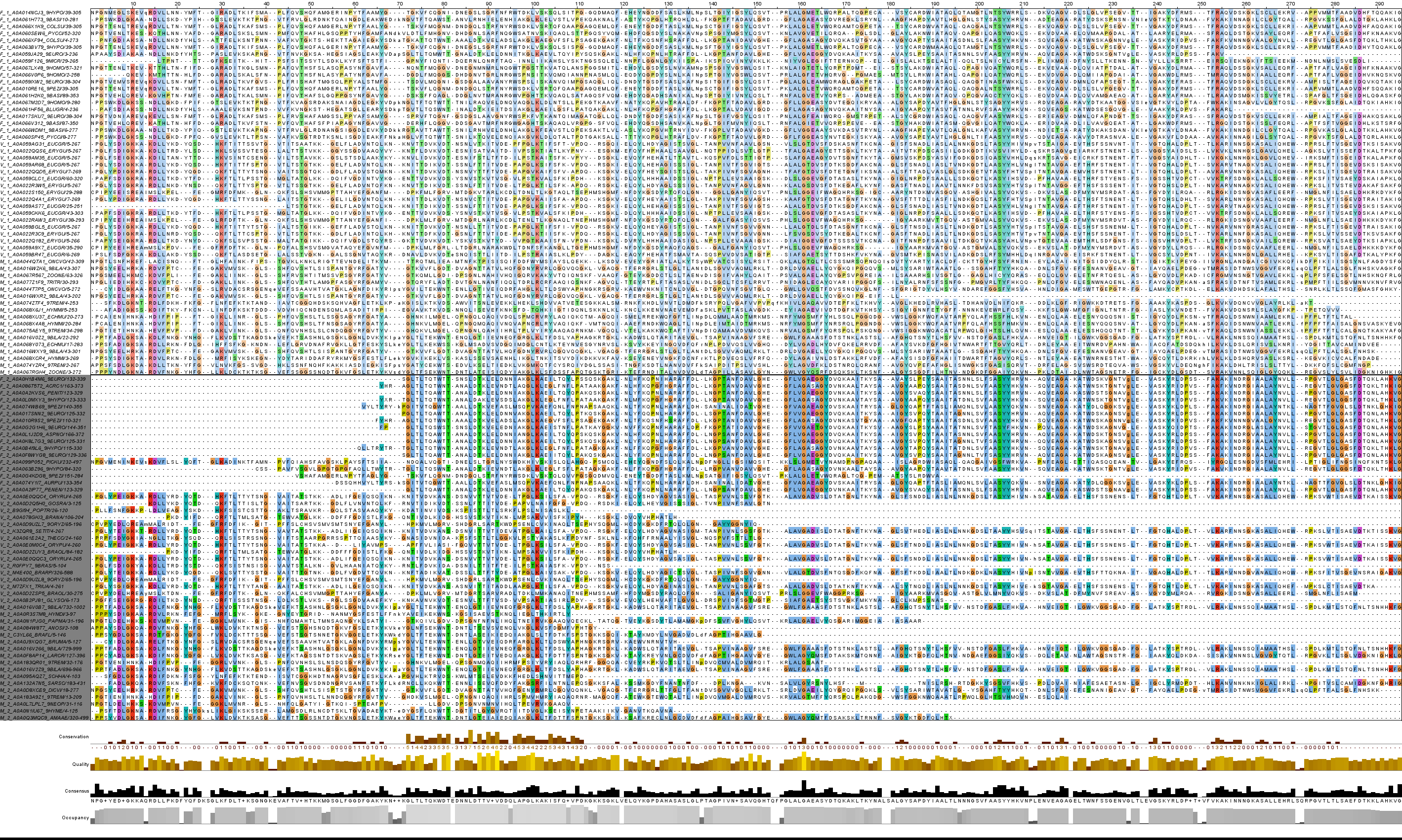 | ||||||
ПОСТРОЕНИЕ ДЕРЕВА | ||||||
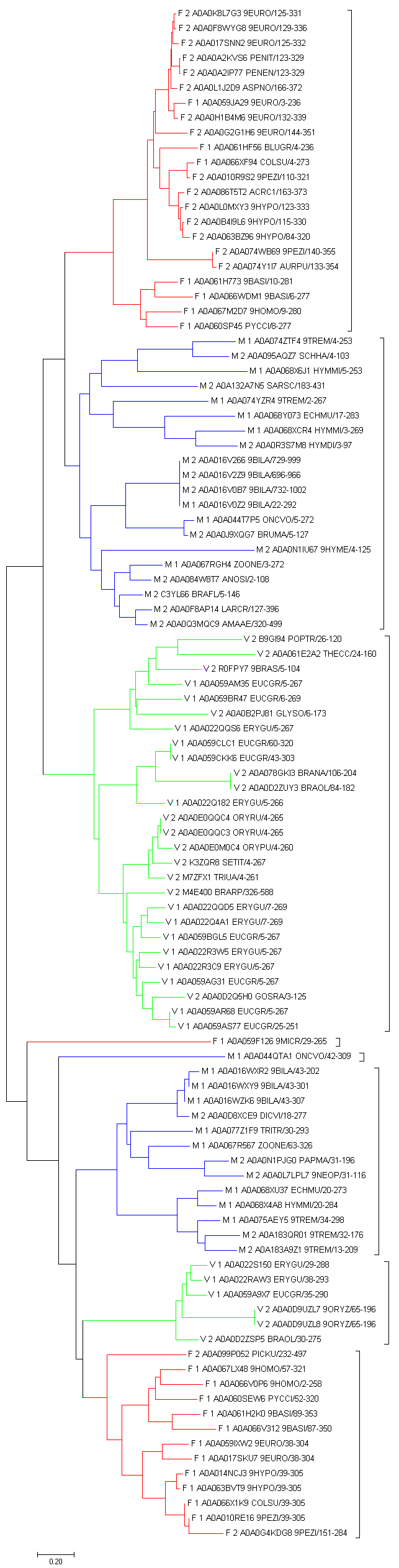
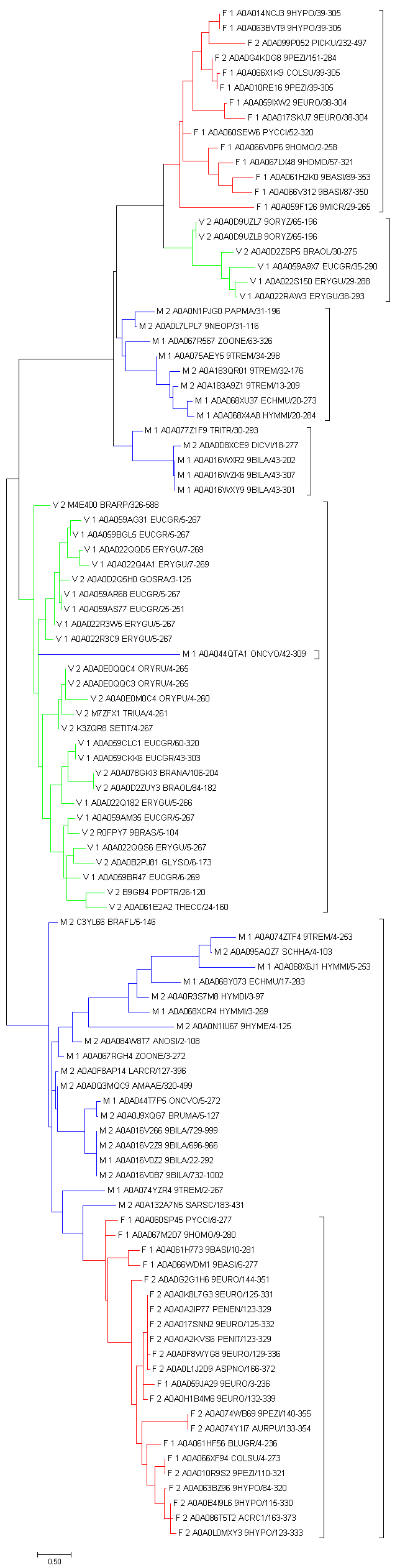
По представленному выше выравниванию производилось построение дерева двумя методами - Maximum Likelihood и Neighbour joining. Для облегчения восприятия приведены две возможные формы отображения дерева. Клады, отображающие подтаксон Fungi окрашены на деревьях красным цветом, клады Metazoa - синим, Viridiplantae - зеленым. | ||||||
| ||||||
Как видно, построенные деревья оказались довольно близки. | ||||||
Любопытным оказался тот факт, что не наблюдается разделения в линии эволюции ни по доменным архитектурам, ни по таксонам. Так, видно, что последовательности из одного таксона оказываются объеденины в довольно крупные группы, однако не образуют целую кладу, а соседничают с другими таксонами. Из этого можно предположить, что разделение белка с данным доменом на две группы произошло еще до разделения данных таксонов, отсюда у для каждого таксона представлено две отдаленные друг от друга клады на дереве, сближенные с двумя другими небольшими кладами из двух других таксонов. Всего таких групп 6 - 3 и 3 по числу таксонов. При этом различные доменные архитектуры повсемесстно на деревьях оказываются на соседних ветвях, без каких-либо четких наблюдаемых разграничений. | ||||||
| Для обоих деревьев` можно ознакомиться со скобочными формулами: | ||||||
| Главнaя страница | ||||||