ОПРЕДЕЛЕНИЕ ЦЕЛЕВОГО СЕМЕЙСТВА |
В рамках данного задания был рассмторен домен PFAM из предыдущего практикума - PF01459. Этот домен составляет основу потенциал-зависимых анионных каналов на наружней эукариотической митохондриальной мембране. Данные белки ведут себя как диффузные поры, проницаемые для небольших гидрофобных молекул.[1] В бызе данных Uniprot был выполнен поиск по данному домену, среди организмов, относящихся к типу Chordata со слудующим запросом: database:(type:pfam id:PF01459) taxonomy:"Chordata [7711]" AND reviewed:yes В результате было найдено 32 белка, содержащих данный домен и относящихся к данному типу. Таблица на выдаче была преобразована так, что содержала следующие параметры:
C Полученной таблицей в формате tab-separated можно ознакомиться по ссылке. Из полученных данных стало понятно, что все найденные последовательности содержат только доменную архитектуру 1 из предыдущего практикума (наиболее популярную - 2460 последовательностей PFAM из 2735): |
 |
ПОСТРОЕНИЕ И КАЛИБРОВКА ПРОФИЛЯ ДОМЕНА |
| Далее по выбранным последовательностям было осуществленно построение выравнивания, приведенное на рисунке ниже. Из выравнивания были удалены начальные фрагменты некоторых
последовательностей, а также последовательность VDAC2_MESAU. Среди последовательностей оказались два типа белков, довольно существенно отличающиеся друг от друга,
(VDAC и TOM/TM), однако оба типа по данным PFAM имеют одинаковую доменную архитектуру и относятся к белкам транспортерам на наружней
митохондриальной мембране, поэтому были оставлены в выравнивании. Окраска Clustal, %Идентичности - 20%.
Выравнивание содержит последовательности из отдаленных таксонов, входящих в chordata (грызуны, бычьи, гоминиды и др.)
Проект выравнивания доступен по ссылке. |
 |
| Далее при помощи программы hmm2build по данному выравниванию был построен HMM профиль с использованием следующей команды: |
hmm2build -g profile.out chordata_porin3.mfa |
После калибровки полученного профиля командой: |
hmm2calibrate profile.out |
| Был получен слудующий HMM профиль |
ПОИСК ПО ПРОФИЛЮ В БАНКЕ SWISSPROT |
Поиск по профилю в swissprot осуществлялся при помощи следующей команды (для подбора порога необходимо сопостовимое с числом правильных находок число неправильных находок, в связи с этим были выставлены параметры посика --domE 1000 и --domT -50 - пороговое значение E-value = 1000 и снижение порога веса) |
hmm2search --domE 1000 --domT -50 profile.out /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > find.fata |
На выходе программы был получен файл, содержащий список параметров запуска, таблицы найденных последовательностей, таблицы найденных доменов, выравниваний находок относительно профиля, гитсограммы весов находок. Таблица найденных доменов была импортирована в итоговую таблицу Excel, правильно найденные последовательности были помечены Y. Всего было найдено 64 доменов, из них 24 правильно. |
ПОСТРОЕНИЕ ROC КРИВОЙ |
Для находок, представленных в таблице найденных доменов была построена гистограмма весов этих находок (Рис.1) |
Рис. 1. Гистограмма весов находок.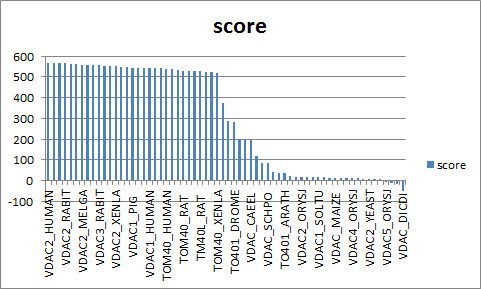 |
На основании этой гистограммы был выбран порог для веса - 371.8, имеенно на этом значении проиходит скачек весов в гистограмме. Среди всех находок лишь одна TP находка имела скор ниже порогового. |
Для разных пороговых значений (веса находок >= 0.9, 0.8, 0.7, 0.5, 0.3, 0 от общего числа) были вычеслены значения Sensitivity и 1- Specificity. Был построен график зависимости этих двух параметров друг от друга, ROC-кривая (Рис.2). |
Рис. 2. ROC кривая. |
| Таким образом, значение выбранного порога - 371. Для этого значения чувствительность - 0,95, специфичность - 0,775, точность профиля - 0.375, точность порога - 0.72. |
На мой взгляд, данный профиль не является эффективным для поиска, не смотря на то, что по нему находятся все (кроме одной) целевые последовательности, список находок включает в себя 25% побочных находок (8 из 31). |
ИТОГОВАЯ ТАБЛИЦА |
ССЫЛКИ |
| Главнaя страница |