|
Карань Анна |
|||
| Главная | О себе | Учеба | ФББ МГУ |
Реконструкция филогении по РНК
Задание 1. Построение дерева по нуклеотидным последовательностям
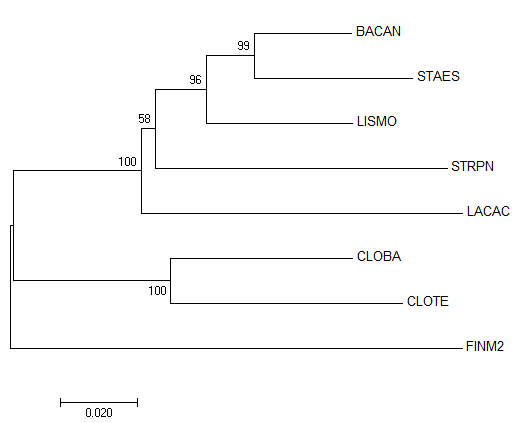
Деревья строились на основе выравнивания последовательностей РНК малой субъединицы рибосомы (16S rRNA). Они были взяты из базы полных геномов NCBI ftp://ftp.ncbi.nlm.nih.gov/genomes/archive/old_refseq/Bacteria/. Взяты последовательности видов из 1-го практикума . Получен файл последовательностей. По нему в программе Mega было построено выравнивание. Далее в Mega методом Neighbor-Joining было построено дерево (Рис.1.)
 |
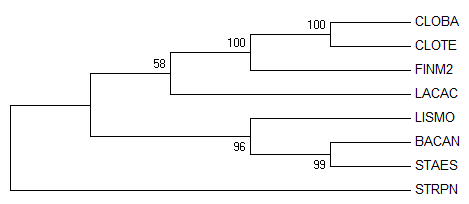 |
Рис.1. Здесь показаны деревья, построенные в Mega методом Neighbor-Joining. Слева - Original tree, справа - Bootstrap consensus tree | |
С первого раза может показататься, что деревья, изображенные на Рис.1. отличаются, но это не так. Bootstrap consensus tree - то же самое, что и Original tree, вот только внешне укорененное в ветвь STRPN. (Однако, если построить укоренение по STRPN дерево получается сильно не сбалансированным, что, конечно, не приветствуется.) Так что пока сравним без учета укоренения ветви с правильным деревом. Единственное отличие от верного дерева - LACAC и STRPN не отделились в одну тривиальную ветвь. Дерево получилось гораздо лучше, чем по белкам (в случае белков верной была только 1 ветвь), что логично, так как rRNA (особенно 16s) очень распространены для определения и дальних, и близких родственных связей из-за нераспространенности горизонтального переноса rRNA (такие случаи известны, вот недавно на конференции слушала про водоросль с гпг rRNA от малярийного плазмодия (статьи еще нет, ссылку привести не могу), это считается чем-то удивительным)
Задание 2. Построение и анализ дерева, содержащего паралоги
| Таблица 1. Соответствие находки и мнемоники в выравнивании и дереве | |
| Мнемоника | Название белка (мнемоники видов как в прошлых практикумах) |
| STRPN_Clpx | ATP-dependent Clp protease ATP-binding subunit ClpX |
| BACAC_Clpx | ATP-dependent Clp protease ATP-binding subunit ClpX |
| LISMO_Clpx | ATP-dependent protease ATP-binding subunit ClpX |
| STAES_Clpx | ATP-dependent protease ATP-binding subunit ClpX |
| CLOBA_Clpx | ATP-dependent Clp protease ATP-binding subunit ClpX |
| FINM2_Clpx | ATP-dependent Clp protease ATP-binding subunit ClpX |
| LACAC_Clpx | ATP-dependent Clp protease ATP-binding subunit ClpX |
| CLOTE_Clpx | ATP-dependent Clp protease ATP-binding subunit ClpX |
| LACAC_Hsluv | HslU--HslV peptidase ATPase subunit |
| BACAC_Hslu | ATP-dependent protease ATP-binding subunit HslU |
| STAES_Hslu | ATP-dependent protease ATP-binding subunit HslU |
| LISMO_Hslu | ATP-dependent protease ATPase subunit HslU |
| CLOBA_Clpb | ATP-dependent chaperone ClpB |
| STRPN_dr | dihydrofolate reductase |
| CLOTE_Clpc | ATP-dependent Clp protease ATP-binding subunit ClpC |
В данном задании необходимо реконструировать дерево по достоверным гомологам белка "ATP-dependent Clp protease ATP-binding subunit ClpX OS=Bacillus subtilis". В Таблице 1 представлены выбранные гомологичные белки и сопоставленные им мнемоники, поиск гомологичных белков осуществлялся с помощью blastp на сайте NCBI с ограничениям по нужным организмам. Все находки с e-value значительно ниже 0,001 Далее все белки были выравнены программой Musle в Mega - выравнивание Потом было построено дерево методом Neighbor-Joining.
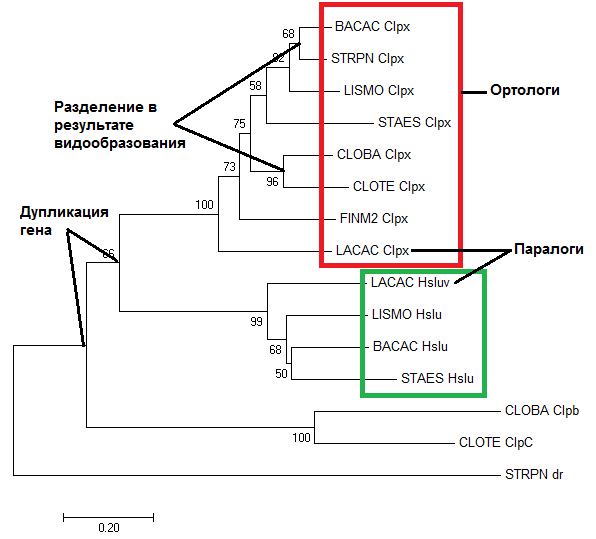
Рис.2 Дерево, построенное в Mega. Цветными прямоугольниками выделены группы ортологов, и выделен пример паралогов.
На Рис.2 приведены примеры ортологов и паралогов. Красный прямоугольник последовательности субъединицы Clpx, зеленый - HslU, это судъединицы, выполняющие схожие функции в разных протеазах, так что они достаточно похоже. А паралоги - это гомологичные белка в одном организме, так что это паралоги - любые 2 последовательности из этих двух блоков из одной бактерии. Так же на Рис.2 показаны отраженные на дереве эволюционные события двух типов: 1) дупликация гена; 2) разделение путей эволюции белков в результате видообразования, на каждый тип по 2 примера.
©Карань Анна, 2015