Вычисление информационное содержание (IC) последовательностей Козак в геноме данио рерио
Для выполнения задния был выбран четвётный варинат выравниваний. Ссылка на xlsx таблицу с выравниваним и промежуточными вычислениями. Алгоритм выполнения задания:
Также выравнивание было загружено в сервис webLOGO для получения диаграммы LOGO. Её можно увидить на Рис.1.
Рис.1 LOGO выравнивания из варианта 4
Поиск мотива в геноме Bat coronavirus CDPHE15/USA/2006 с помощью программы FIMO
Для выполнения этого задания использовался мотив из прошлого практикума . По html выдаче meme проводился поиск мотива в геномах исходного генома, в геноме другого штамма - Myotis lucifugus coronavirus, а также в геноме родственного короновируса Rousettus bat coronavirus HKU10. Порог E-value был выставлен в 0.001. Команда запуска:
fimo --motif 1 --norc --verbosity 1 --output-pthresh 1.0E-3 meme_out2/meme.html NC_022103.1.fasta
Для других геномов fimo запускалось с аналогичными параметрами. результат работы fimo можно скачать:
Также находки fimo для исходного генома представлены на Рис.2. Можно увидеть, что с хорошими e-value fimo нашел мотив как раз в upstream областях генов. Хотя одна неплохая находка почему-то нашлась между генами PP1a и S. В других геномах нашлось 15 и 29 находок соответственно. Обилие находок с хорошим e-value как в другом штамме, так и в соседнем виде, указывает на то, что мотив распространенный. В геноме Myotis lucifugus coronavirus расположение мотивов частично совпадает с мотивами в исходном геноме, что вобщем-то и ожидалось.
Для исходного вируса также были найдены последовательности Козак и построено LOGO в вебсервисе webLogo (Рис.3). Его требовалось сравнить с Logo последовательностb Козак человека (Рис.4). Изображение взято из Википедии. Как видно, они похожи только в паре позиций - +2 от ATG, где чаще встречается цитозин и в -3 от ATG, где чаще аденин. Возможно, белки этого конкретного вируса было бы сложнее транслировать рибосоме человека, раз последовательности Козак так непохожи (зато, наверное, просто во всяких инфузориях и слизевиках, у них, судя по Википедии, как раз много аденинов в последовательности Козак).
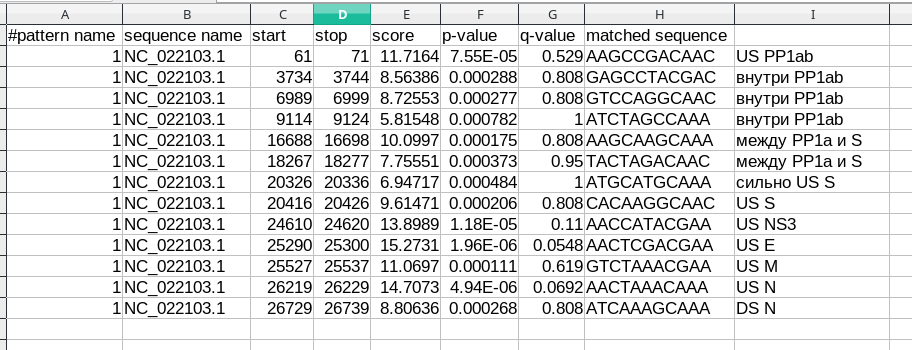
Рис.2 Результат работы fimo с подписями в Libre Office

Рис.3 Logo последовательности Козак для Bat coronavirus CDPHE15/USA/2006
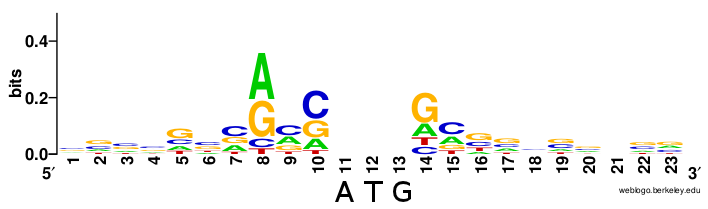
Рис.4 Logo последовательности Козак человека