Pfam
Часть 1
Для выполнения этого практикума необходимо было выбрать бактериальный домен, по которому следовало составить архитектурный профиль. Было решено взять N-терминаьный домен Rho фактора, который у прокариот может вызывать терминацию транскрипции. В списке возможных архитектур c N-концевым доменом Rho фактора нашлась неуникальная (257 последовательностей в Pfam) архитектура в два домена: ChaB, Rho_N. Сcылка на список возможных архитектур c Rho_N. ChaB - это домен транспортера катионов.
Далее поиск последовательностей проводился в Uniprot. Строка поиска: taxonomy:"Bacteria [2]" database:(type:pfam pf06150) database:(type:pfam pf07498) . Было найдено 815 последовательностей, все - в Tremble. После добавления нужных таксономических колонок находки были скачены в формате Excel.
Интересно, что почти все находки относятся к отряду Actinobacteria, ещё 5 - к Proteobacteria и 1 - к Chloroflexi. В исследовании 2013 года консервативности и филогении Rho фактора на картинке 4 видно, что эти три отряда находятся на одной ветке, то есть, возможно, такая архитектура появилась довольно давно и почему-то закрепилась у некоторых актинобактерий. "Классический" N-концевой домен Rho-фактора состоит из N-терминирующего домена, РНК-связывающего и АТФ-синтазного доменов, в связи с чем возникает вопрос, как найденная структура работает и действительно ли она функционирует как Rho-фактор. Кроме того, в скаченных 815 последовательностях не было ни одного мультидоменного, то есть, видимо, они встречаются вместе только в такой комбинации. Дополнительный поиск по Pfam подтвердил, что ChaB встречается либо сам по себе, либо с Rho_N, либо с неким YflT (в составе неохарактеризованного белка), про которого только известно, что он индуцирется теплом.
Мода длины была посчитана в Libre Office и равна 139, нижний квартиль - 133, верхний - 139. Так как верхний квартиль совпадает с модой, мне кажется, можно расширить правую границу интервала самых частовстречающихся длин до 142. Примерный интервал длин - [132:142].
Часть 2
Примерный интервал длин был ещё раз определен с помощью построения гистограммы длин (Рис.1). Его размер - 120-160 ак. Для дальнейшего анализа были выбраны 49 последовательностей характерной длины, из каждого представительного семейства было выбрано по 2-3 последовательности. Как уже было описано выше, преимущетсвенно последовательности из отдела Actinobacteria, в выборку также попали 2 последовательности отдела Proteobacteria. С семействами и родами можно ознакомиться в файле, дотсупном по ссылке выше. Далее в Jalview были загружены последовательности (Fetch sequences => Uniprot => Retrieve IDs) A0A1H0T055_9ACTN почему-то не нашлась, поэтому последовательностей оказалось 48, они были выравнены с помощью программы muscle. Полученное выравнивание оказалось довольно консервативным.
Ревизия
Перед N-концевым блоком было удалено 13 столбцов, хотя были некоторые сомнения в необходимости. С C-конца удалено 3 колонки, всё, что раньше посчитала C-концевым блоком. Исправления можно увидеть на Рис. 2-5.(В последствии оказалось, что я обрезала слишком сильно и HHMER находил слишком много находок, поэтому в дальнейшем использовалась необрезанная версия) Далее с помощью пакета HMMER был построен и откалиброван профиль. Команды запуска:
hmm2build build2.txt aligned_Rho.fasta
hmm2calibrate build2.txt
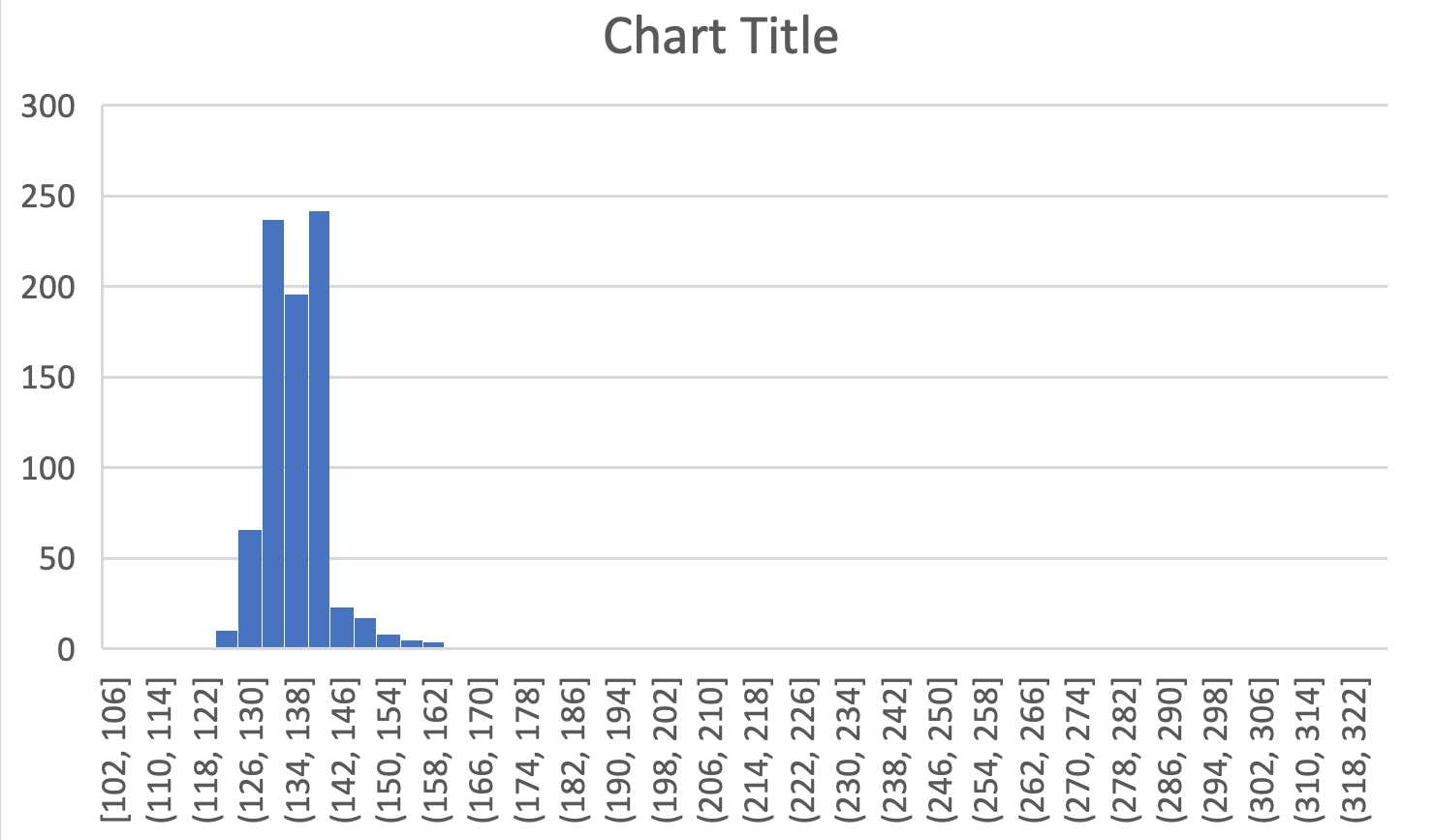
Рис. 1. Гистограмма длин
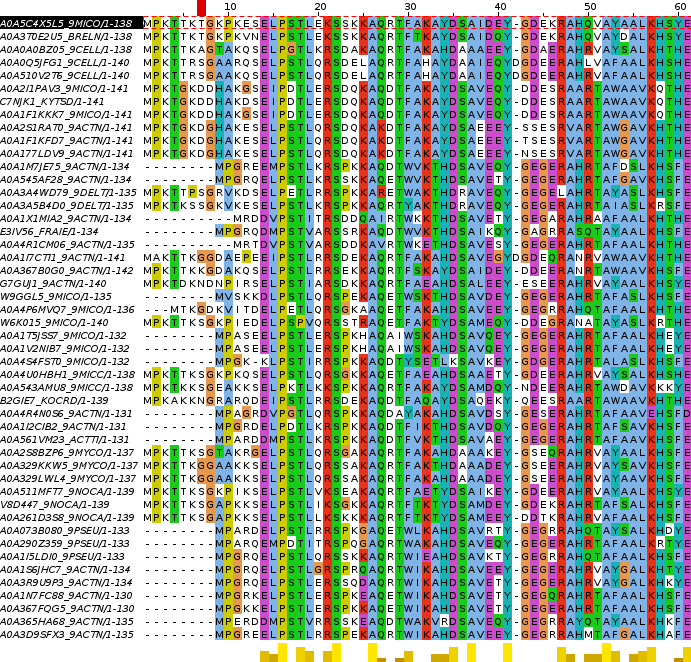
Рис. 2. N-конец в первоначальном виде

Рис. 3. N-конец после удаления первых 13 столбцов

Рис. 4. C-конец в первоначальном виде

Рис. 5. C-конец после удаления последних 3 столбцов
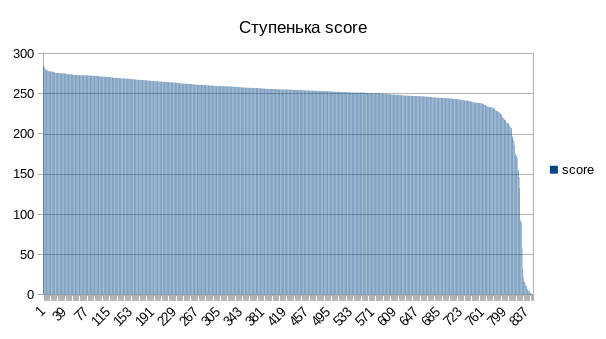
Рис. 6. Score ступенька
Далее с сайта Uniprot были скачены fasta последовательности, содержащие домен pf07498 - он же Rho_N (для другого домена находок было слишком мало). Их нашлось 29651. Чтобы найти среди скачанных последовательностей последовательности с выбранной доменной архитектурой была выполнена команда:
hmm2search -E 0.1 build2.txt findings_pf07498.fasta > result2.txt
Результат доступен для скачивания. После этого в Excel была построена таблица с колонкой, показывающей, находится ли данная последовательность в списке изветсных последовательностей с данной архитектурой для определения специфичности и сенсетивности. Кривая ROС и данные для расчёта доступны по ссылке. Ступенька score приведена на Рис.6. По результатам построения обоих графиков, порог score был определен приблизительно в 220. Однако по максимальному значение параметра F1 порог выходил равным 146. Ориентируясь на это значение (и пренебрегая специфичностью) была создана таблица, по которой можно судить о качестве моего предсказания (Рис.7).
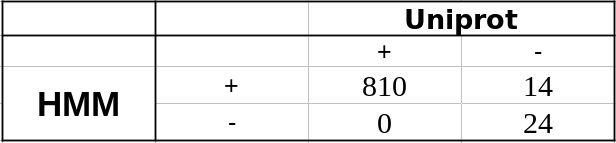
Рис. 7. Таблица на основе установленного порога