10. Эволюция доменной архитектуры
Описание выбранного домена
| AC | PF00224 | ||||||||
| ID | PK | ||||||||
| Функция | Баррель-домен пируваткиназы. Этот домен является небольшим доменом типа бета-баррель, расположенным внутри большого TIM-барреля. Активный центр располагается в щели между двумя доменами. Пируваткиназа (ПК) катализирует последний этап гликолиза (превращение фосфоенолпирувата в пируват, сопровождяющееся фосфорилированием АДФ до АТФ). |
||||||||
| Число разных доменных архитектур с этим доменом | 22 | ||||||||
| Число представителей в разных такосонах высшего порядка |
|
Выбранные доменные архитектуры
| Архитектура | Число представителей | |
| 1: PK |  |
401 п-ть |
| 2: PK + PK_C* |  |
3451 п-тей |
*Описание домена PK_C
| AC | PF02887 | ||||||||
| ID | PK_C | ||||||||
| Функция | Альфа/бета-домен пируваткиназы. Наряду с тем, что является компонентом пируваткиназ, обнаружен также как самостоятельный домен в некоторых белках бактерий. | ||||||||
| Число разных доменных архитектур с этим доменом | 21 | ||||||||
| Число представителей в разных такосонах высшего порядка |
|
Выравнивание JalView.
Таблица Excel со всем необходимым (листы swisspfam_to_xls.out (raw data) --- length).
11. Профили
1. Построение филогенетического дерева выборки последовательностей домена PK
Зашифровываю доменные архитектуры и сравниваемые таксоны в названиях последовательностей:
| Архитектура | 1 | Архитектура №1 (PK) |
| 2 | Архитектура №2 (PK + PK_C) | |
| Таксон | B | Bacteria |
| E | Eukaryota |
Строю филогенетическое дерево выбранных последовательностей, используя известные мне программы. В деревьях, построенных с помощью программ fprotpars (метод maximal parsimony) и fneighbor (метод UPGMA), установить какие-либо закономерности эволюции домена не удается.
В деревьях, сконструированных с использованием бутстрэп-анализа и программой fneighbor (метод NJ), более-менее прослеживается ветвь, содержащая эукариотические последовательности:
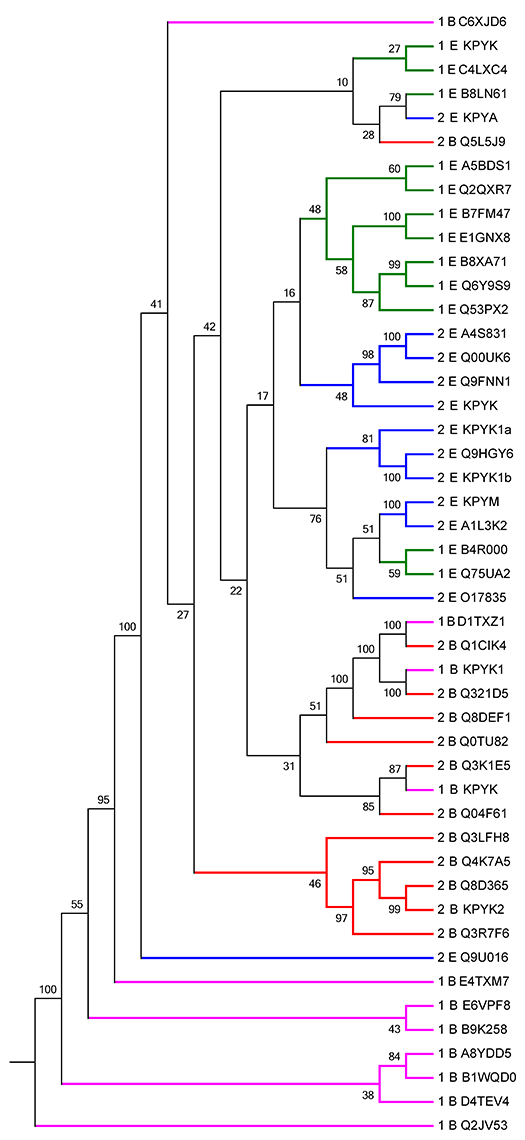
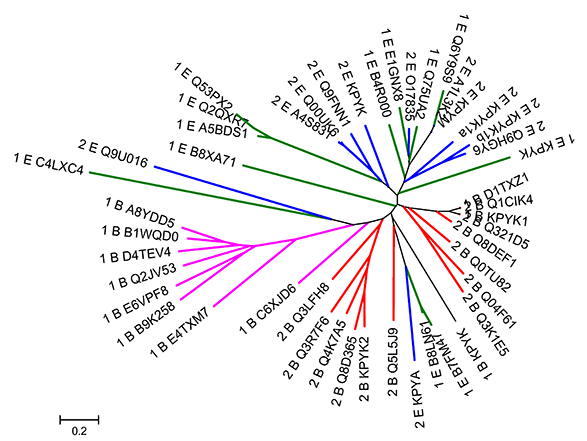
Укоренение дерева, посроенного методом NJ, в среднюю точку с помощью программы retree не дало утешительных результатов.
Кажется вероятной независимая эволюция доменных архитектур у представителей бактерий и эукариот. Никаких предположений относительно того, какая архитектура была характерна для LUCA.
Бактериальные последовательности из однодоменной архитектуры выделяются в обособленную ветвь на дереве, построенном методом NJ, и на большинстве укорененных деревьев. Соответственно можно представить, что из ранее существующей у бактерий двухдоменной архитектуры каким-то образом произошло обособление этого домена. Эукариотические последовательности не образуют клад, коррелирующих с доменной архитектурой. Также ничего нельзя сказать о том, какая из архитектур была представлена у общего предка эукариот.
2. Разделение выравнивания на две группы
Разделяю выравнивание на две группы по таксономии организмов, поскольку на дереве, построенном методом NJ, можно проследить некоторое соответствие таксономическим группам:
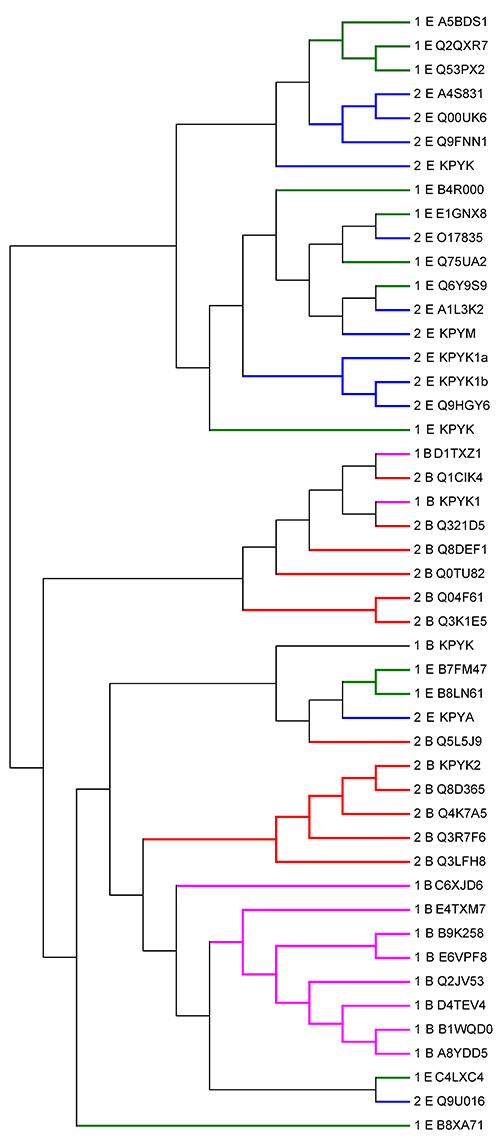
3. Построение профиля, отличающего бактериальные последовательности от эукариотических
В качестве выравниваний, используемых далее для создания профиля, я приготовил три файла: b.msf (содержит все бактериальные последовательности из выравнивания, с которым я работал ранее), e.msf (все эукариотические последовательности) и e_part.msf (содержит все эукариотические последовательности из выравнивания, с которым я работал ранее, за исключением тех, которые часто "примешивались" к бактериальными на построенных деревьях).
Результаты поиска при помощи полученных профилей по исходным последовательностям: Таблица Excel (листы b, e, e_part).
Результаты поиска при помощи полученных профилей по банку SwissProt: Таблица Excel (листы b sw, e sw, e_part sw).
Привожу результаты поиска при помощи профилей по банку SwissProt:
| Профиль | Найдено п-тей бактерий | Найдено п-тей эукариот |
| B | 32 | 14 |
| E | 26 | 42 |
| E_part | 26 | 41 |
Видно, что при выбранных пороговых значениях score число "нужных" последовательностей в выдаче превышает число "ненужных" приблизительно в два раза, поэтому, наверное, можно считать, что нужные профили создать удалось.